| 基于TBtools做基因家族分析教程 (全) | 您所在的位置:网站首页 › 结构域图怎么做 › 基于TBtools做基因家族分析教程 (全) |
基于TBtools做基因家族分析教程 (全)
|
基因家族分析笔记-全部开始记录
一、 写在前面
2023年4月中旬自己开始做基因家族的分析,对于这块自己没有接触过,因此也是一个挑战,没事!!!(安慰自己),对于基因家族的分析网上的教程很多,跟着步骤走就可以。在这部分,我自己主要是做生信这块,实验验证是师姐在做,所以论文结构自己不用操心。此外,可视化的工具很多,也很方便,不需要自己特意去学。我们这里就60%使用TBtools软件进行可视化和分析。 此外,本次分析80%的内容都是基于TBtools。确实牛X!!自己开始接触TBtools是在2019年吧,也是通过一个师兄的推荐才知道的。2019年CJ还没将TBtools发表在MP上,那时还是预印版本吧。但是,引用已经有了很多,了不起哦。后面TBtools一直在开发新的的小“软件” or“程序”,将生信分析的门槛一降再降。点赞点赞!!! –Du
如想获得本文文档,可看文末!! 注意:此教程有些话语可能会带有自己的方言,读不通时也不要在意!![泪目!] 一,在Pfam数据中获得基因家族我们这里预测作物中某一个基因家族的基因,目前在此作物中未报道。因此,使用Pfam数据库中一致的基因进行同源搜索(其实,你也可以使用已知作物中的基因进行同源搜索,获得结果基本一致)。那么我们就根据文章中和报道的Pfam数据库中的基因作为基序,进行同源搜索。 在Pfam数据库中下载FBNs基因家族(Pfam 04755),Pfam网址:https://pfam-legacy.xfam.org/ 打开网址:http://www.ebi.ac.uk/interpro/entry/pfam/?search=0477#table 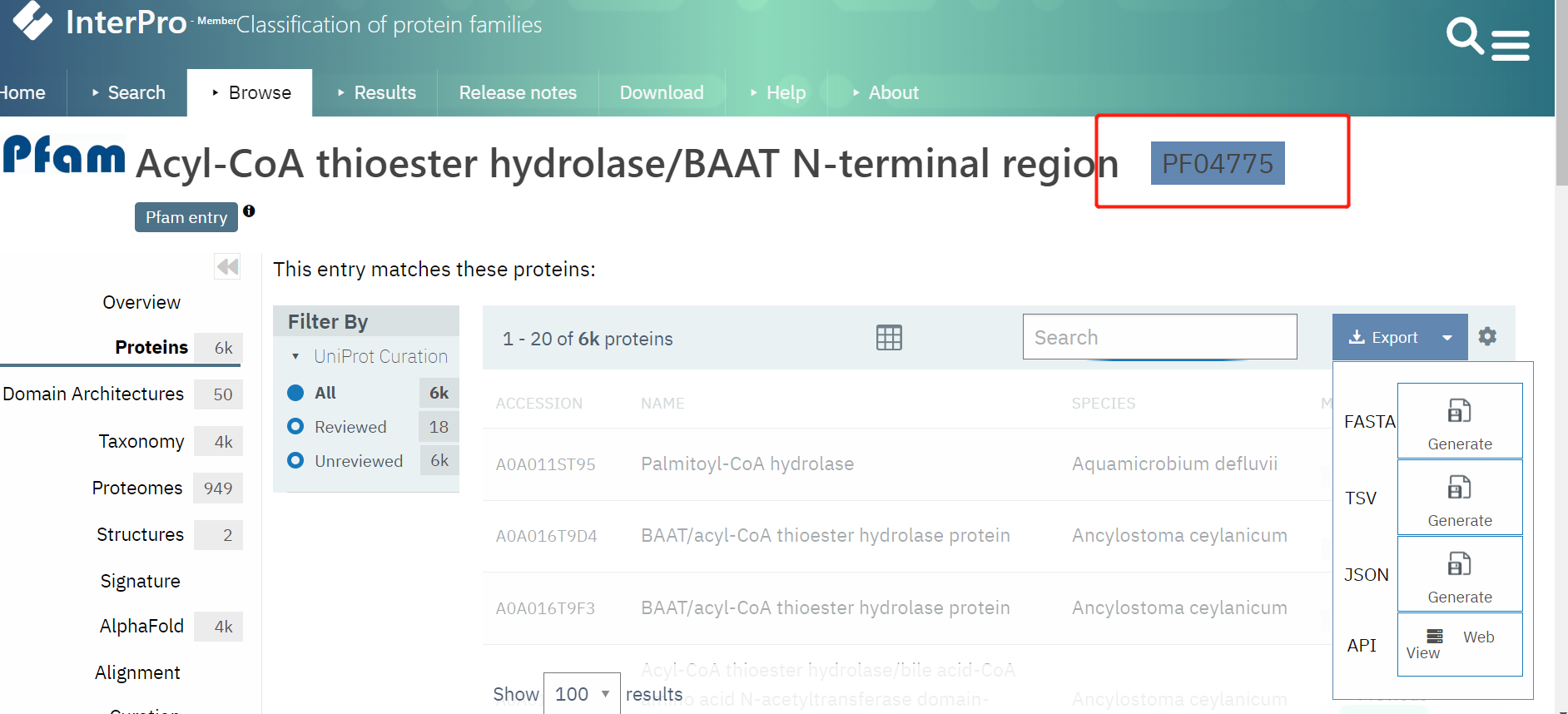
以上只是其中的一种方法,但为获得FBN基因家族的蛋白序列。下面使用Pfam数据中搜索 打开网页。https://pfam-legacy.xfam.org/ 搜索 搜索 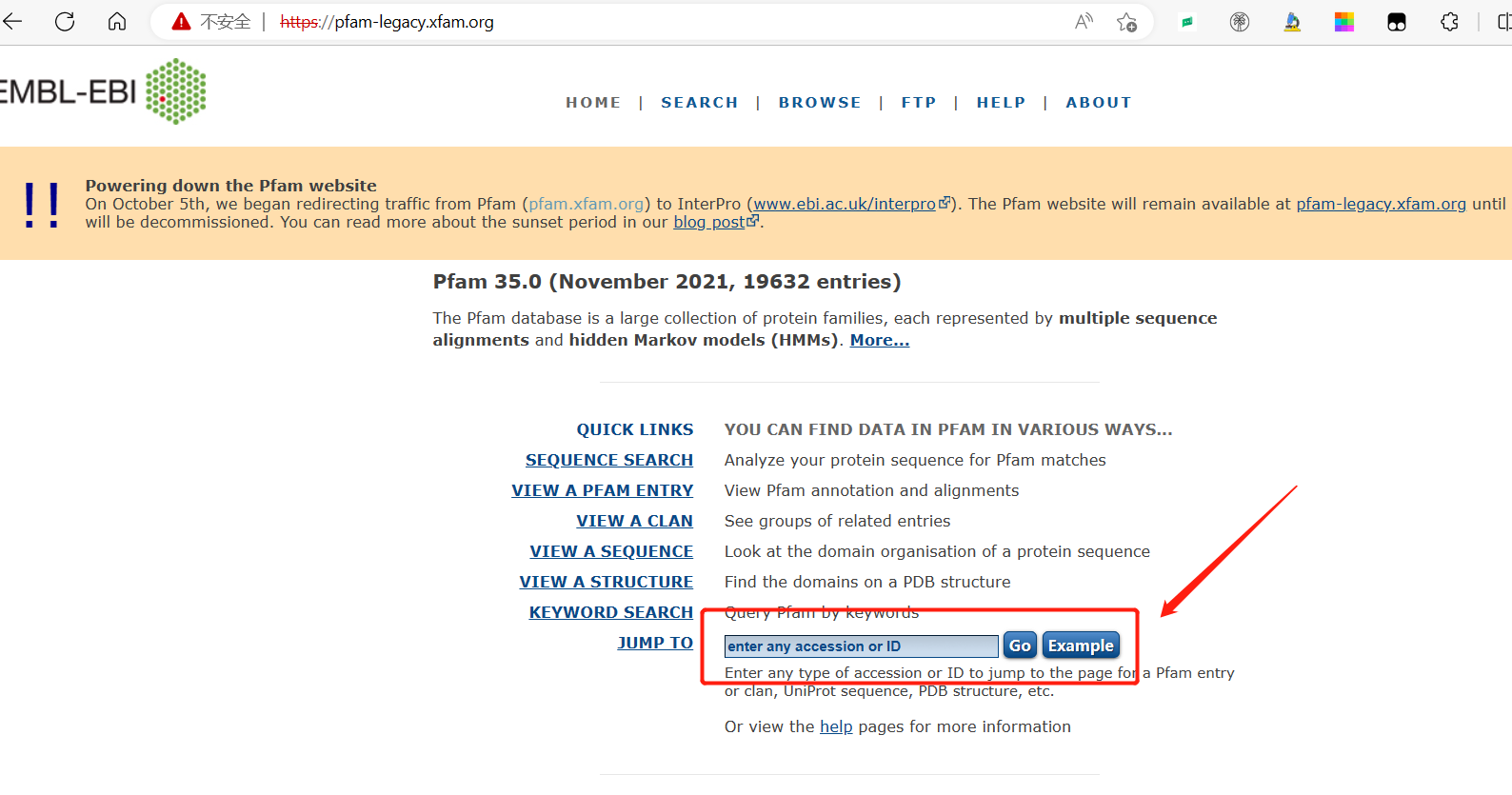 进入 进入 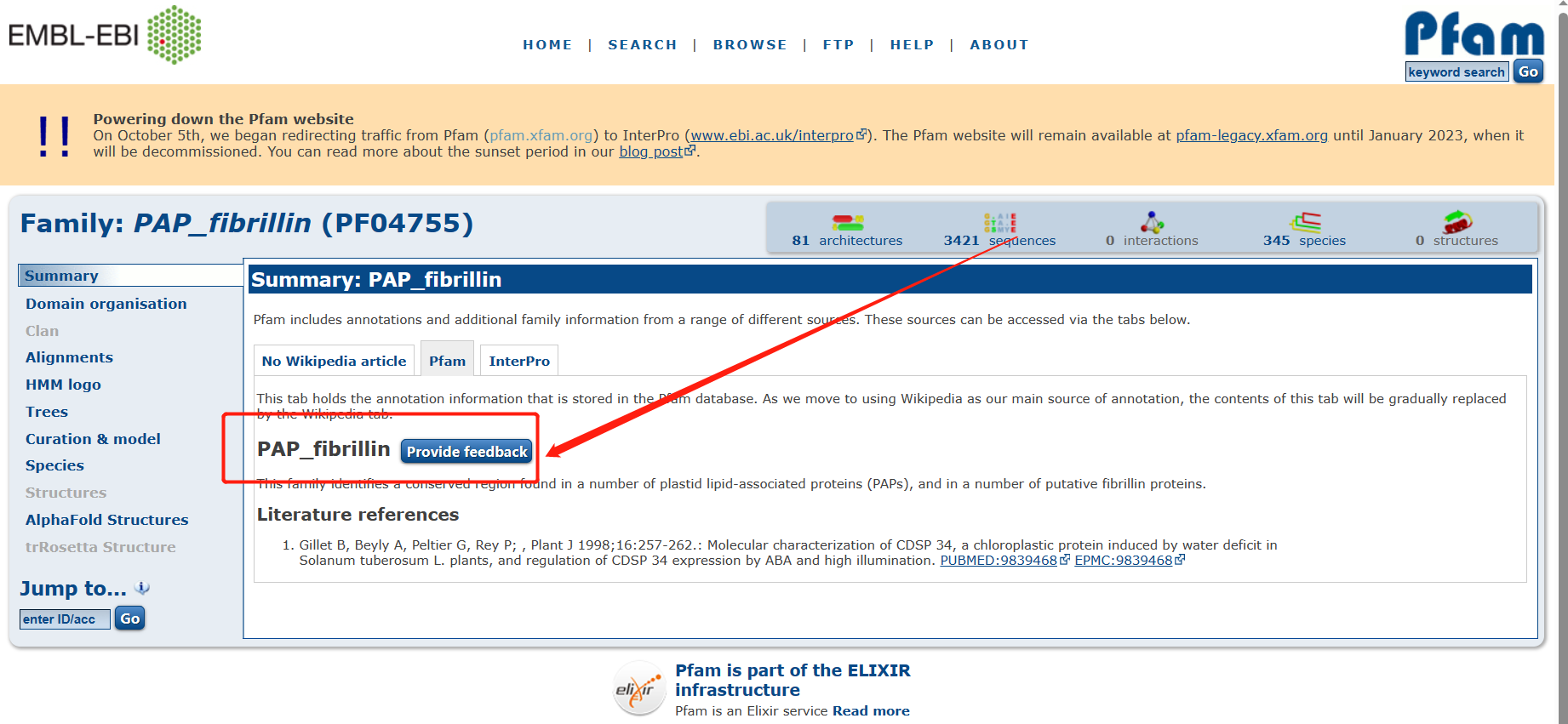 搜索后获得PAP_fibrillin 搜索后获得PAP_fibrillin 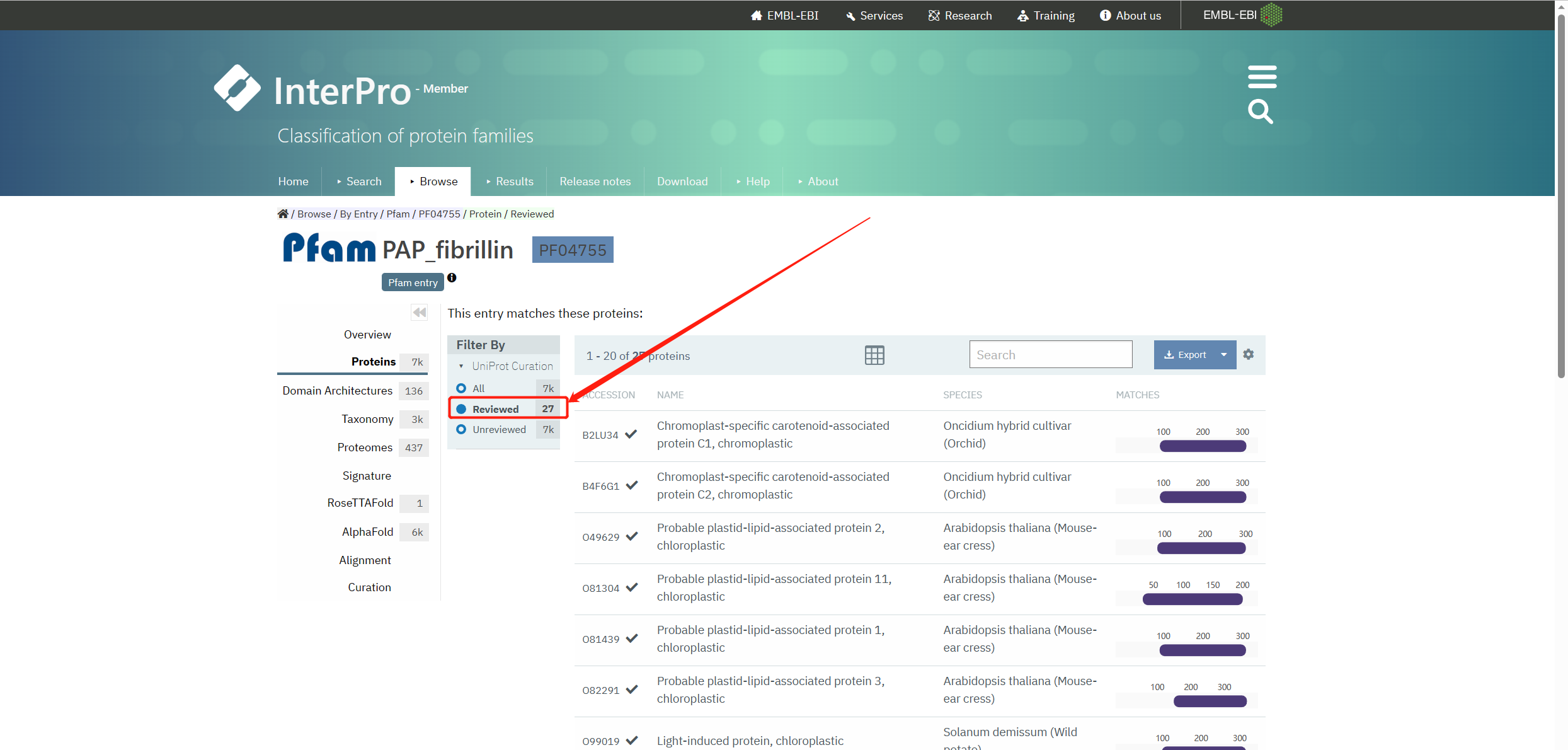 下载Reviewed的PF04755序列 下载Reviewed的PF04755序列 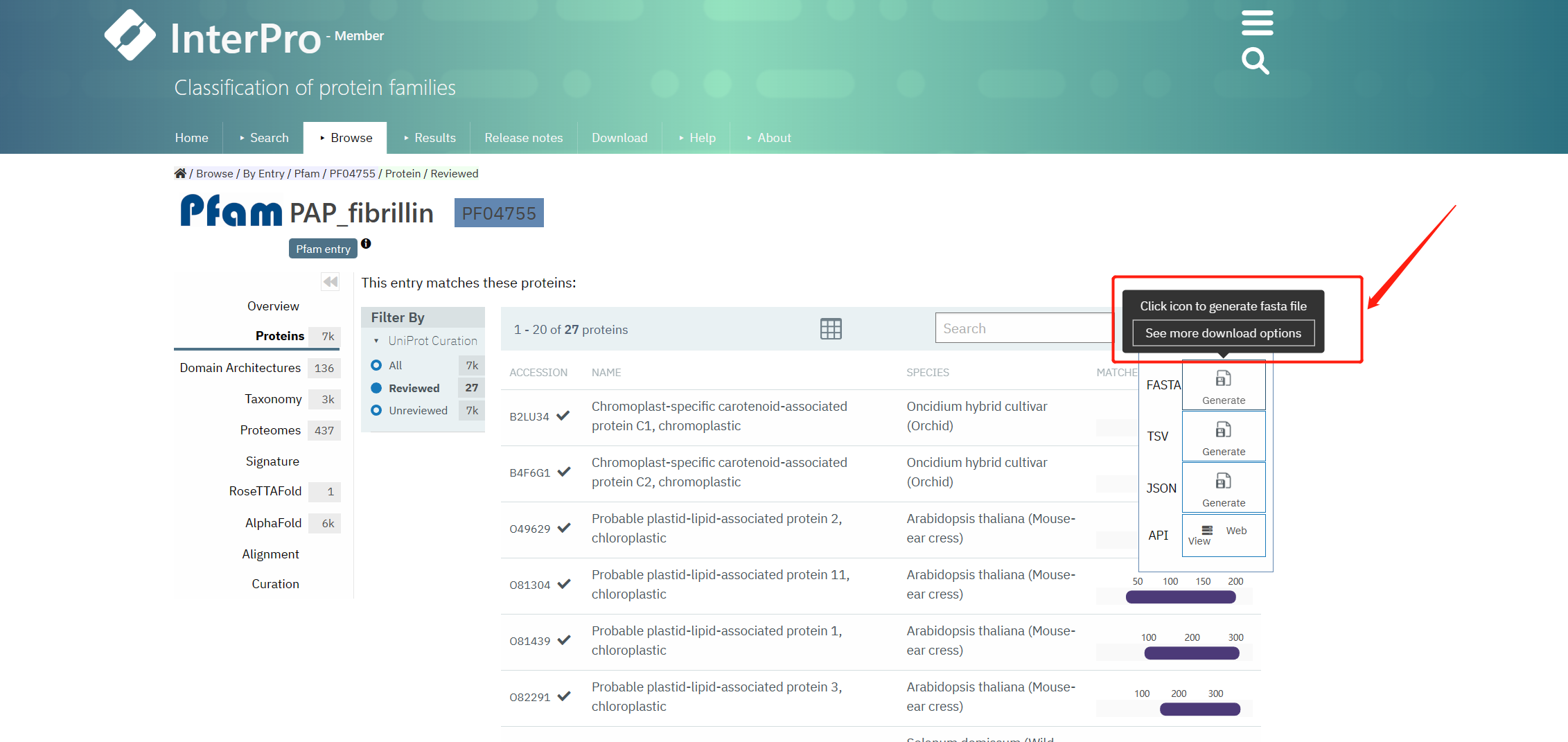 二、同源序列检索预测
二、同源序列检索预测
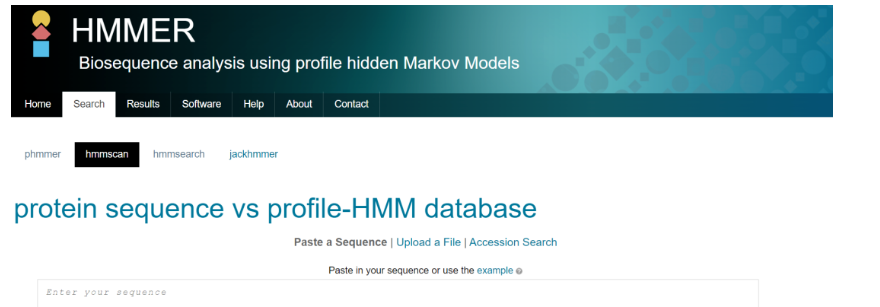
对于同源基因的搜索,很多基因家族的文章都使用HMMER进行检索,也有一些文章是使用BLAST。你任选其中一个即可,都能获得你想要的结果同源基因。在做分析的时候,我将使用Hmmer寻找同源基因的文章分享在公众号中,在评论区有一个大佬对HMMER和BLAST之间的差异给出回答。 这两个方法原理上区别,balstp是基于序列同源性进行打分的,有打分矩阵,hmm是基于隐马尔可夫模型,对序列结构域进行比对。来自“泼皮混混”的评论。 2.1 HMMER同源结构域搜索 2.1.1 Hmmer的安装
安装,主要是使用源码安装或是是使用conda进行安装即可。 conda安装 conda install -y hmmer 源码安装: 官网:http://www.hmmer.org/  任意下载一个版本即可,安装步骤不再做说明。
2.1.2 使用hmmbuild构建.hmm文件 任意下载一个版本即可,安装步骤不再做说明。
2.1.2 使用hmmbuild构建.hmm文件
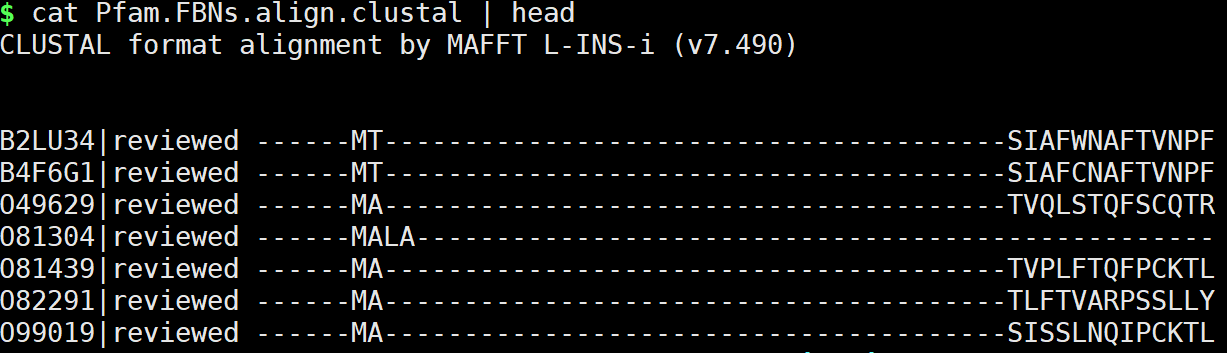
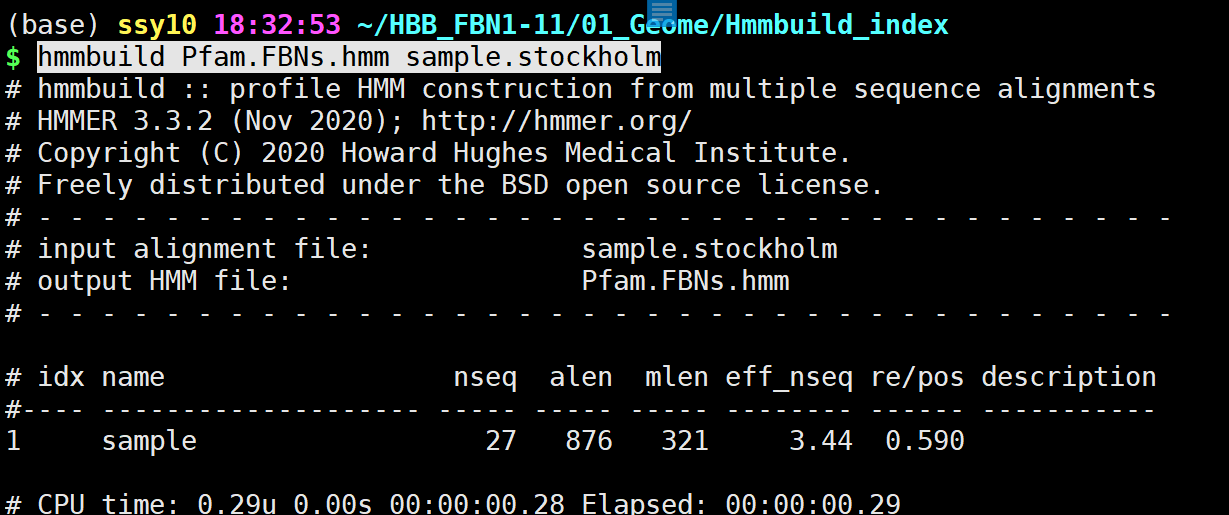
在有些数据库中是有.hmm文件,只需要下载即可。但是,这仅仅只限于有些大数据库。对于我们自己使用,不可能全部都有,这就需要我们自己构建,很多教程到这步就是让你收费了…。 在本教程,讲述其中一种方法吧,希望对大家有所帮助。 hmmbuild构建时,需要使用.sto文件进行构建。因此,我们必须获得.sto文件。 使用mafft软件进行间序列进行对齐 mafft --auto --clustalout ../Pfam_PF04755_reviewed.fasta > Hmmbuild_index/Pfam.FBNs.align.clustal
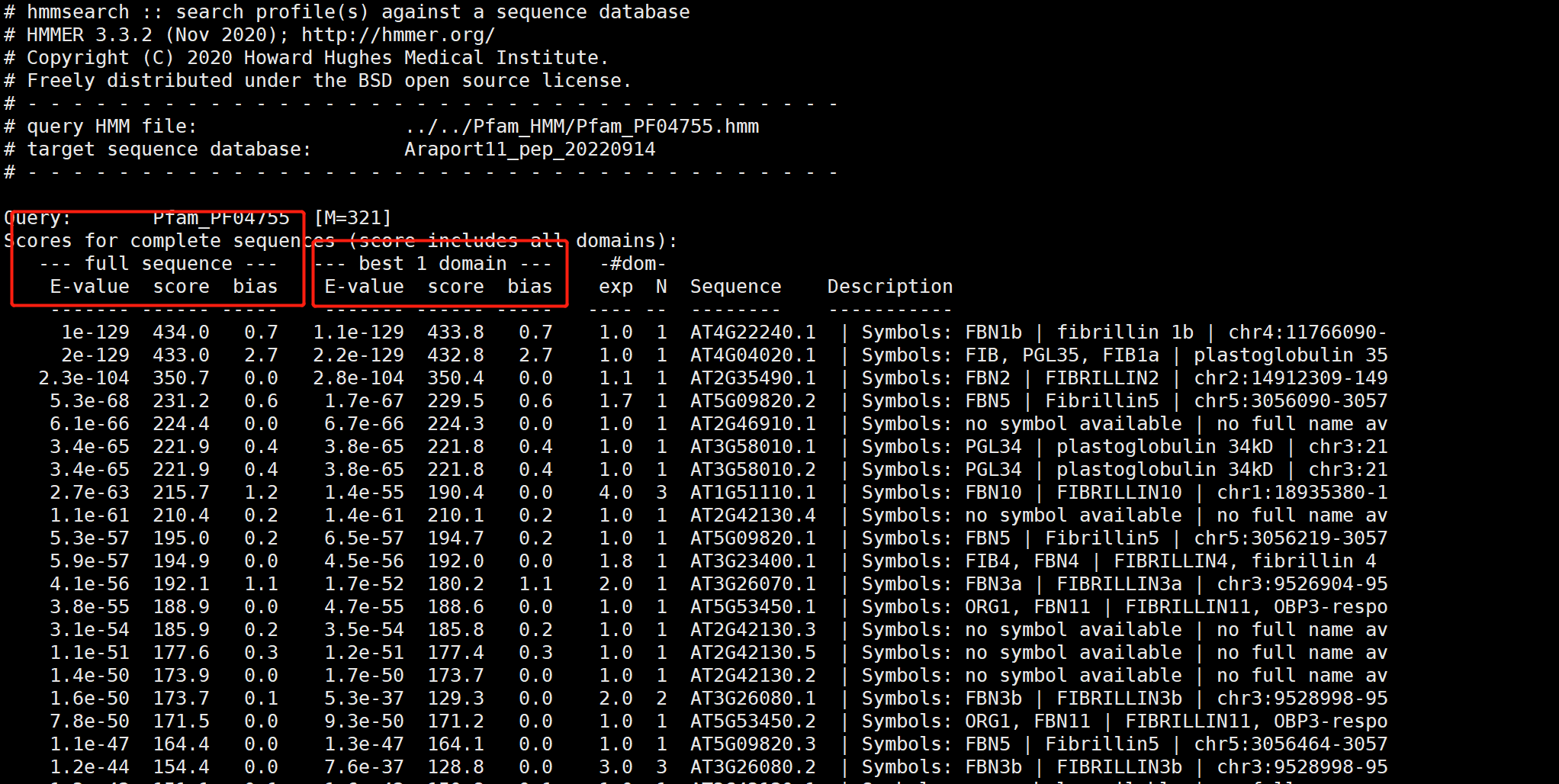
日志:通过Hmmsearch获得同源基因的ID,那么后面对目的同源基因进行进化树、结构域、motif等的分析,这些分析都需使用目的同源基因的序列。 如何获得同源基因序列?? 使用脚本获得使用ggffead获得,需要获得同源基因的.gtf文件等信息。生信工具获得、如TBtools等。对于这步、我们就多做讲解,使用自己拿手的方式获得即可。 问:后面的分析使用核酸序列 or蛋白序列呢?? 答:都可以。 FBN 家族的分析日志。使用Pfam、拟南芥(11)和水稻的FBN家族基因同源搜索马铃薯中的FBN同源基因 ## 水稻中的FBN家族基因 cat all.pep | grep ">" | grep fibrillin |awk -F "|" '{print $1}' | awk -F " " '{print $1}' | sed 's/>//g' > O_sativa.FBN.id.txt ##拟南芥中FBN家族基因 可以在拟南芥网址中的同源搜索,也可以在拟南芥蛋白数据中搜索 cat Araport11_pep_20220914 | grep FBN | awk -F "|" '{print $1}' | sed 's/>//g' > Araport11_FBN.id 2.3 使用TBtools提取目的基因说实话,TBtools确实是个很牛的生信工具,基本可以让你不写代码获得你想要的东西。以及,各种类型的小脚本软件都一直在开发。赞赞!! 2.3.1 TBtools软件的下载 网址:https://github.com/CJ-Chen/TBtools安装。动手运行 2.3.2 提取序列 准备作物所有的蛋白序列文件(or基因文件)目的基因的ID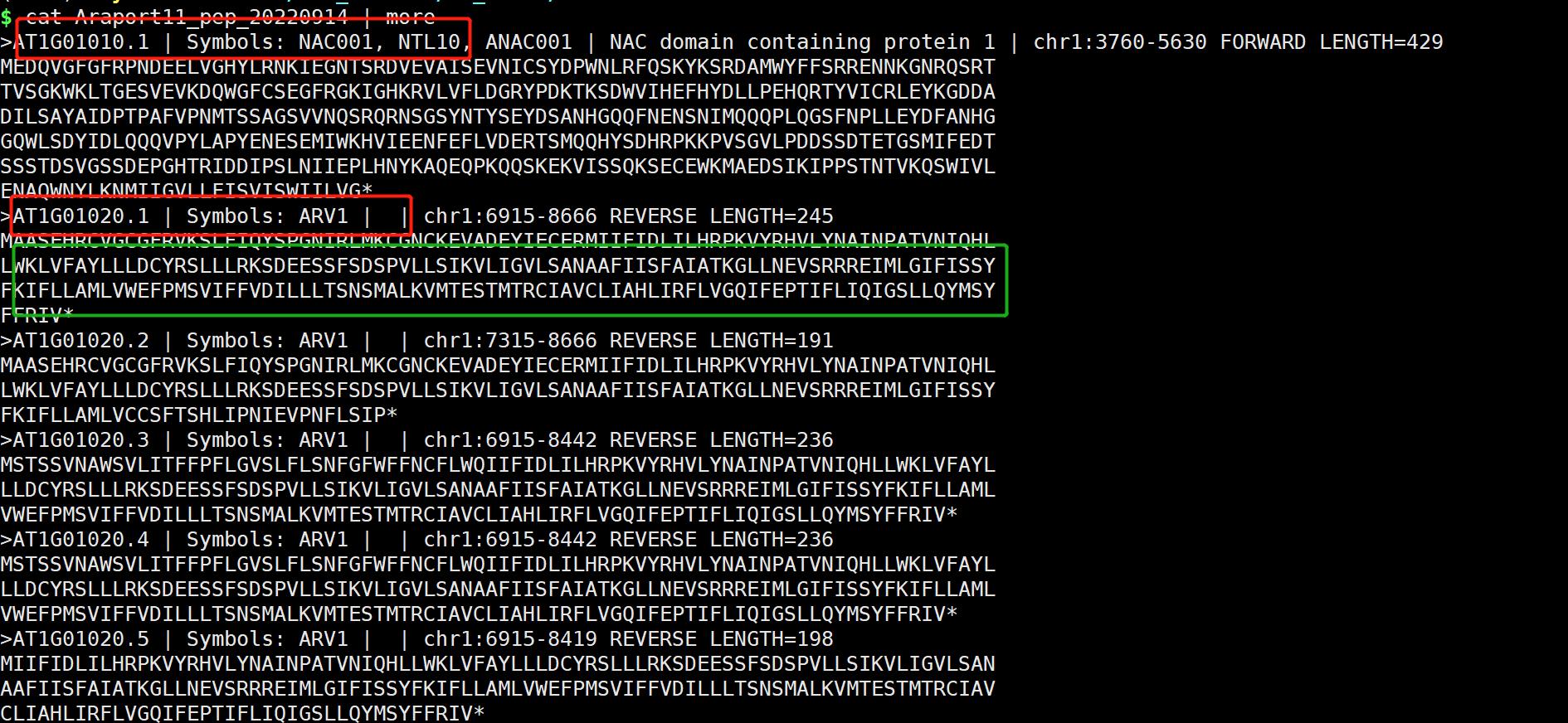 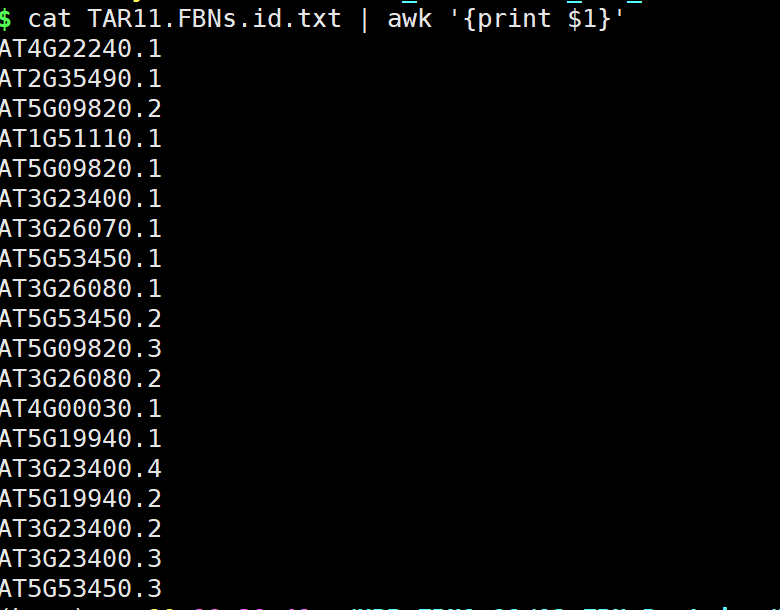 打开TBtools,Fasta Extract or Filter (Qyick) 打开TBtools,Fasta Extract or Filter (Qyick) 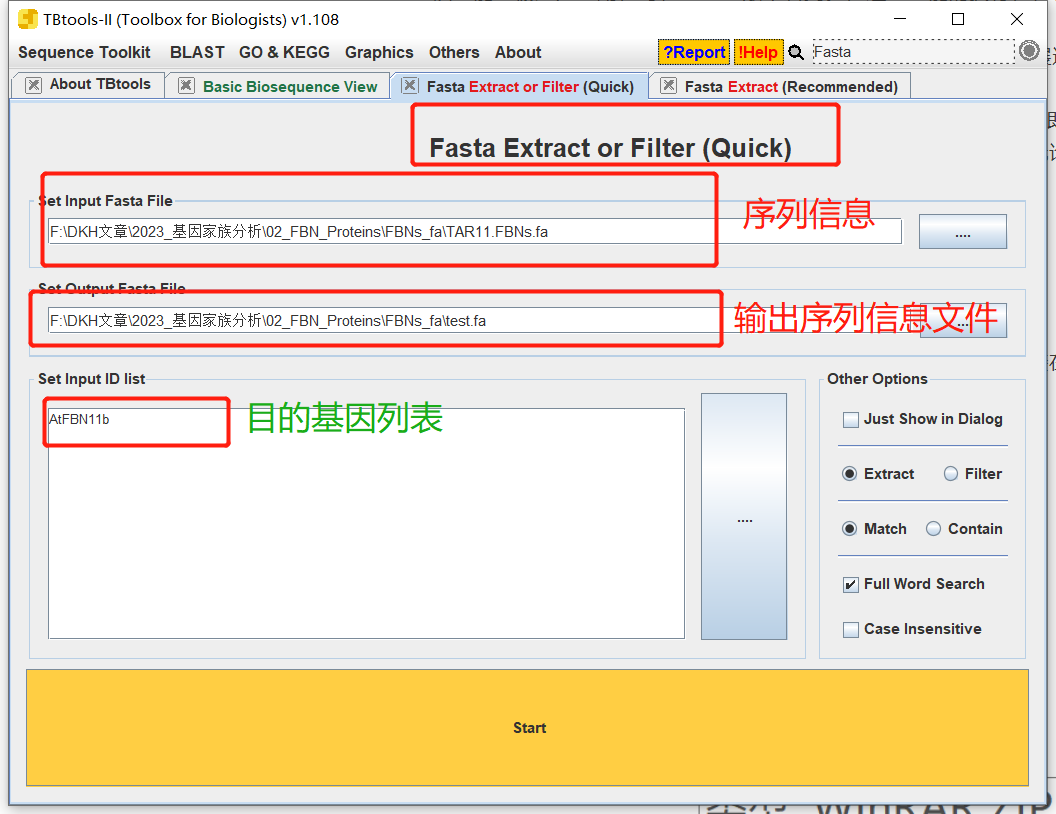 获得结果 获得结果 
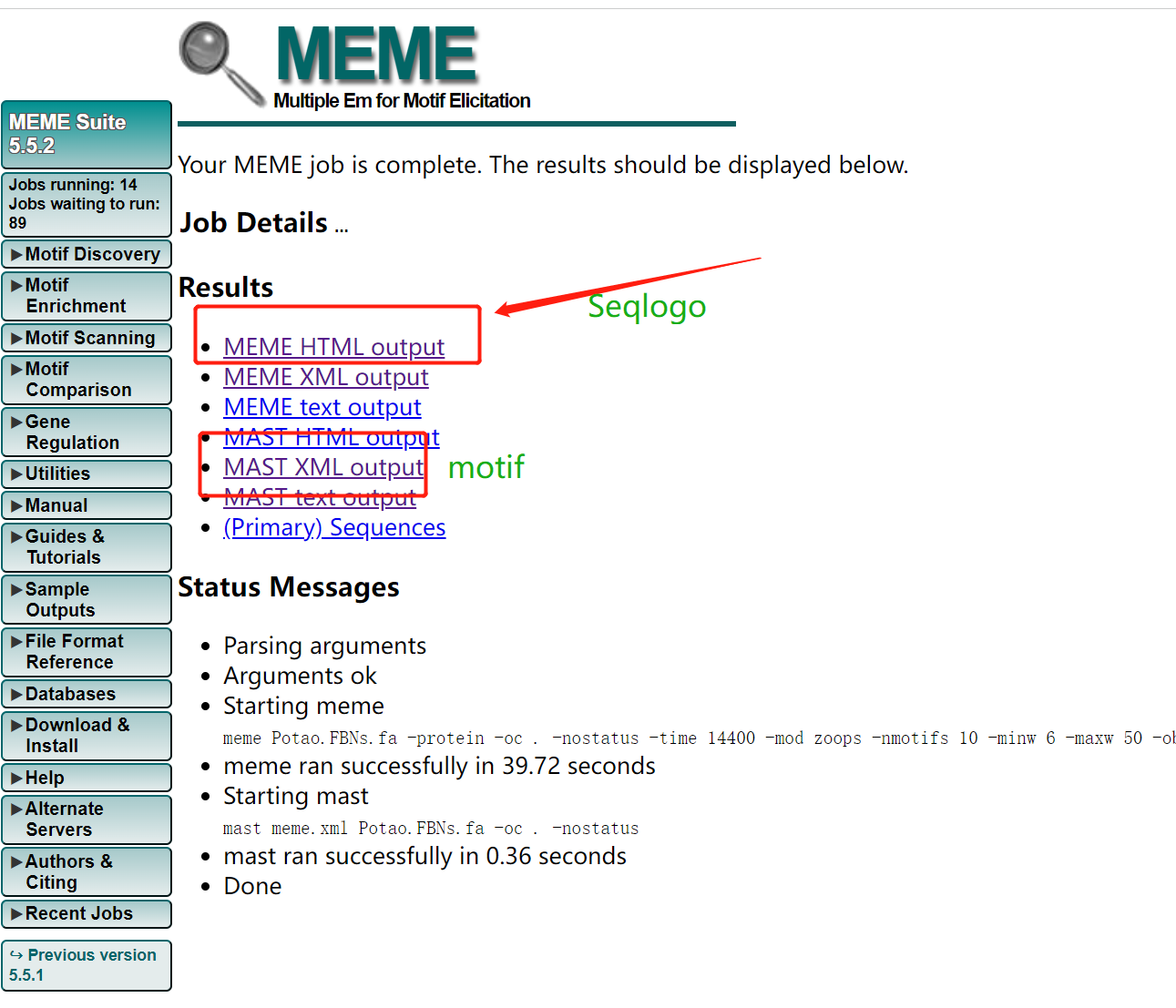
##2.4 目的同源基因motif分析 2.4.1 使用MEME进行motif预测 网址:https://meme-suite.org/meme/tools/meme 上传相关的fa文件,以及修改相关的参数,进行提交
上传相关的fa文件,以及修改相关的参数,进行提交 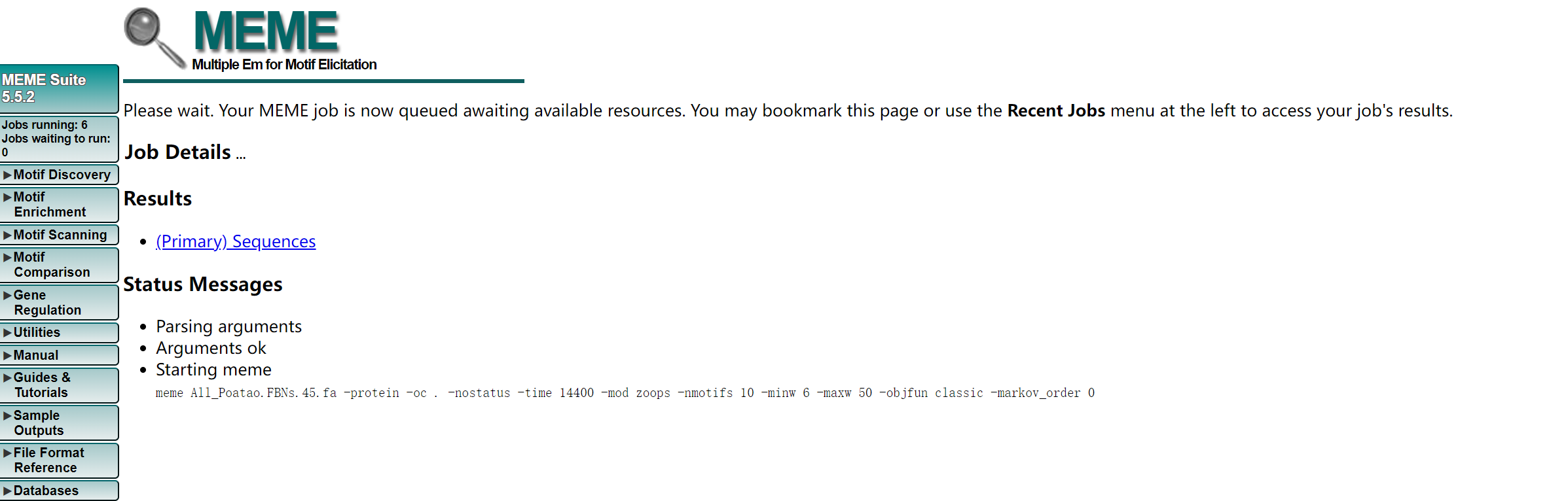 输出结果 输出结果 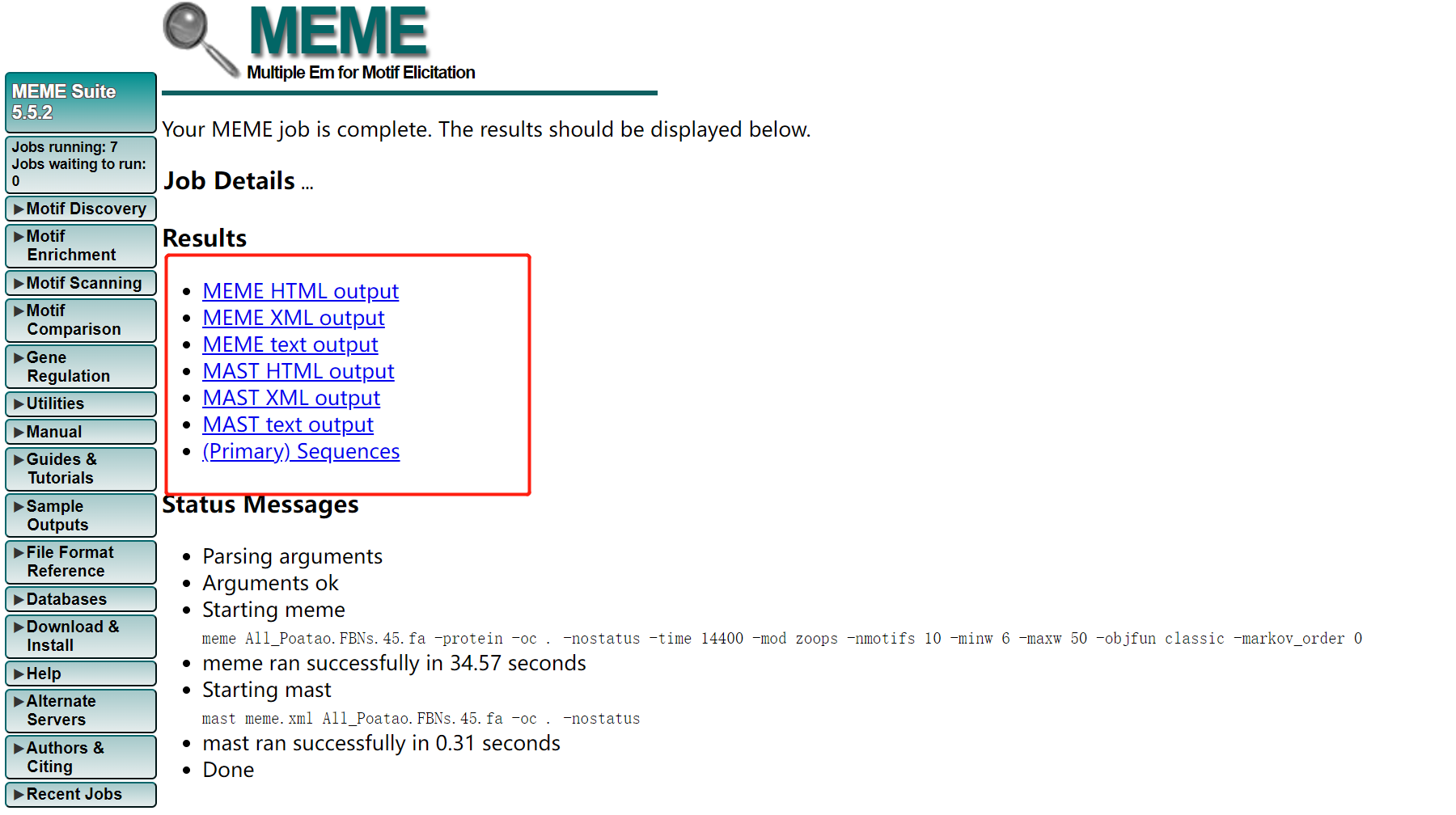 输出结果很快,有以下几个结果文件。
2.4.2 motif可视化 输出结果很快,有以下几个结果文件。
2.4.2 motif可视化
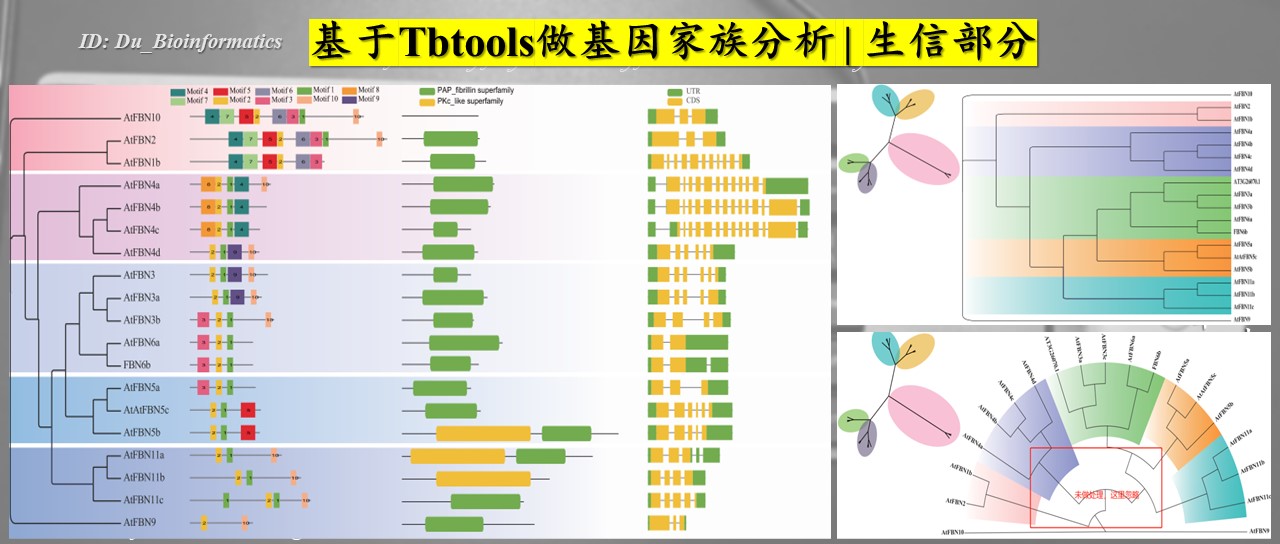
对于motifi分析可以参考一下文章: TBtools | 多图合一至强版教程!进化树 + Motifs + 结构域 + 启动子 + 基因结构 + …,TBtools开发者本人的教程TBtools | 基因家族分析 (进化树、Motifs、结构域)或是本篇教程MEME网址结果可以给我们的seqlogo信息和motif信息。 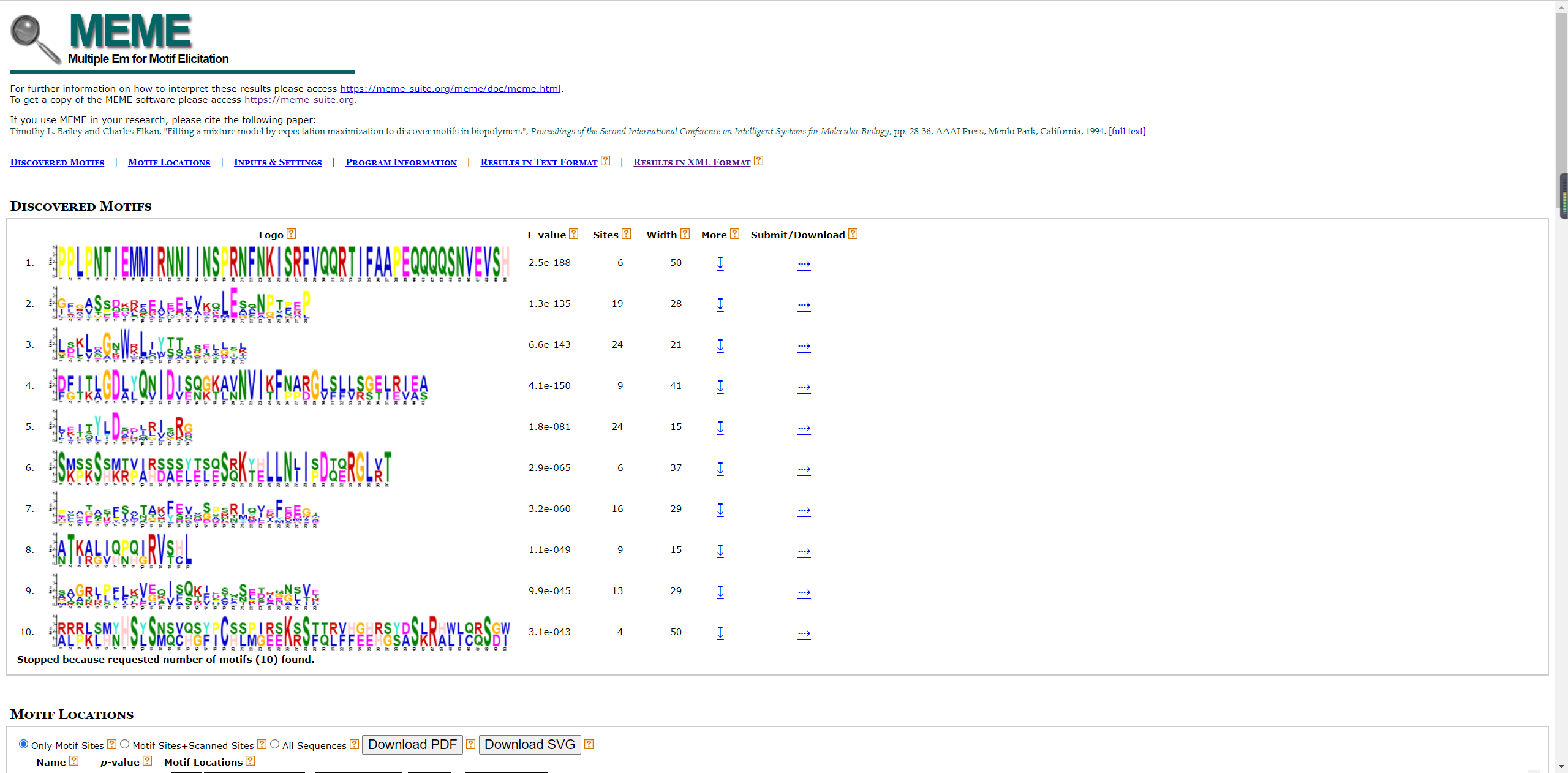 结果文件中就有seqlogo文件信息。 也可以自己的下载后绘制。 结果文件中就有seqlogo文件信息。 也可以自己的下载后绘制。 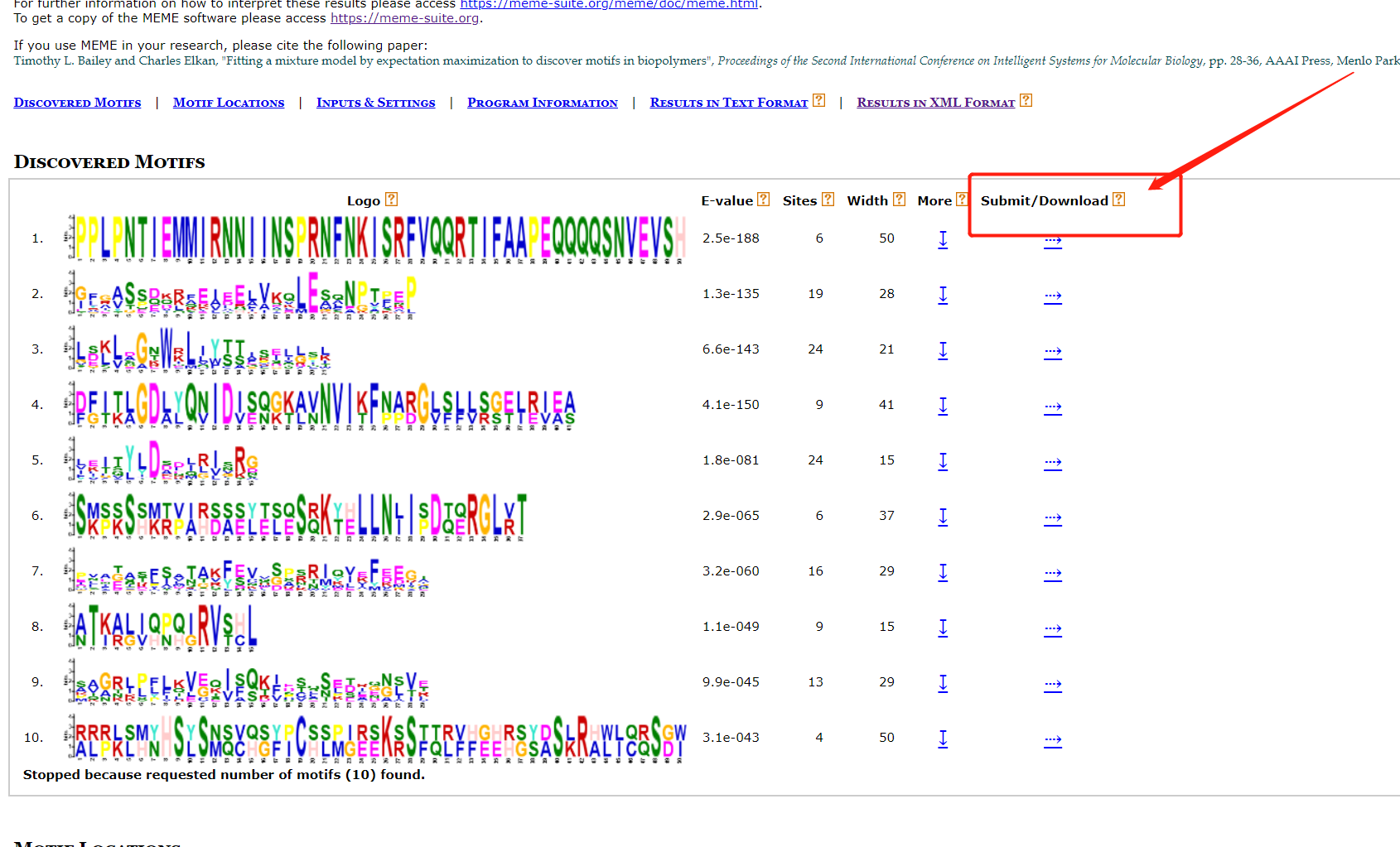 按以下操作即可下载序列。 按以下操作即可下载序列。 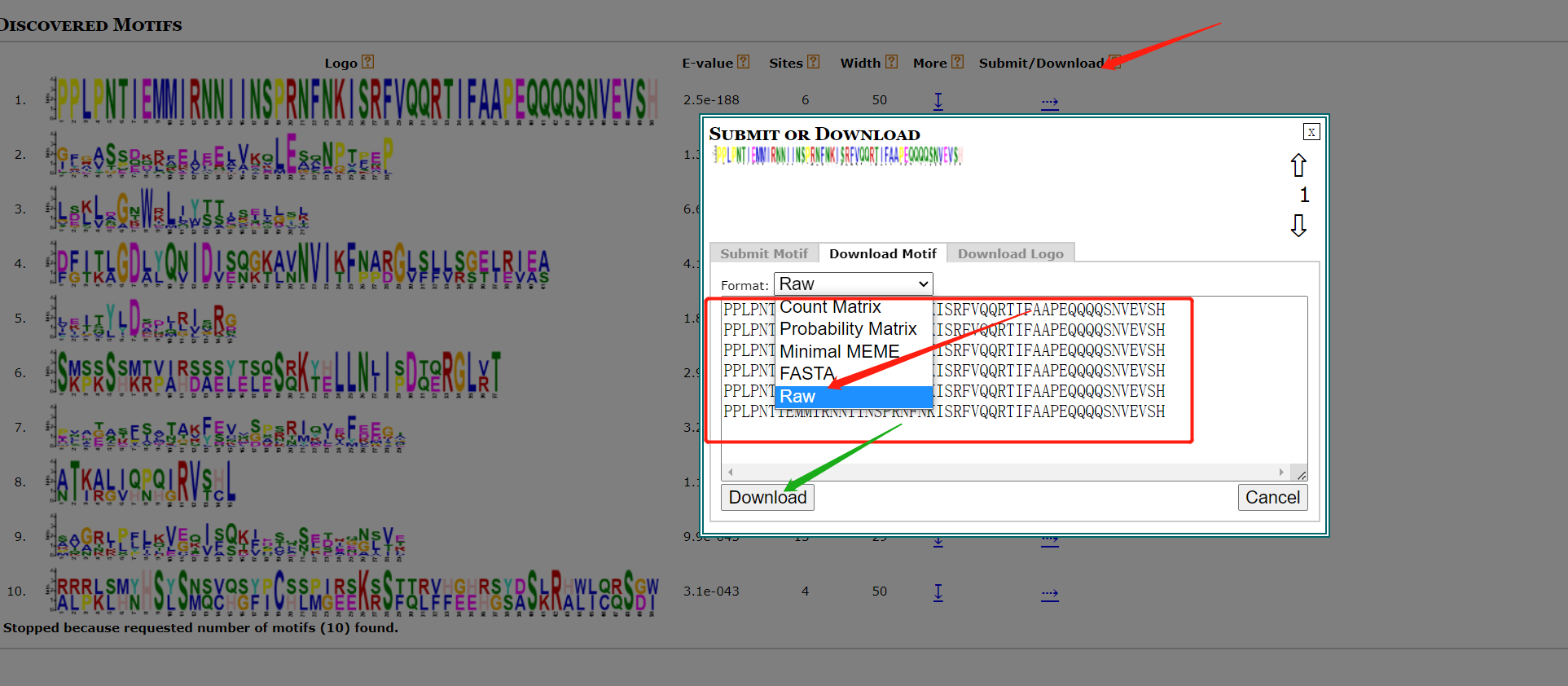 也可以下载已有的seqlogo图片。 也可以下载已有的seqlogo图片。 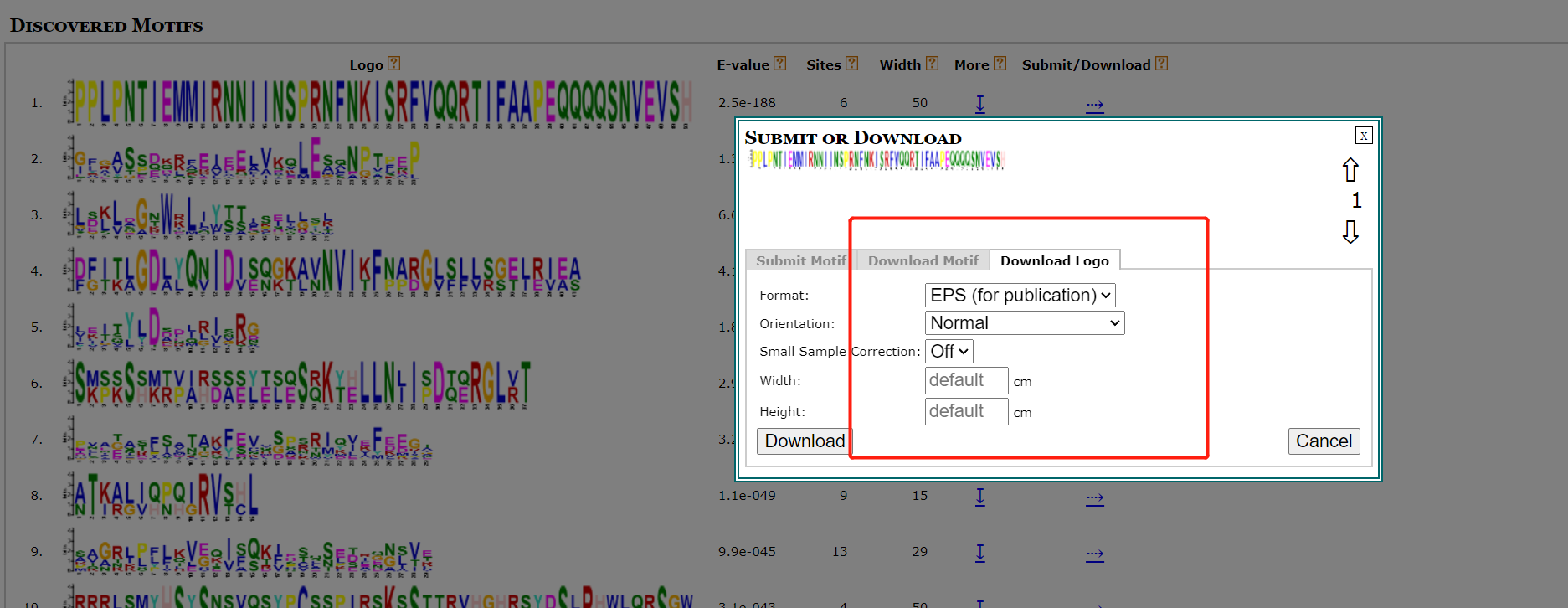 下载后所有的motif序列信息。 下载后所有的motif序列信息。 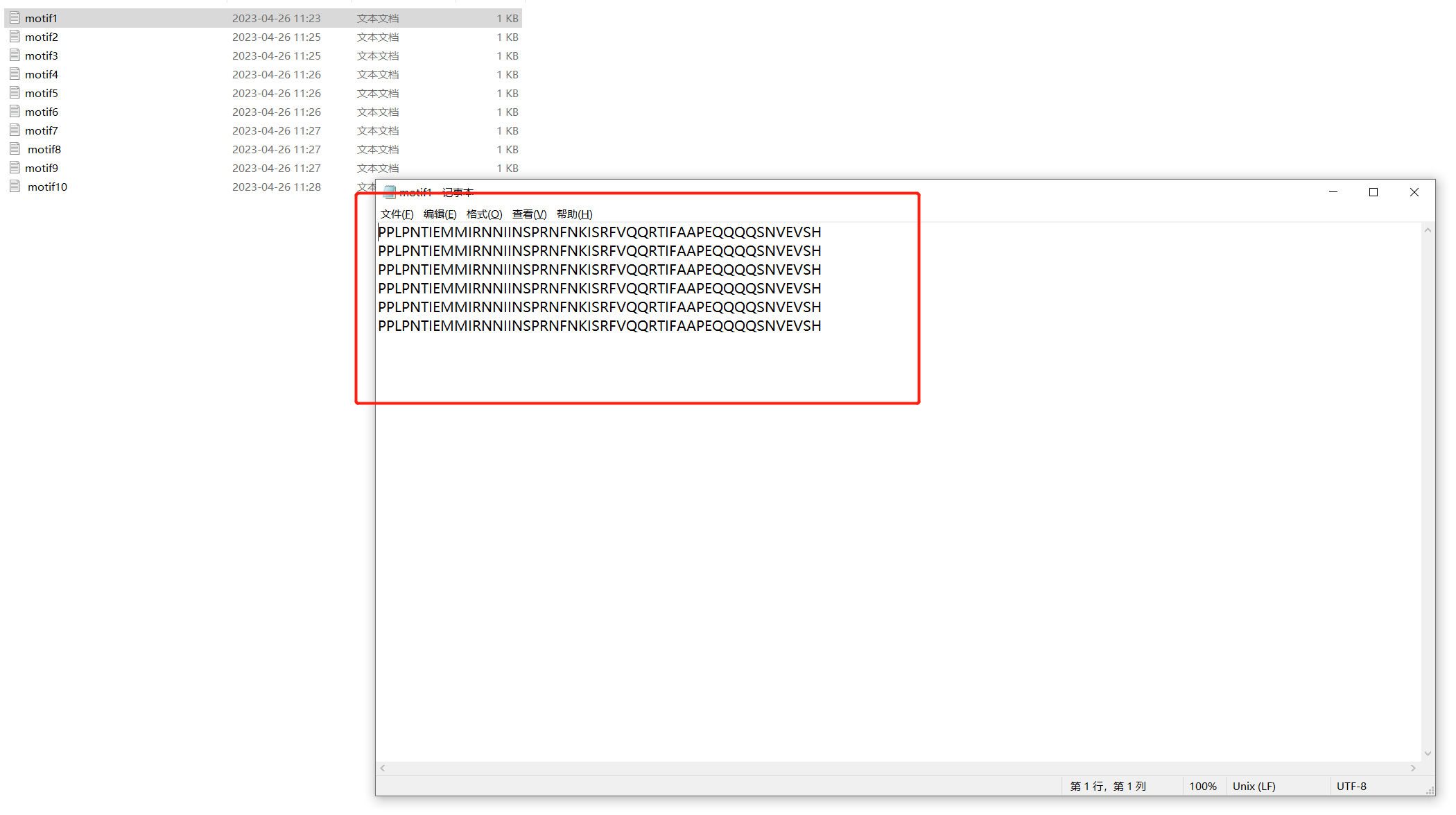 使用R语言对Seqlogo序列进行可视化 这里借用这篇教程,基因结构及motif分析。批量生产Seqlogo可视化。
使用R语言对Seqlogo序列进行可视化 这里借用这篇教程,基因结构及motif分析。批量生产Seqlogo可视化。
我们可以根据自己的motifi数量进行命名,我自己只有10个motif信息。所以命名为motif1-10.txt。 ## 加载所需要的包 library(ggplot2) #BiocManager::install("ggseqlogo") library(ggseqlogo) ## 批量生产文件名 filelist = c(paste0('motif',1:10,'.txt')) filelen B4F6G1 MTSIAFCNAFTVNPFLAAARRSPPPLTPLTSVALSPARKPRILAIFHPRTFPSFRVQAIAEDEWESEKKTLKGVVGSVAL AEDEKTGADLVVSDLKKKLIDQLFGTDRGLKATSETRAEVNELITQLEAKNPNPAPTEALSLLNGKWILAYTSFVGLFPL LGAESLQQLLKVDEISQTIDSEGFTVQNSVRFVGPFSSTSVTTNAKFEVRSPKRVQIKFEEGIIGTPQLTDSIVIPDKVE FFGQNIDLSPFKGVISSLQDTASSVAKTISSQPPIKFPISNSNAQSWLLTTYLDDELRISRADGGSVFVLILESSPLLT >O49629 MATVQLSTQFSCQTRVSISPNSKSISKPPFLVPVTSIIHRPMISTGGIAVSPRRVFKVRATDTGEIGSALLAAEEAIEDV EETERLKRSLVDSLYGTDRGLSASSETRAEIGDLITQLESKNPTPAPTEALFLLNGKWILAYTSFVNLFPLLSRGIVPLI KVDEISQTIDSDNFTVQNSVRFAGPLGTNSISTNAKFEIRSPKRVQIKFEQGVIGTPQLTDSIEIPEYVEVLGQKIDLNP IRGLLTSVQDTASSVARTISSQPPLKFSLPADNAQSWLLTTYLDKDIRISRGDGGSVFVLIKEGSPLLNP mafft比对使用mafft将序列对齐。 mafft test.fa > test.aligend.fa
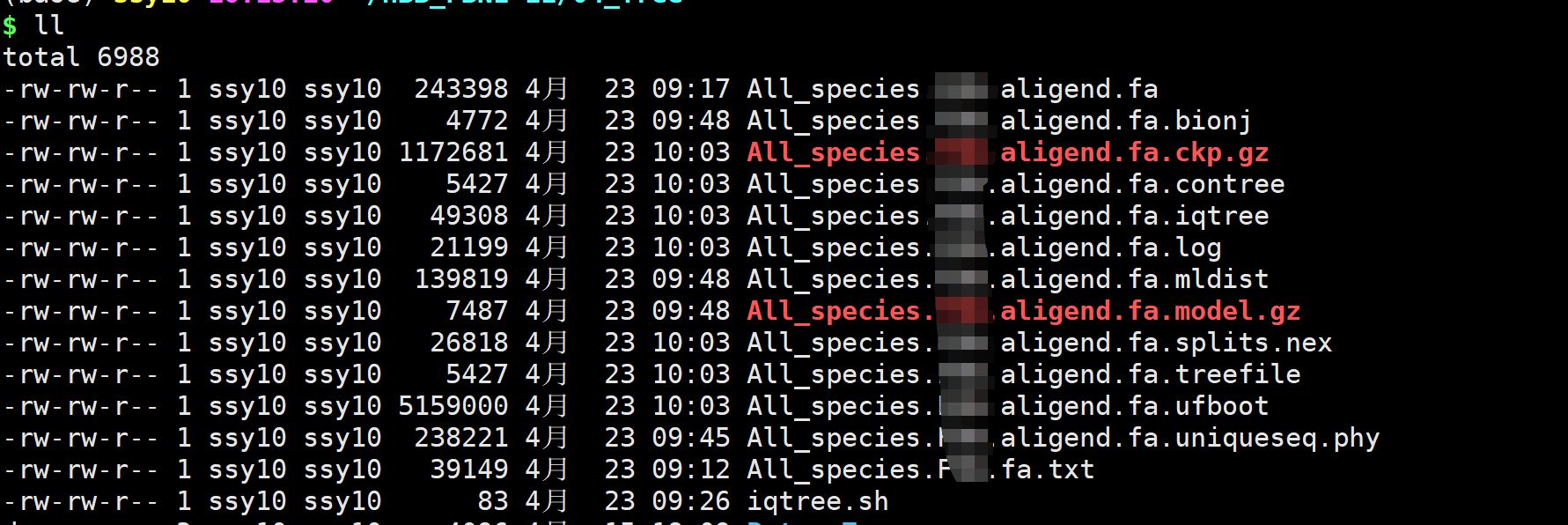
关于iqtree的使用,可以看这篇教程IQ-TREE的使用 - 超快速用极大似然法构建进化树,讲的很详细。 必须参数: -s 输入多序列比对文件 -nt 多线程,AUTO是自动多线程 -bb 1000 指定了要用快速BS法做1000次最终,我们可以获得以下结果文件。 这里,我们使用基迪奥的教程,如何绘制添加分类色块的进化树?,这个教程也是讲解得很详细。 注意:我们这里使用iqtree输出文件test.aligend.fa.treefile作为输入文件。 #载入相关的R包; library(ggtree) library(treeio) library(ggplot2) #读入newick格式的进化树文件; tr = read.newick("test.aligend.fa.treefile") ggtree(tr)
|
【本文地址】

 转换: http://sequenceconversion.bugaco.com/converter/biology/sequences/fasta_to_phylip.php
转换: http://sequenceconversion.bugaco.com/converter/biology/sequences/fasta_to_phylip.php 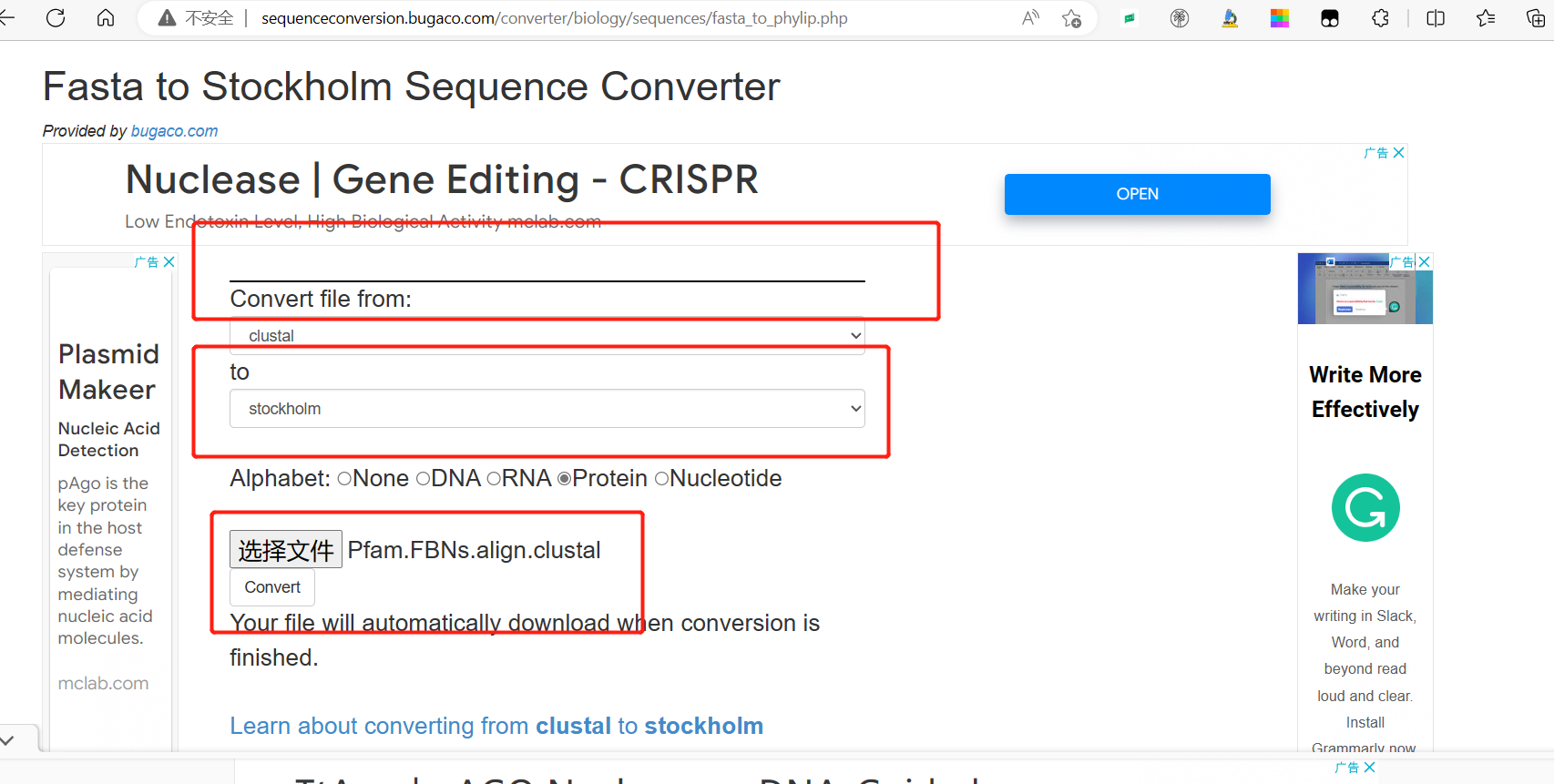
 我们获得对齐后的数据格式。
我们获得对齐后的数据格式。