| GSEA法基因功能富集分析原理详解! | 您所在的位置:网站首页 › 基因count值代表什么 › GSEA法基因功能富集分析原理详解! |
GSEA法基因功能富集分析原理详解!
|
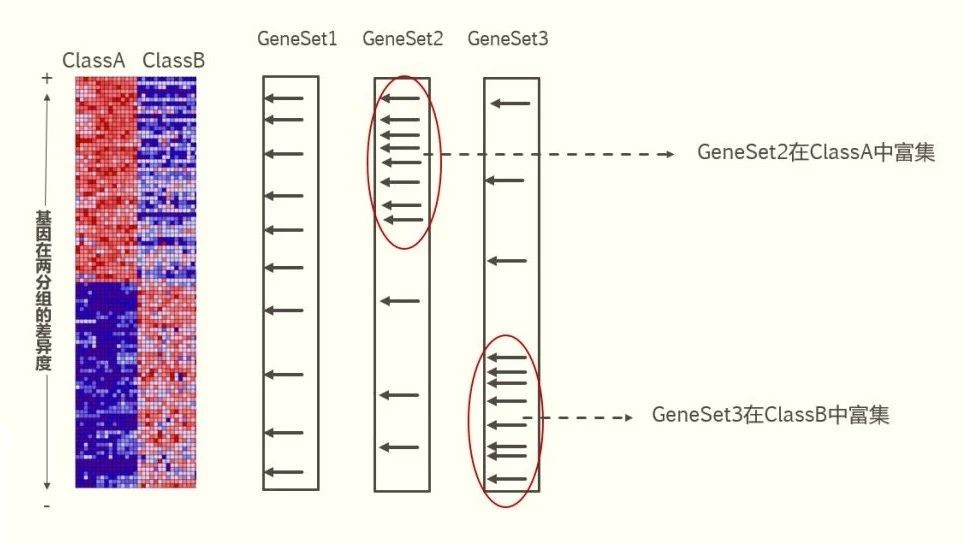
在组学大讲堂之前的一篇微信文章:关注的功能基因集在转录组结果中表现如何?中,跟大家介绍了GSEA(Gene Set Enrichment Analysis)及其分析结果。GSEA是一种基于基因集的富集分析方法,在对基因表达数据分析时,首先确定分析的目的,即选择MSigDB中的一个或多个功能基因集进行分析,然后基于基因表达数据与表型的关联度(也可以理解为表达量的变化)的大小进行排序。然后判断每个基因集内的基因是否富集于表型相关度排序后基因列表的上部或下部,从而判断此基因集内基因的协同变化对表型变化的影响。以上其实就是GSEA的分析原理,光看文字大家是不是还是很疑惑?下面我们就借助一张图来帮助大家更好的理解GSEA的分析原理。 GSEA分析原理: 第一步 基因排序:
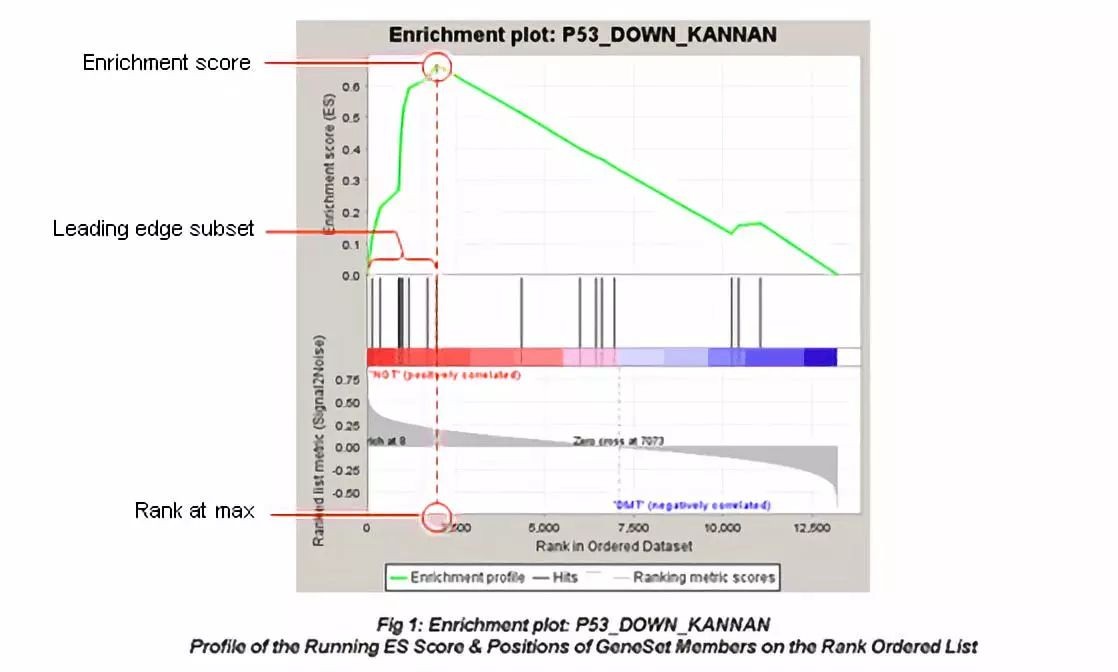
如上图左边的热图所示,GSEA分析的第一步就是利用所有基因的表达数据,然后计算每个基因在两个分组(或者表型)ClassA、ClassB中的差异度(GSEA提供了6种算法,默认方法是signal2 noise,GSEA官网有提供公式),然后按照在两个表型种的差异度从大到小排序,形成一个排好序的基因列表。 第二步 分析基因集是否富集: 这里的基因集,是事先根据功能或者其他的一些原理把很多的基因分类成不同的基因集合,具体一个基因集可以是某一个通路或者go term中的所有基因,也可以是一个miRNA靶标对应的多个基因。GSEA提供了多个分类基因集,在分析数据时,只需要选择不同基因集就可以,当然也可以自己制作基因集。我们可以对每一个小的基因集(GeneSet )里面的基因对应一下上一步排序表里面的位置,例如上图中的GeneSet1 (一个箭头代表一个基因),看基因集里的成员在基因列表里面的分布情况是否均匀,例如GeneSet1就在基因列表中均匀分布,GeneSet2里面的成员主要分布在基因列表的顶部,GeneSet3里面的成员主要分布在基因列表的底部。如果基因集中的成员在基因列表中均匀分布,说明这个基因集不在这两个表型中富集。如果基因集中的成员在基因列表的顶端例如图中的GeneSet2,说明这个基因集在第一个表型ClassA中富集。如果基因集中的成员在基因列表的底部例如图中GeneSet3,说明这个基因集在第二个表型ClassB中富集。 GSEA分析的第三步就是计算基因集的ES值(enrichment score),之后对基因集的ES值进行显著性检验及多重假设检验,从而计算出显著富集的基因集。 以上就是GSEA的分析原理,那么进行GSEA的结果怎样解读呢? GSEA分析结果最常见的是下图:
1、图最上面部分展示的是富集分数(ES, enrichment score)值计算过程,从左至右每到一个基因,计算出一个ES值,连成线。在最左侧或最右侧有一个特别明显的峰值就是基因集表型上的ES值。图中间部分每一条线代表基因集中的一个基因,及其在基因列表中的排序位置。 2、最下面部分展示的是基因与表型关联的矩阵,红色为与第一个表型(class A)正相关,在class A中表达高,蓝色与第二个表型(class B)正相关,在class B中表达高。 3、Leading-edge subset 对富集得分贡献最大的基因成员。若富集得分为正值,则是峰左侧的基因;若富集得分为负值,则是峰右侧的基因。 4、FDR GSEA默认提供所有的分析结果,并且设定FDR |
【本文地址】