| 利用高通量测序对蛋白质 | 您所在的位置:网站首页 › nova1cpu › 利用高通量测序对蛋白质 |
利用高通量测序对蛋白质
|
CLIP也被用于研究Rrm4在丝状真菌黑孢菌(Ustilago maydis)中的RNA结合,证明了CLIP应用于其他生物系统的可能性。此外,对小鼠大脑中RBP Cugbp1的CLIP研究显示,在8型脊髓小脑共济失调中有几种RNA获得功能效应。CLIP也被用来描述小鼠Msy2与piRNAs的相互作用,而Msy-RNA复合物被发现定位于染色质、细胞质RNPs和多聚体。同样使用CLIP,我们也证明了隐性帕金森蛋白DJ-1和胞质酸受体(RARα)的RNA结合活性。
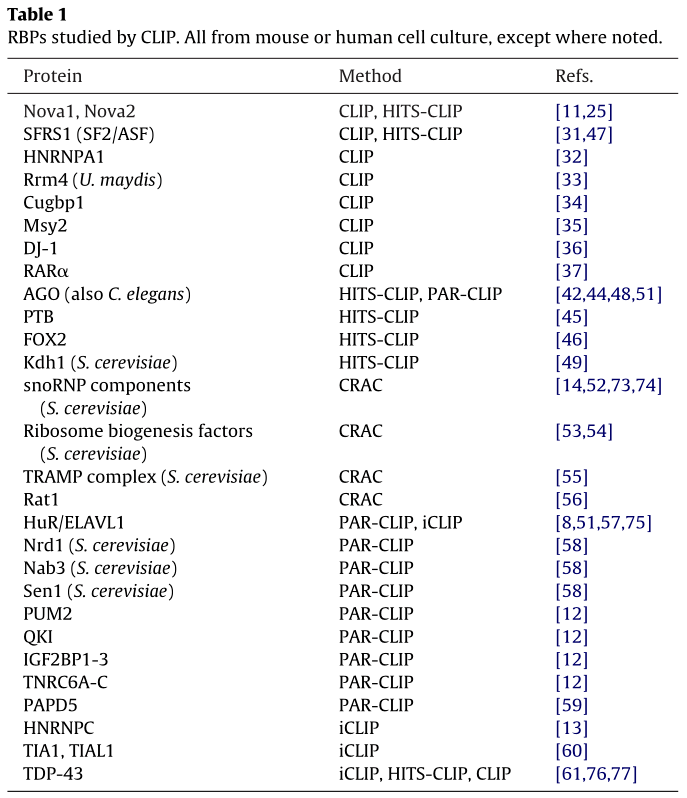
总之,CLIP的一个主要优点是紫外线照射可以应用于不同的生物材料:从培养细胞到小鼠的组织匀质。蛋白质RNA交联的确切机制尚不完全清楚,但被认为是核酸碱基吸收紫外光提高电子的能态,从而形成新的共价键。 High-throughput sequencing of RNA isolated by cross-linking and immunoprecipitation (HITS-CLIP) 传统的CLIP cDNA文库Sanger测序获得的序列数量少,限制了早期基于CLIP的方法的全部潜力。高通量测序方法的出现使交联RNA分子的无偏和详尽的测序得以获得给定RBP的转录组结合模式。在第一项使用HITS-CLIP(也称为CLIP-seq)的研究中,CLIP与NGS结合鉴定了鼠神经特异性拼接因子Nova2的RNA靶标。与上面描述的Nova1相比,Nova2的表达更为广泛,但在其他方面有相似的目标。Darnell和他的同事们遵循了最初的CLIP方法来纯化Nova2-RNA复合物,并从交联RNA片段中生成cDNA。通过454/Roche焦磷酸测序平台对cDNA文库进行测序,得到约41.2万个序列标签,是Nova1 CLIP cDNA sanger测序得到序列数量的1000多倍。大约41%的Nova2片段序列可以唯一地比对到小鼠基因组,主要是mRNA编码基因。该片段标签映射到19000个序列簇,代表潜在的结合位点,在Nova2共识序列中高度富集。构建了全基因组Nova2相互作用图谱,并证实了其在选择性剪接中的作用。进一步分析表明,在3'UTRs中发现大量Nova2标记,提示Nova2在选择性poly(A)位点选择的调控中具有新的功能。 HITS-CLIP成功应用于其他一些RBPs的研究(表1)。Xue和同事对剪接抑制因子PTB进行了HITS-CLIP。他们的分析揭示了富含CU的六聚体结合位点和PTB用来调节外显子包含或跳跃的策略。对人类剪接因子FOX2和SFRS1的HITS-CLIP研究对理解剪接调控机制有重要贡献。FOX2被证明以位置依赖的方式调控外显子增加或跳跃,而SFRS1通过调控一个功能多样的转录子子集对基因表达有广泛的影响。 HITS-CLIP也被应用于不同的模式生物。利用HITS-CLIP确定了酿酒酵母中Kdh1的RNA靶标。交联后,作者使用亲和树脂而不是抗体来纯化TAP标记的1 RNA复合物,发现Khd1调节FLO11的不对称表达,从而决定了丝状生长过程中子细胞的命运。 Cross-linking and analysis of cDNA (CRAC) 带有抗体的RBPs的免疫沉淀需要自然条件。使用不依赖蛋白质-肽相互作用的亲和树脂可以在变性条件下进行严格的洗涤步骤。这种方法被称为CRAC (Cross-linking and analysis of cDNA),由David Tollervey的实验室开发,用于研究酵母中snoRNP组分(Nop1、Nop56、Nop58和Rrp9)的RNA-蛋白互作。在用IgG琼脂糖纯化并用TEV蛋白酶首次洗脱后,TAP标记的Nop1、Nop56、Nop58和Rrp9 RNP复合物被氯化胍变性并结合到镍树脂上,随后进行广泛的洗涤和洗脱。经过温和的RNAse处理后,对恢复的RNA片段进行连接和cDNA扩增。对与snoRNP蛋白相关的RNA的分析显示了非常高的信噪比,并确定了这些蛋白在U3 RNA上的特定结合位点,为U3 snoRNP的结构提供了深入了解。 Photoactivatable-ribonucleoside-enhanced cross-linking and immunoprecipitation (PAR-CLIP) 大多数CLIP方法依赖于254 nm照射诱导的蛋白RNA交联,这导致相对较低的交联效果,用纯化蛋白和放射性标记RNA估计在1%到5%之间。由Thomas Tuschl和他的同事开发的PAR-CLIP,通过使用光反应核苷类似物(4-硫脲,4-SU,和6-硫鸟苷,6-SG)在新陈代谢中标记细胞,克服了这个问题。这些修饰的核苷很容易被哺乳动物细胞吸收,没有任何明显的毒性,并进入新生的RNA。蛋白质与RNA的交联是通过365nm UV照射活细胞完成的。以RBP IGF2BP1为例,研究表明,365nm的交联加上RNA的4-SU或6-SG标记,比在254 nm紫外光下交联有效得多。 与原始的CLIP方案类似,在RBP RNA复合物免疫沉淀和RNAse处理后,将回收的RNA片段连接到接头上,进行逆转录,PCR扩增并在Illumina平台上测序。当序列与参考转录组比对时,进行了有趣的观察。当交联时,4-SU或6-SG经常导致cDNA序列中的T到C或G到A转换,提供了从非交联序列中筛选交联的特征。此外,代表假定结合位点的排列序列簇可以根据T到C的转变数目进行排序。 类似于HITS-CLIP,PAR-CIP已被应用于研究几个RBPS和RNPs的RNA靶标。在最初的PAR-CHIP研究中,作者鉴定了几个被深入研究的人类蛋白质的结合位点和调控序列,即PUM2、QKI、IGF2BP1-3,AGO/EIF2C1-4和TNRC6A-C。这些发现包括确定的结合位点的序列基序,观察外显子和内显子以及编码和非编码转录区域的不同偏好,这与之前对这些蛋白质的研究基本一致。最近,三篇关于人类RBP HuR/ELAVL1转录全调控相互作用的独立报道用PAR-CLIP发表。总之,这些报告显示,HuR与20000多个结合位点结合,大多数位于3'-UTR区域,但也与内含子结合。另外的发现是,HuR稳定了其靶mRNA,调节了选择性剪接,并抑制miR-7的表达。 PAR-CLIP也成功地应用于酵母,其生长在添加4-硫氧嘧啶的培养基中。研究表明,RBPs Nrd1、Nab3和RNA解旋酶Sen1与许多意想不到的非编码RNA靶标和tRNA基因转录本结合,而作者还证明,Nrd1和Nab3的结合靶标在应激条件下会发生变化。此外,PAR-CLIP被用于补充生化研究,并定义了PAPD5的结合基序,PAPD5是非典型的poly(A)聚合酶,表明PAPD5催化不同类型的RNA底物。 综上所述,PAR-CLIP的主要优点包括结合核苷酸分辨率的结合位点的确定和基于诊断核苷酸转移的数目对假定的结合位点(序列簇)进行排序的能力。 Individual-nucleotide resolution UV cross-linking and immunoprecipitation (iCLIP) 3 展望 新一代测序技术极大地促进了RNA蛋白相互作用的研究。近年来发展的各种CLIP技术允许研究人员选择适当的方法来研究感兴趣的生物系统中的RBP。除了分析活性稳定的RNA相互作用外,还可以研究具有酶活性的RNA相互作用蛋白,这些蛋白可能只是短暂地与RNA相互作用。鉴定聚合酶、解旋酶、核酸酶和RNA修饰酶的RNA靶标已成为可能,并将扩大未来蛋白质-RNA相互作用的范围。 为了收集和整合海量已发表的CLIP数据,建立了多个在线开放存取数据库。CLIPZ为实验确定的RBPs结合位点提供了一个数据库和分析环境,而starBASE则允许从Ago HITS-CLIP数据集推断microRNA mRNA的相互作用。doRiNA (http://dorina.mdc-berlin.de)存储并集成了RBPs和microRNAs的转录全结合位点数据,并允许组合查询搜索。 在接下来的几年里,挑战将是将高分辨率蛋白-RNA相互作用图与其他高通量数据结合起来,以阐明系统水平上细胞蛋白-RNA相互作用的功能和动态。一些新颖和创新的高通量方法允许评估RNPs对mRNA和蛋白表达的影响。NGS可用于全转录组RNA结构的探测。综上所述,与NGS相结合的CLIP方法的技术进步为以前所未有的深度探索功能蛋白RNA相互作用提供了基础,并已经提供了新的和意想不到的生物学见解。目前该领域的动态将极大地增强我们的知识,有助于更全面地理解RNPs的细胞功能,目的是阐明生长、分化和疾病的转录后调控机制。
|
【本文地址】
 返回搜狐,查看更多
返回搜狐,查看更多