| 头疼的org.Hs.eg.db还是没有下载好 | 您所在的位置:网站首页 › 10转2股派2元好不好 › 头疼的org.Hs.eg.db还是没有下载好 |
头疼的org.Hs.eg.db还是没有下载好
|
首先是运行代码 if (!require("BiocManager", quietly = TRUE)) install.packages("BiocManager") BiocManager::install("AnnotationDbi")这条代码适用于Bioconductor version: Release (3.16)以及R (version “4.2”) 注意:要根据自己的R的版本进行选择。 但由于我的R比较旧,是4.0版本的,适用于Bioconductor version: Release (3.12) 所以代码是 BiocManager::install(version = '3.12') library("BiocManager") BiocManager::install("org.Hs.eg.db")但是之后安装又遇到bug: 错误: 句法分析器1行里不能有多字节字符–停止执行写入异常,显示写入无权限退出时状态非0本来想着的是改变下载R包的地方,但似乎没有效果,于是又在百度上疯狂搜索。搜到两种主要方法: 先下载依赖包DBI和AnnotationDbi,再下载org.Hs.eg.db。参考【下载org.Hs.eg.db遇到的坑(已解决)】 BiocManager::install('DBI',dependencies=TRUE) #dependencies为可选参数 #成功 BiocManager::install("AnnotationDbi",dependencies=TRUE) #成功 BiocManager::install('org.Hs.eg.db',dependencies=TRUE,INSTALL_opts = c('--no-lock')) 以管理员方式打开R,再进行下载包(解决不能写入问题)找找有没有之前下载但没有成功使用的同名软件,建议用everything,方便快捷、全面找到目的文件/软件。参考【安装程序包‘xxxx’时退出狀態的值不是0】更换合适的repos
还是会报错…… 所以目前状况就是…… org.Hs.eg.db包下载完整,但是每次到最后始终是反映为非0状态,导致安装失败…… 重启后发现更糟糕,BiocManager链接都上不了……,尝试以下方法(from 建明老师) > options(download.file.method="libcurl") > options(url.method="libcurl") > options()$repos CRAN "@CRAN@" > options()$Bio_mirror NULL > options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/") > options("repos"=c(CRAN="/CRAN/")) > options()$repos CRAN "/CRAN/" > options()$BioC_mirror [1] "https://mirrors.ustc.edu.cn/bioc/" > if (!requireNamespace("BiocManager",quietly=TRUE)) + install.packages("BioManager") > BiocManager::install(c("org.Hs.eg.db","hgu133plus2.db"),ask=F,update=F) #报错信息 Warning messages: 1: In file(con, "r") : URL 'https://bioconductor.org/config.yaml': status was 'Couldn't connect to server' 2: In file(con, "r") : URL 'http://bioconductor.org/config.yaml': status was 'Couldn't connect to server' 找到非零状态含义
更具体的非零退出状态(https://www.jianshu.com/p/213361f11ae0) 1.包加载安装过程中编译不能通过,因此执行安装加载通过不了。 2.library中路径有中文字符出现 3.library,没有指定安装成功。 4.缺少包的依赖。 5.依赖包冲突:依赖包版本过低或过高,需要remove或delete 6.R的依赖包的镜像不在国内,需要翻墙获取依赖包 7.使用R语言的人对Rstudio和RGUi没有正确安装,导致无法加载到路径中去 8.安装部分R语言包需要以管理员身份运行软件,使得相关依赖包能够写入到library中 解决方法: 一、手动下载依赖包 二、加入参数 dependencies=TRUE, INSTALL_opts = c(‘–no-lock’) 交代清楚依赖关系,并返回无法锁定目录 |
【本文地址】
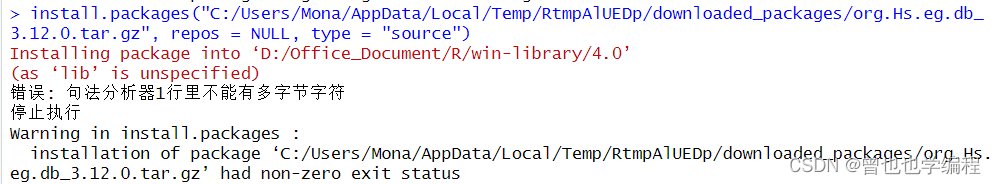 结果还是…… 有点要崩溃 再试一下删除重复的org.Hs.eg.db_3.12.0.tar.gz文档(使用everything) 之后重启,再输入代码下载
结果还是…… 有点要崩溃 再试一下删除重复的org.Hs.eg.db_3.12.0.tar.gz文档(使用everything) 之后重启,再输入代码下载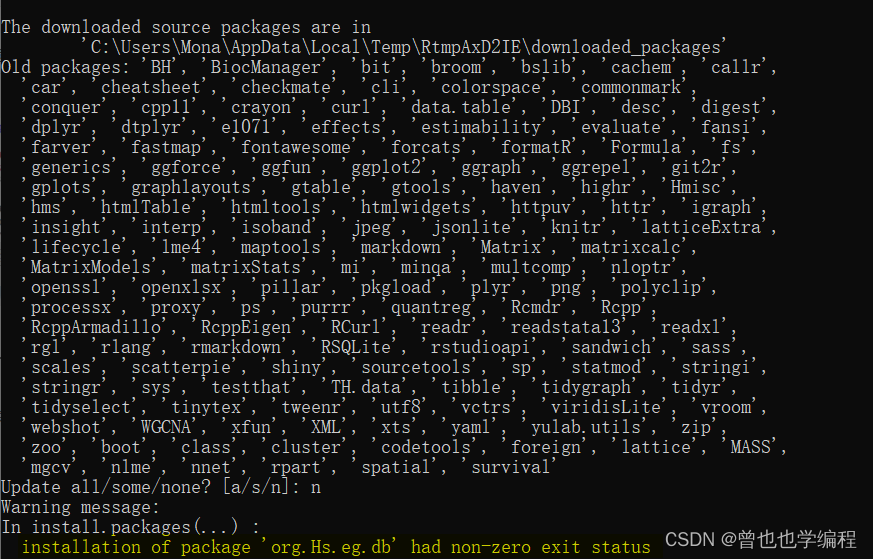
 非零退出状态表示:在这种情况下无法安装软件包。尝试不要一次性安装目的包,而是先安装依赖包的方法,依次安装,也许可以达成目的。 使用自动化安装依赖包,加入参数 dependencies=TRUE,即自动安装依赖包
非零退出状态表示:在这种情况下无法安装软件包。尝试不要一次性安装目的包,而是先安装依赖包的方法,依次安装,也许可以达成目的。 使用自动化安装依赖包,加入参数 dependencies=TRUE,即自动安装依赖包