| 使用evolview美化进化树 | 您所在的位置:网站首页 › 进化树美化网站下载 › 使用evolview美化进化树 |
使用evolview美化进化树
|
1.evolview地址及使用
进化树美化,evolview官方地址:https://evolgenius.info//evolview-v2/#login 要求使用以下浏览器打开,不支持IE浏览器和360浏览器:
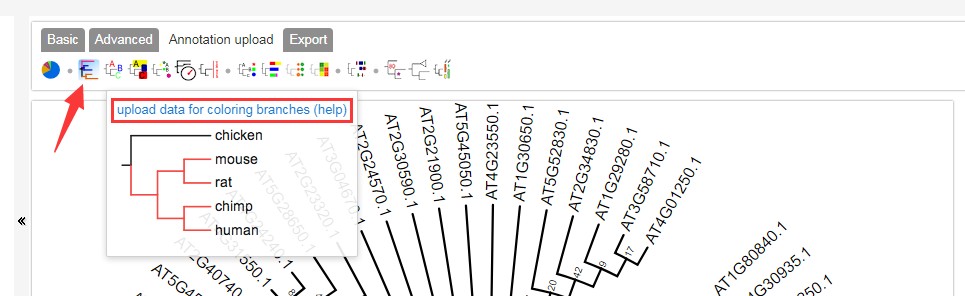
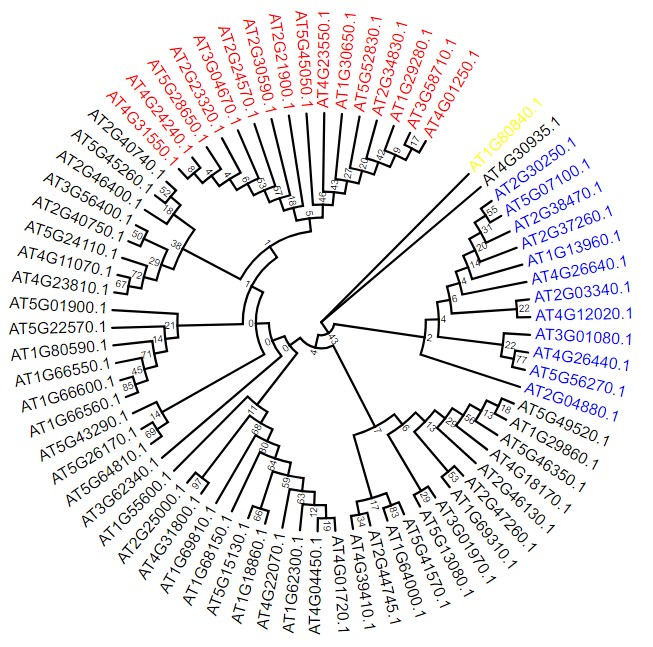
Evolview进化树美化的初级使用可参考:https://www.omicsclass.com/article/590,示例进化树:WRKY_domain_confirmed.nwk 2.1 进化树分支添加颜色可点击以下方箭头处添加分支颜色,可点击下图红框框处查看官方详细配置说明:
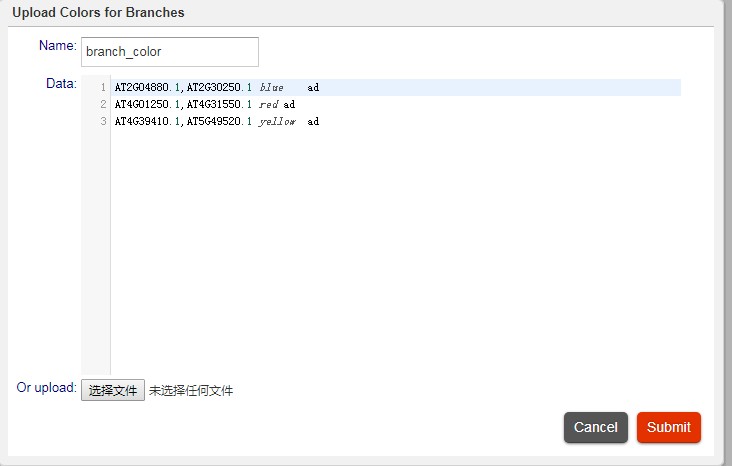
然后在配置文件里面输入,需要添加颜色的分组信息,如下: AT2G04880.1,AT2G30250.1 blue adAT4G01250.1,AT4G31550.1 red adAT4G39410.1,AT5G49520.1 yellow ad第一列为,从一个基因到另一个基因之间,表示范围;第二列,为设置颜色,最后一列 ad表示为所有枝设置颜色;列和列之间注意用tab分隔开;
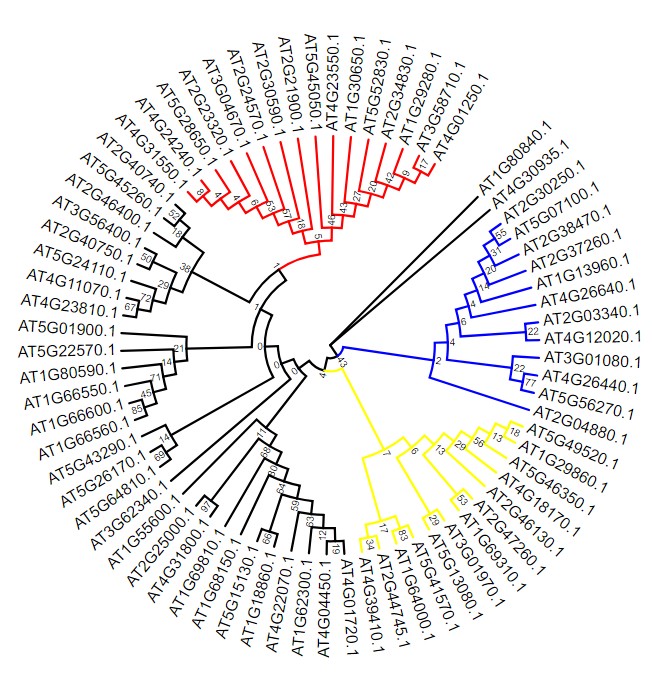
然后提交,就可以得到进化树分支颜色设置后的进化树:
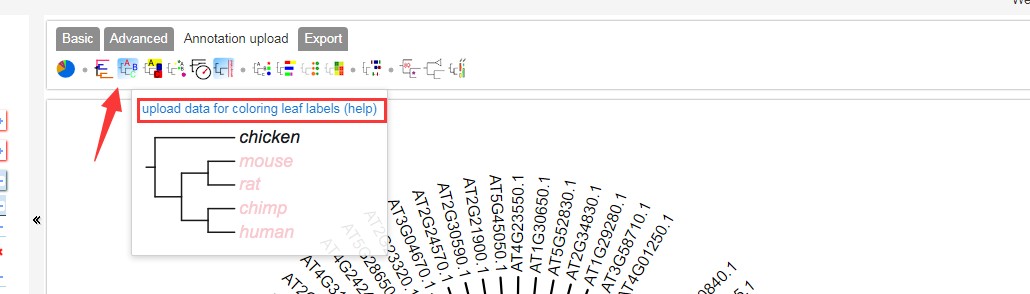
设置标签颜色,详细方法参考:https://www.omicsclass.com/article/631,可点击下图红框框出查看官方详细配置说明:
这是设置的配置文件内容如下 ,与分支颜色设置相似,也支持单独基因设置颜色(最后一行);点击红色箭头处,然后将以下内容粘贴上去:官方配置说明 AT4G31550.1,AT4G01250.1 red adAT2G30250.1,AT2G04880.1 blue adAT1G80840.1 yellow提交之后的结果:
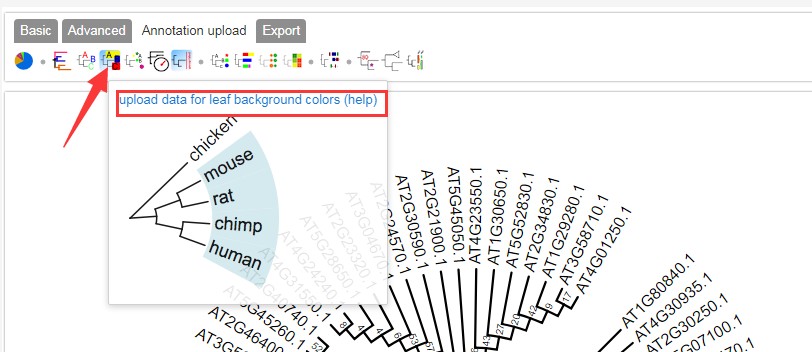
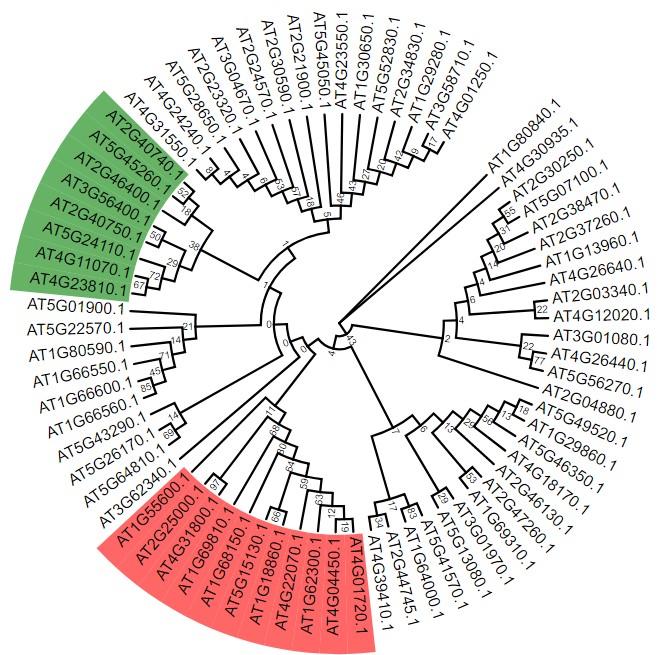
设置背景颜色可看:https://www.omicsclass.com/article/630
可点击上图图红框框处查看官方详细配置说明;配置文件如下:官方说明 AT4G01720.1,AT1G55600.1 red adAT2G40740.1,AT4G23810.1 green ad设置之后结果:
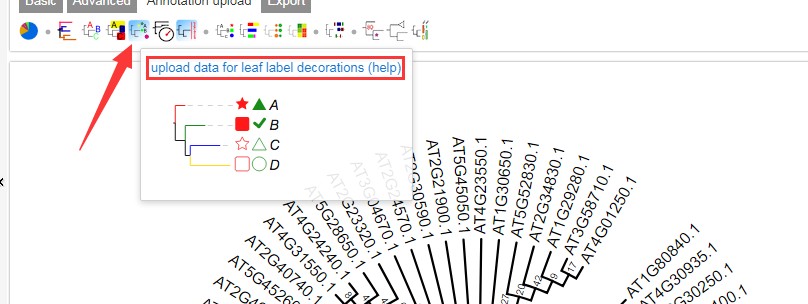
设置星号标签可看:https://www.omicsclass.com/article/664
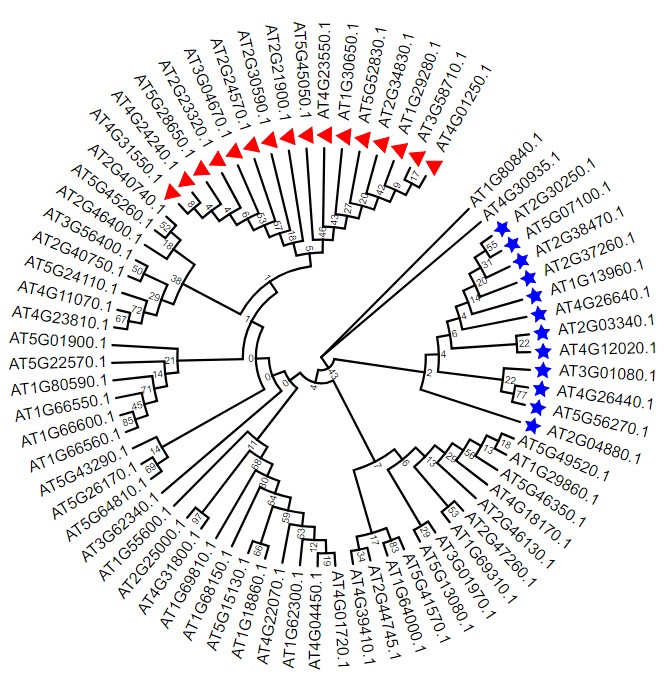
设置参数如下,第一列为基因的ID,后面为设置形状以及颜色,上图图红框框处查看官方详细配置说明。详细说明见官方文档。 AT4G01250.1 triangle,red:redAT3G58710.1 triangle,red:redAT1G29280.1 triangle,red:redAT2G34830.1 triangle,red:redAT1G30650.1 triangle,red:redAT5G52830.1 triangle,red:redAT4G23550.1 triangle,red:redAT2G21900.1 triangle,red:redAT5G45050.1 triangle,red:redAT2G30590.1 triangle,red:redAT2G24570.1 triangle,red:redAT3G04670.1 triangle,red:redAT2G23320.1 triangle,red:redAT5G28650.1 triangle,red:redAT4G24240.1 triangle,red:redAT4G31550.1 triangle,red:redAT2G04880.1 star,blue:blueAT5G56270.1 star,blue:blueAT4G26440.1 star,blue:blueAT3G01080.1 star,blue:blueAT4G12020.1 star,blue:blueAT2G03340.1 star,blue:blueAT4G26640.1 star,blue:blueAT1G13960.1 star,blue:blueAT2G37260.1 star,blue:blueAT2G38470.1 star,blue:blueAT5G07100.1 star,blue:blueAT2G30250.1 star,blue:blue结果如下:
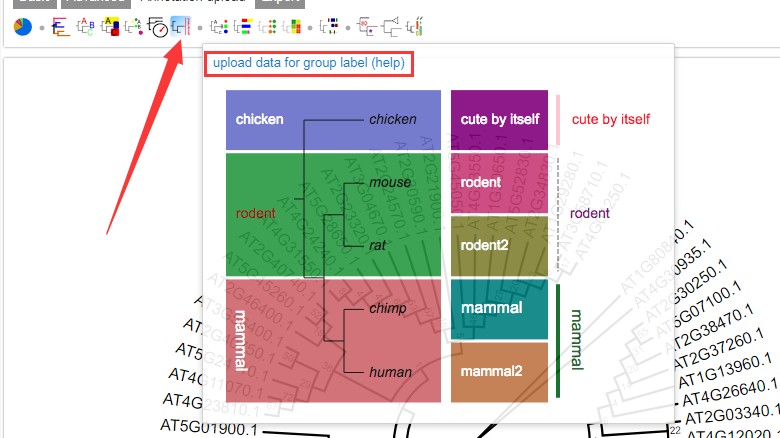
添加分组圆环,设置按钮如下:
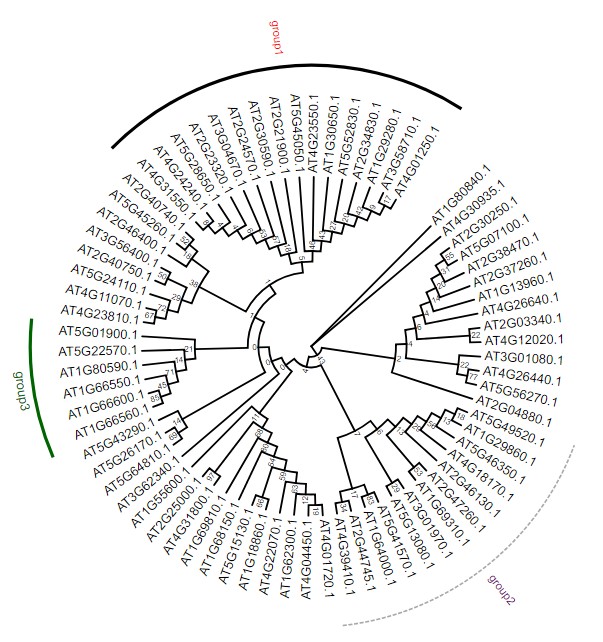
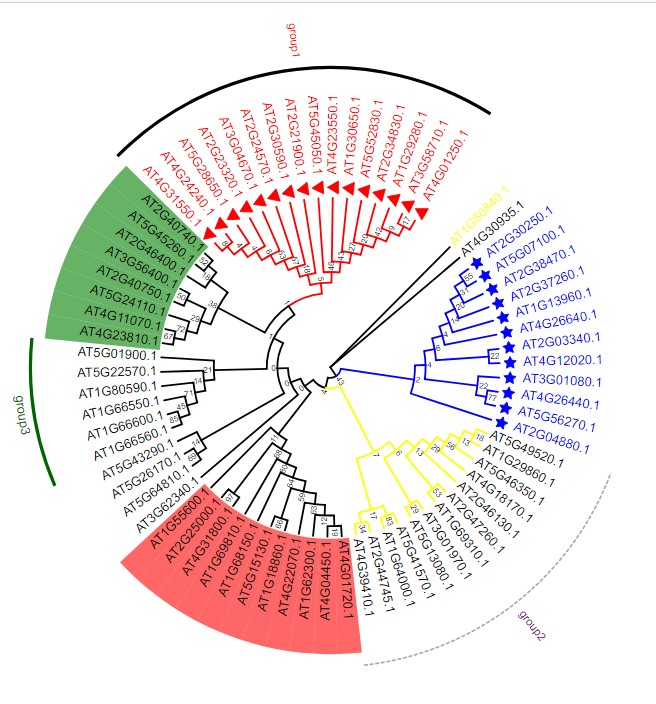
设置文件说明,官方详细说明:DatasetGroupLabel.md: !grouplabelcolor=black,fontsize=14,fontcolor=black,linewidth=2AT4G01250.1,AT4G31550.1 text=group1,fontcolor=red,linewidth=4AT4G39410.1,AT5G49520.1 text=group2,linecolor=darkgrey,fontcolor=purple,linestyle=dashedAT5G01900.1,AT1G66560.1 text=group3,color=darkgreen,textorientation=vertical,linewidth=4,fontsize=16说明如下,带!号的行为全局变量,也就是默认设置参数;其他第一列用,号隔开表示范围,空格之后为各种group分组圆环的显示方式:text为分组名称,fontcolor为字体颜色,linewidth圆环粗细,textorientation控制分组名称对齐样式。
有时候我们想批量重命名基因的名字,可参考iTOL使用:https://www.omicsclass.com/article/604,然后导出nwk格式的文件再到evolview中编辑。 2.7 进化树bootstrap值修改参考可参考:https://www.omicsclass.com/article/625 最后来一个组合图,看看设置的效果,当然我只是教大家如何使用,美观度上还是不足,见谅:
1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程、基因家族文献思路解读 2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读 3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析 4. 转录组数据怎么挖掘?学习链接:转录组标准分析后的数据挖掘、转录组文献解读 5. 微生物16S/ITS/18S分析原理及结果解读、OTU网络图绘制、cytoscape与网络图绘制课程 6. 生物信息入门到精通必修基础课:linux系统使用、biolinux搭建生物信息分析环境、实验室linux生信分析平台搭建、linux命令处理生物大数据、perl入门到精通、perl语言高级、R语言画图、R语言快速入门与提高、python语言入门到精通7. 医学相关数据挖掘课程,不用做实验也能发文章:TCGA-差异基因分析、GEO芯片数据挖掘、 GEO芯片数据不同平台标准化 、GSEA富集分析课程、TCGA临床数据生存分析、TCGA-转录因子分析、TCGA-ceRNA调控网络分析 8.其他,二代测序转录组数据自主分析、NCBI数据上传、二代测序数据解读、 |
【本文地址】
 官方说明文档地址:https://github.com/evolgeniusteam/EvolviewDocumentation
官方说明文档地址:https://github.com/evolgeniusteam/EvolviewDocumentation