| 小白实战课堂开课啦!手把手教你转录因子与靶基因预测操作~! | 您所在的位置:网站首页 › 转录因子结构域怎么找 › 小白实战课堂开课啦!手把手教你转录因子与靶基因预测操作~! |
小白实战课堂开课啦!手把手教你转录因子与靶基因预测操作~!
|
2、转录因子结合位点 基本概念 转录因子结合位点(Tranion factor binding site, TFBS)是转录因子结合在靶基因DNA上的区域。 特征 1)转录因子结合位点序列具有保守性,通常为一段6~12bp的DNA序列,最多不超过20bp; 2)某些转录因子结合位点冗余; 3)转录因子在基因组结合位点不固定,允许错配,理论上转录因子可以结合在DNA任意区域,但是通常只有结合在启动子区域的转录因子才能发挥生物学意义。 表述方式 1)一致性序列:将能与同一个转录因子结合的所有DNA片段按照对应位置进行排列,选择每个位置最可能出现的碱基组成该转录因子结合位点的一致性序列,序列包括A,T,C,G这4中碱基和IUPAC兼并码,后者代表某个位置上可能出现的碱基组合。 2)序列标识图:依次绘出某个DNA motif中各个位置可能出现的碱基字母,碱基字母高度反映该位置的碱基保守性,字母越少高度越高的位置碱基保守性越好,碱基字母的大小与碱基在该位置上出现的频率成正比例关系。 3)位置频率矩阵:矩阵中,行代表转录因子结合位点区域每个碱基位置信息,列代表每个位置四种碱基出现的概率,可以矩阵或表格形式展示。
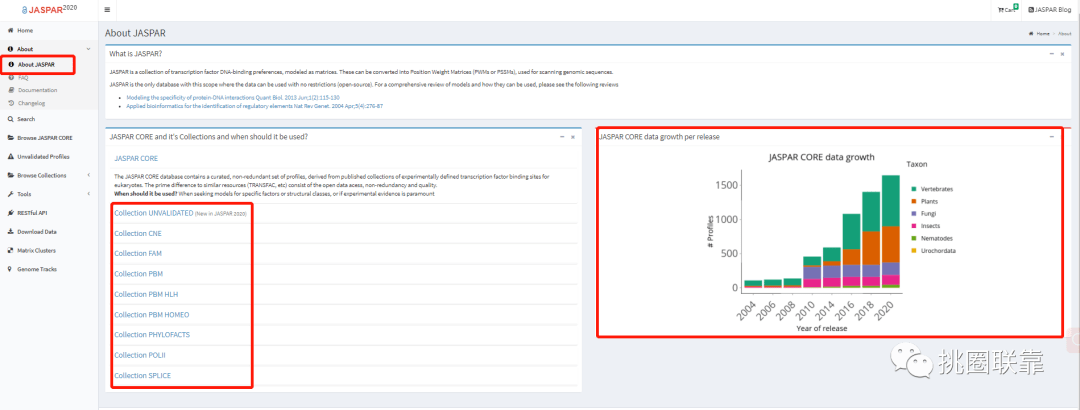
3、在线网站预测转录因子结合位点的缺陷与策略 目前转录因子结合位点的预测主要是基于热力学亲和力,并非真实发生在细胞内的结合,在转录因子结合位点预测时允许碱基错配,因此可能存在大量的假阳性预测结果。另外,转录因子发挥作用时,需要分子伴侣与或组成转录因子复合物发挥转录调控作用,组蛋白表观遗传学修饰引起的染色体结构改变也会影响转录因子的结合,在使用网站预测时没有考虑到这点。综上,我们在做转录因子预测研究时,可以考虑多个数据库的多种算法,将获得的结果取交集,并参考转录因子和靶基因的表达相关性,同时选择一个转录因子复合物内的多个转录因子进行研究,以通量解决概率问题。 JASPAR数据库概览 进入JASPAR主页(http://jaspar.genereg.net/),点击左侧栏About可查看JASPAR基本信息,该数据库为收录转录因子和DNA结合位点信息的开源公共数据库,目前更新到2020年第8版,共有10个子数据集,点击任意一个子数据集,下拉菜单可以看到其简介,右侧显示该子数据集在不同物种中的分布情况,包括脊椎动物、植物、真菌、昆虫和线虫等。
点击左侧栏10个子数据库各自对应的Browse,可进入该模块下各种motif信息的目录界面,分别介绍如下。
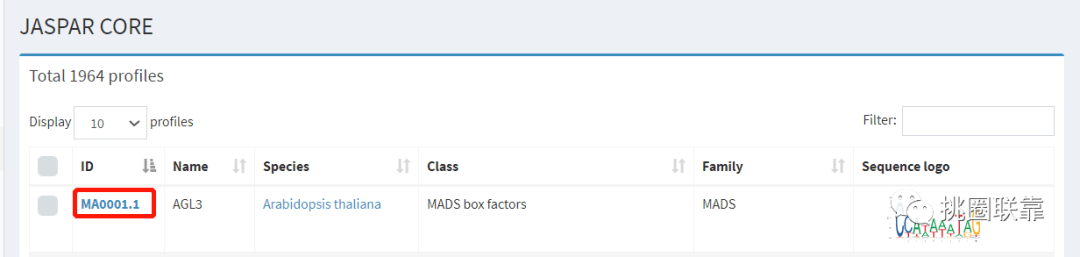
JASPAR CORE子数据库,是JASPAR的核心数据库,也是使用最多的一个数据库,收录来自文献的实验证实的真核生物转录因子结合位点序列信息,是一个经过人工校对过的非冗余数据库,数据质量较高,主要用于特定转录因子或具有特定结构特征的某一类转录因子的结合位点预测。该模块每个motif编号以MA开头。
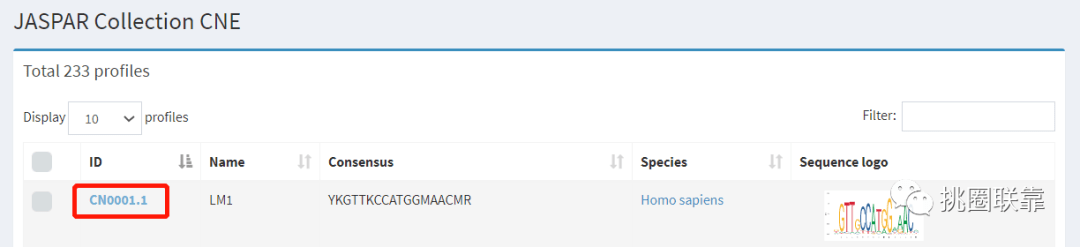
Collection CNE子数据库,包含233个人类基因组中高度保守的非编码基因转录因子结合位点序列信息,大部分可以作为增强子来调控发育和分化相关基因表达,主要用于分析潜在的增强子。该模块每个motif编号以CN开头。
Collection FAM子数据库,是基于JASPAR CORE数据,根据结合位点序列特征将转录因子划分为11个不同的class家族,便于同一位点的多方预测,同时还能发现新的家族模型或将新发现的转录因子进行功能聚类。该模块每个class编号以MF开头。
Collection PBM子数据库,包含基于k-mer微阵列技术获得的104种小鼠转录因子矩阵模式数据。该模块每个motif编号以PB开头。
Collection PBM HLH子数据库,包含基于k-mer微阵列技术获得的19种线虫Helix-turn-helix转录因子模型数据。该模块下的motif编号以PL开头。
Collection PBM HOMEO子数据库,包含基于k-mer微阵列技术获得的176种鼠同源结构转录因子矩阵模式数据。该模块每个motif编号以PH开头。
Collection PHYLOFACTS子数据库,包含174种进化上保守的基因上游结合元件,基于实验验证和尚未定义的转录因子结合位点信息,与JASPAR CORE数据互补,主要用于分析启动子的组织特异性和结构特异性。该部分每个motif的编号以PF开头。
Collection POLII子数据库,包含已知的与RNA聚合酶II核心启动子相关的DNA基序,用于分析潜在的核心启动子。该部分每个motfi编号以PL开头。
Collection SPLICE子数据库,包含6种典型的或非典型的剪切位点序列模型,主要用于剪切位点和可变剪切相关研究。6个motif编号均以SA开头。
Collection UNVALIDATED子数据库,2020年新增板块,包含一些未被验证的现有文献未见报道的转录因子结合位点信息,官方不建议使用。该部分每个motfi编号以UN开头。
点击About目录下Documentation子目录,可以查看每个子数据集包含的具体数据条目,以JASPAR CORE为例,该条目下包含每个Motif的ID编号,转录因子类型和家族信息,种属,文献PMID号等信息。
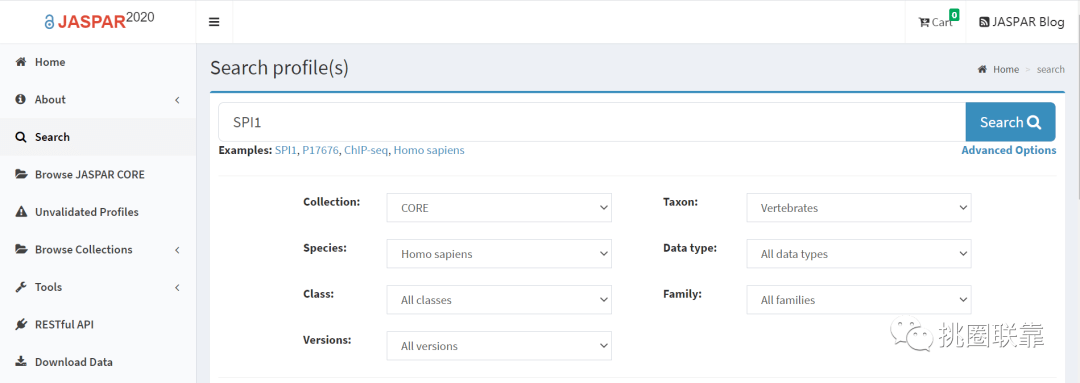
JASPAR功能及操作演示 一、转录因子TFBS信息查询 以JASPAR数据库示例转录因子SPI1为例,进入JASPAR主页,检索框输入SPI1,高级设置选项分别选择感兴趣的子数据库(Collection)、物种(Taxon)、种属(Species)、数据类型(Date type)、转录因子类型(Class)、转录因子家族(Family)和JASPAR版本。
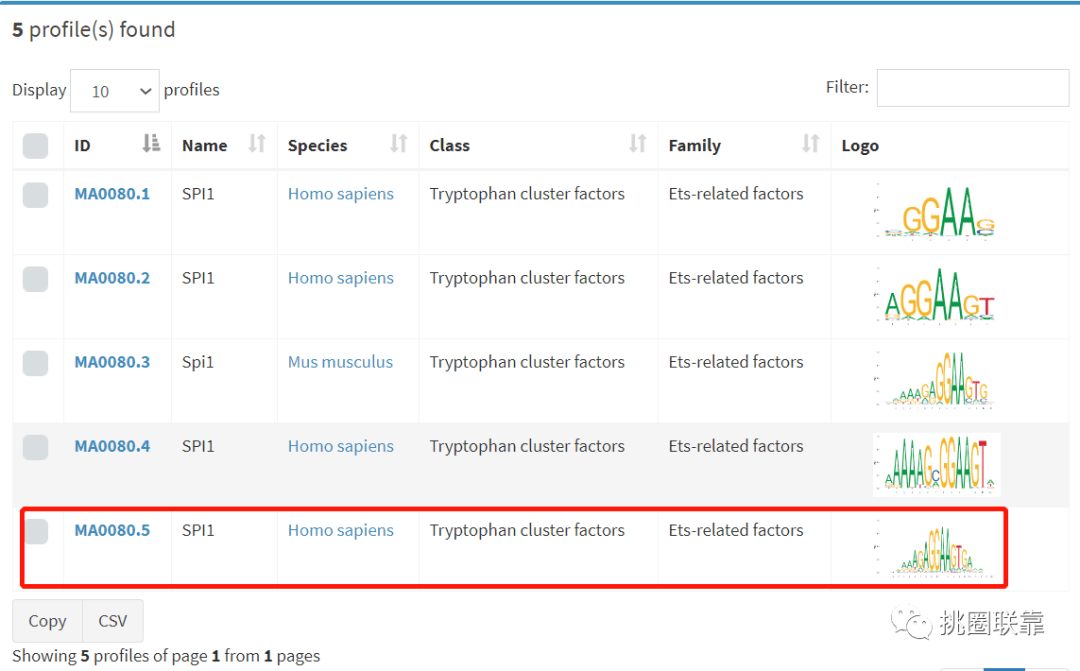
点击Search进入检索结果页面,转录因子SPI1共得到5个版本结果,点击最新版ID进入详情界面。
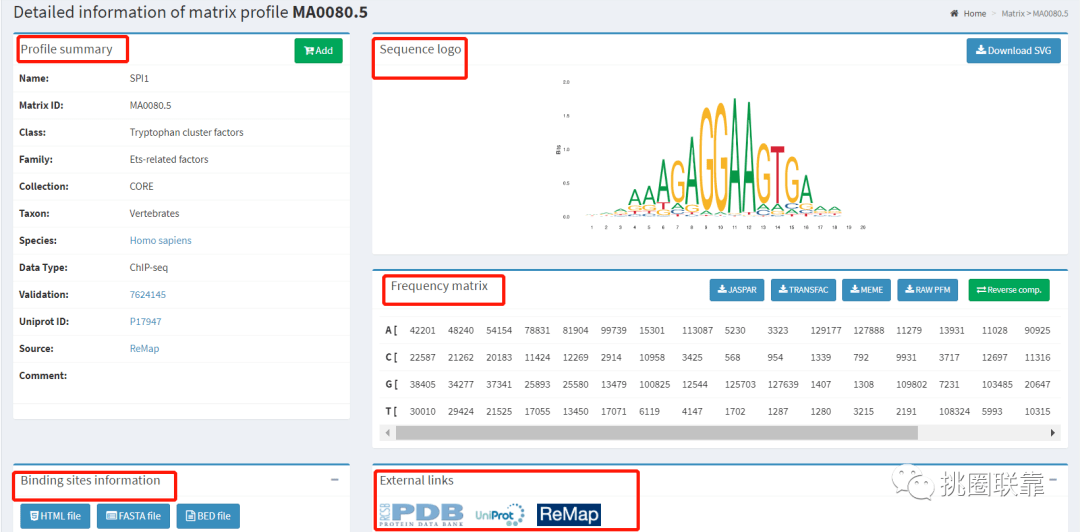
Profile summary提供该转录因子Matrix ID,转录因子名称、类型和家族信息,所属的子数据集,种属,文献PMID号,以及对应的各种数据库ID号,如Uniprot ID、PAZAR ID和TFBSshape ID等。该页面还提供序列标识图和位置频率矩阵,均提供下载功能。另外,可点击外链直接进入PDB、Uniprot和Remap数据库。
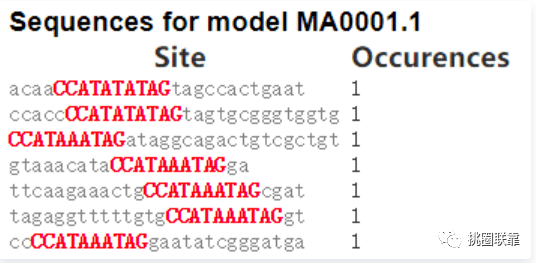
点击Binding sites information下HTML file查看结合位点序列,红色标识即就是motif对应的具体的序列信息。
页面下拉,点击ChIP-seq centrality,展示ChIP-seq数据的Motif富集结果,P值小于0.05,即logP |
【本文地址】