| 老司机教你玩转小胶质细胞形态学分析 | 您所在的位置:网站首页 › 细胞的胞体是什么 › 老司机教你玩转小胶质细胞形态学分析 |
老司机教你玩转小胶质细胞形态学分析
|
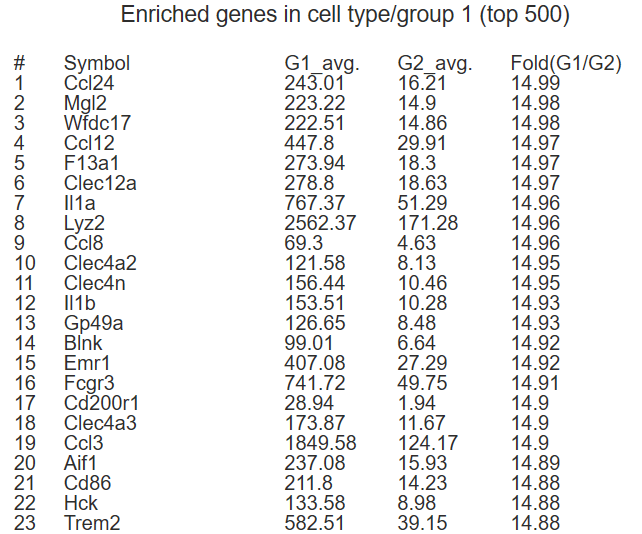
因此对MG形态学分析可评价其活化状态,而MG活化状态又与脑内受损部位的严重程度有着密切的关系。曾经有师妹问我,为什么投稿时我统计了MG的数量来说明MG激活审稿人不认同?这是因为MG的数量变化不足以说明其激活状态。今天半夏就给大家分享一些MG形态学分析的常见指标与分析方法,帮助大家轻松搞定MG定量分析。 1 小胶质细胞标志物 工欲善其事,必先利其器。要做好MG形态学分析首先需要好的标记抗体,那么哪些抗体可以用来标记MG呢?我们使用脑、肺血管单细胞数据库寻找特异性定位于MG的基因,数据库链接为:http://betsholtzlab.org/VascularSingleCells/database.html。 Group1勾选Microglia,Group2勾选除Microglia以外的所有细胞,点击Compare即可查找在MG特异性表达的基因:
富集在MG前23的基因如下:
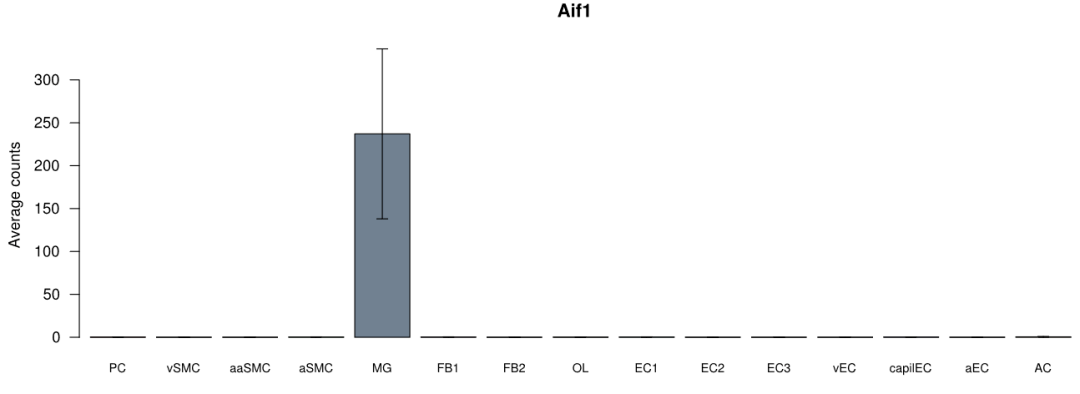
其中就有被广泛用作小胶质细胞标志物的钙结合蛋白Iba1(基因名Aif1),此外近年表达于小胶质细胞表面的明星分子Trem2也在其中。使用脑、肺血管单细胞数据库验证Aif1不同细胞类型的表达如下:
对于抗体购买推荐使用CiteAb(https://www.citeab.com/search)根据引用次数进行查找。 2 小胶质细胞成像技巧 好的图像是形态分析的基础。MG高度分支且不在同一平面,因此很难在单一焦平面将单个MG的完整形态拍清楚:
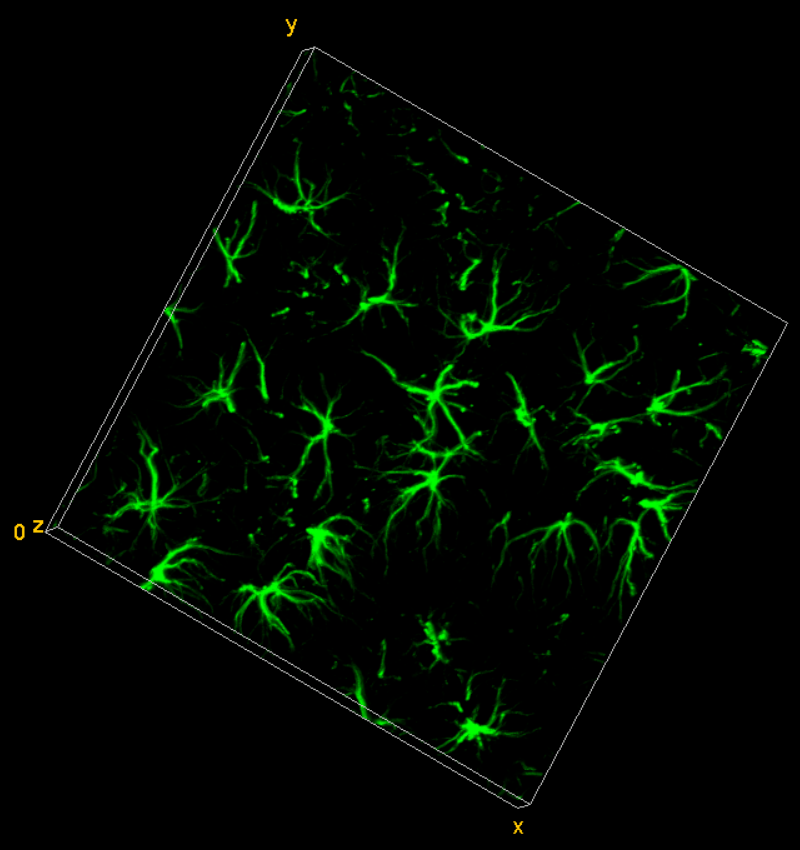
最佳成像方法为使用冰冻切片将脑片切至20-30μm,进行免疫荧光染色后使用共聚焦进行Z stack层扫,得到具有三维立体的MG【3】:
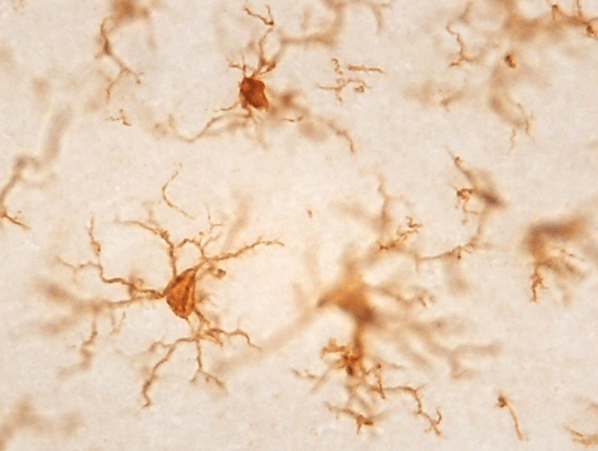
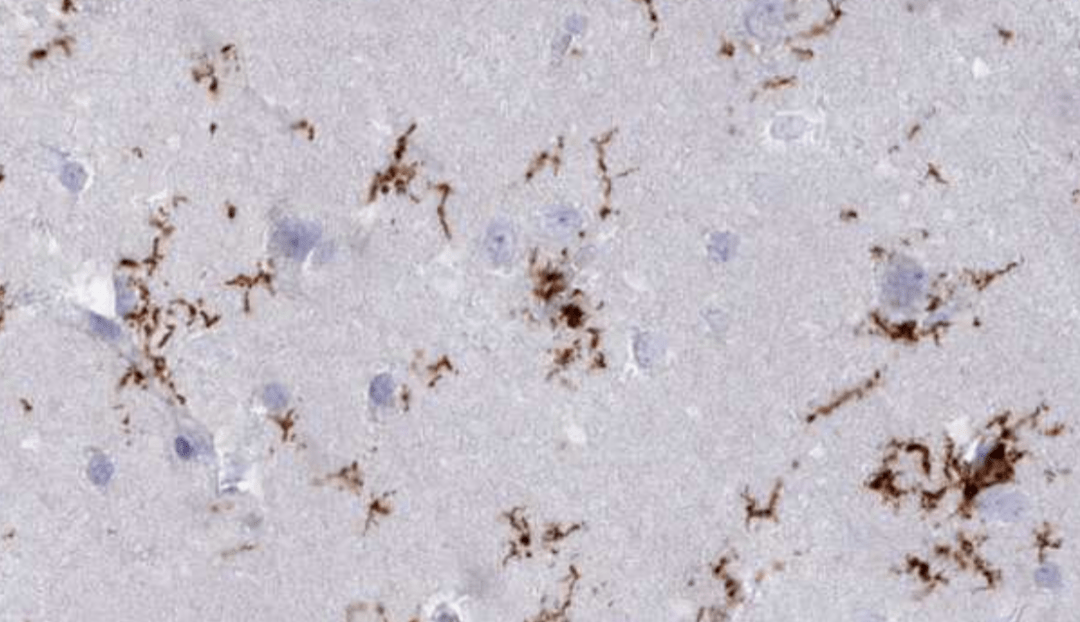
石蜡切片的厚度一般在4~6μm,为更好的保存MG的完整形态不建议使用石蜡切片。人类蛋白组图谱(https://www.proteinatlas.org/)中查询Aif1,石蜡切片的MG形态呈碎片化:
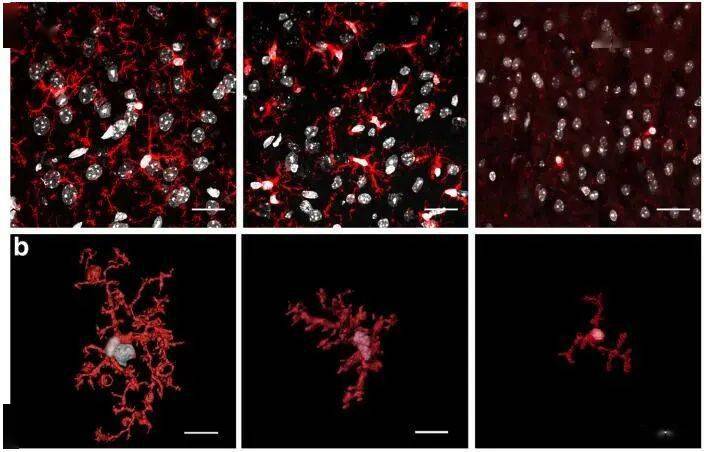
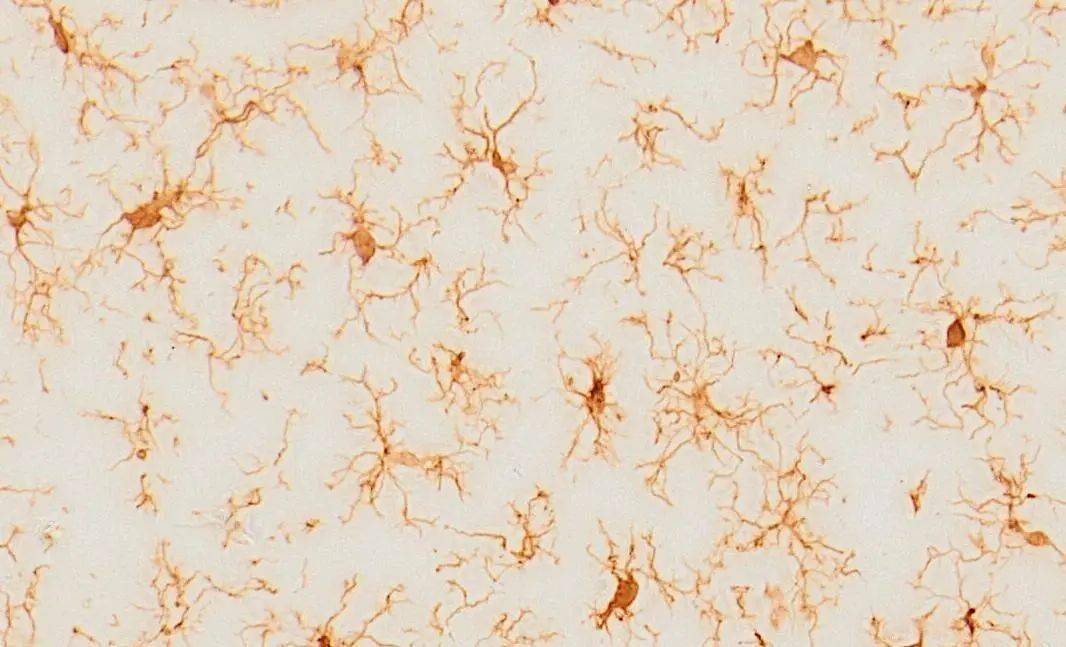
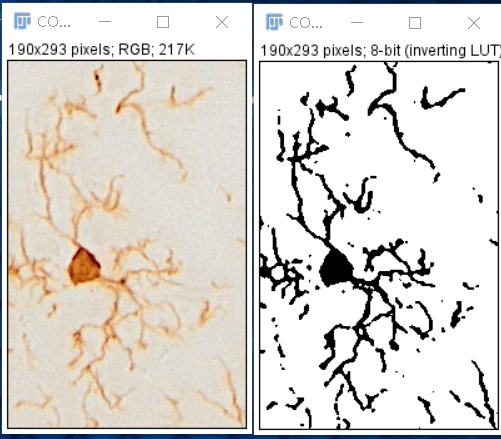
那么,免疫组化是否不适合MG形态学分析呢?也不是,半夏曾经使用免疫组化获得过背景干净、形态完整的MG,效果如下:
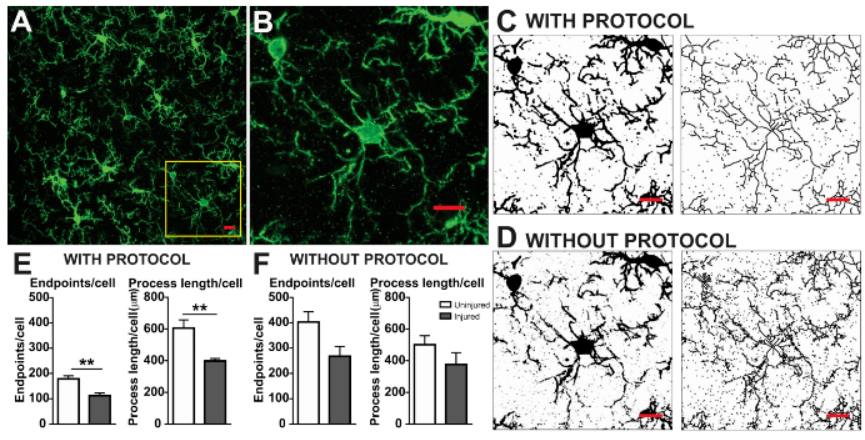
经验如下: 1. 一抗孵育时间为36-48h; 2. 缩短DAB显色时间,即显色时肉眼观察无明显着色; 3. 使用Olympus进行多层扫描后进行扩展景深处理。 附,景深扩展功能为Olympus(sv120)显微镜自带功能,可轻松为超出焦深的样本生成图像。手动景深扩展功能可以连续调整焦距从而在Z轴上组合多幅图像并将其作为单一图像输出。 3 小胶质细胞形态学分析 1. 二维分析指标 1.1 Area(面积),MG density为cell number/100μm2。值得注意的是MG计数标准为计算有胞体的MG数量,碎片化的突起无法计数。 1.2 Branches(分支点数目):MG分支点的数目。 1.3 End-point voxels(分枝末端的数目):MG分枝末端的总数。 1.4 Average Branch Length(突起平均长度):突起总长度除以突起数目。 1.5 Maximum Branch Length(最长突起长度):所有突起中最长的突起的长度。 1.6 Branch Length(分支长度):每个分支的长度。
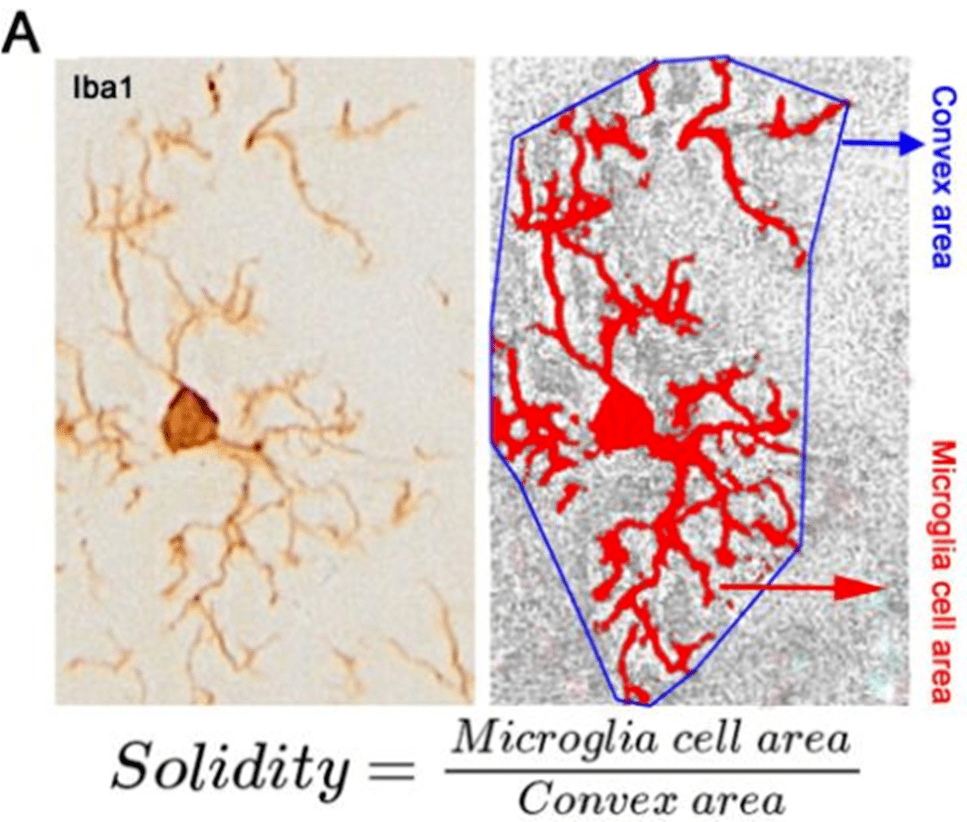
指标1.1-1.6基于MG激活过程中突起回缩,ImageJ分析上述指标请参考往期推文:ImageJ小胶质细胞形态学分析 ,基本步骤为MG图像进行二值化与骨骼化后使用AnalyzeSkeleton(2D / 3D)插件分析骨架【4】。 1.7 Solidity(坚固性):细胞面积/突起面积。
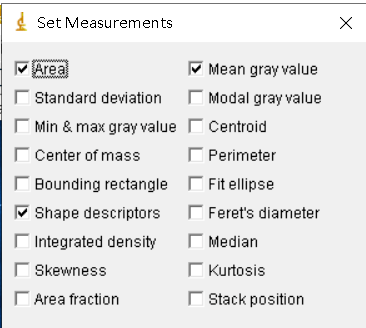
ImageJ -> Set Measurements下勾选Shape deors后结果中有Solidity指标。
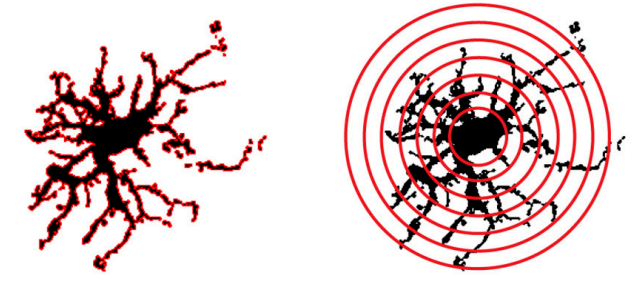
1.8 对MG进行Sholl analysis。Sholl Analysis不仅是分析神经元复杂性的经典方法,也可以用来分析MG的复杂性(例如统计Maximal intersection)。Sholl Analysis以细胞胞体为圆心(不包括胞体)画一系列同心圆,得到随离胞体距离变化的突起交点(Intersections/Crossings)个数,以此来反映细胞的复杂性,MG激活过程中突起复杂性降低。
ImageJ/Fiji自带Sholl Analysis插件,如使用普通ImageJ可于http://fiji.sc/Sholl处下载插件。 分析步骤: (1)ImageJ软件打开MG图片,Image -> type -> 8-bit将图片转换为8-bit灰度图片。
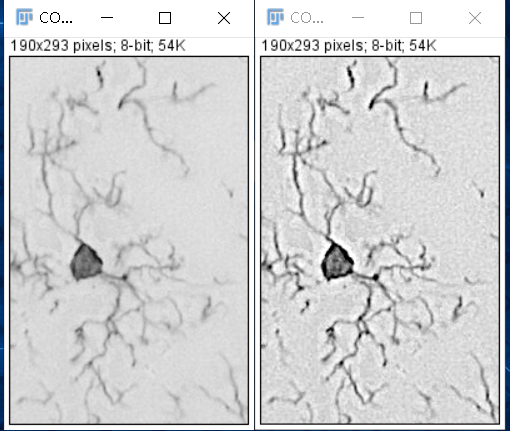
(2)Process -> Filters -> Unsharp Mask增加对比度(右图):
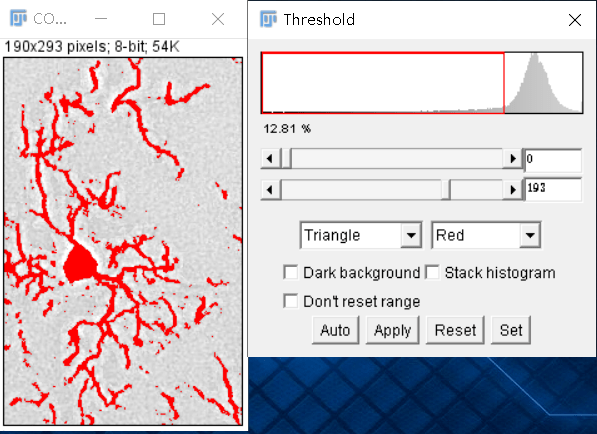
(3)Image -> Adjust -> Threshold选中MG,Apply进行二值化:
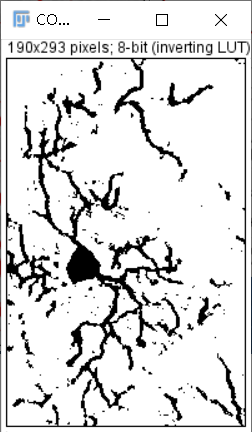
Process -> Noise -> Despeckle去除噪点:
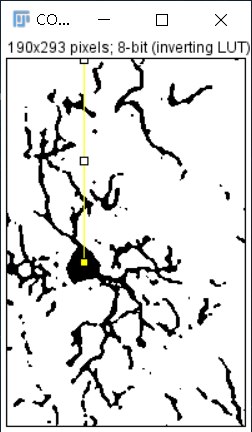
(4)使用直线工具以MG胞体中心为起点向最长突起处画直线:
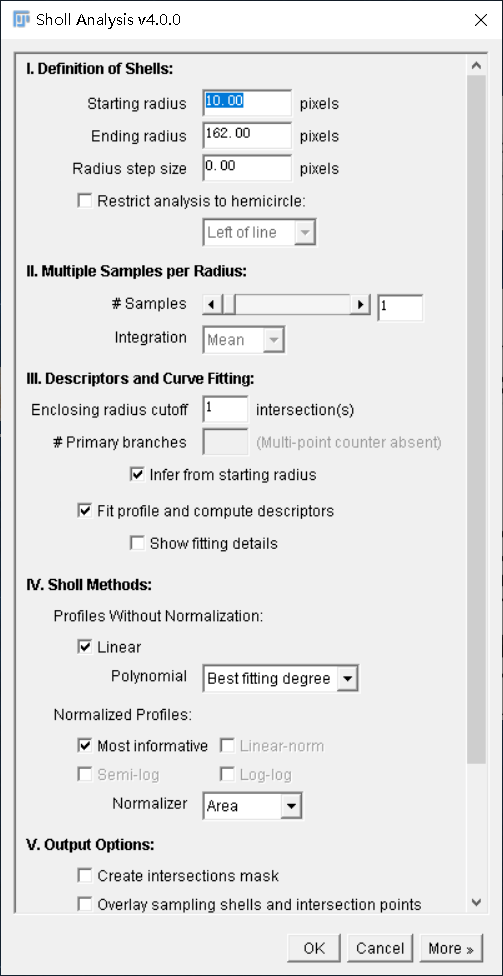
Analyze -> Sholl -> Deprecated -> Sholl Analysis,进入Sholl Analysis参数界面:
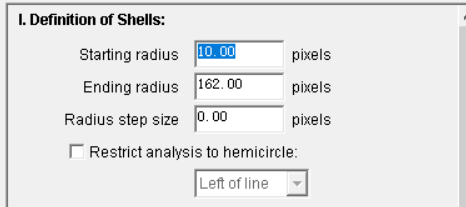
Definition of Shells设置开始Sholl分析的半径(即胞体的大小),Ending radius为最长突起的位置即Sholl分析结束的半径,Radius step size为两个分析半径之间的步进:
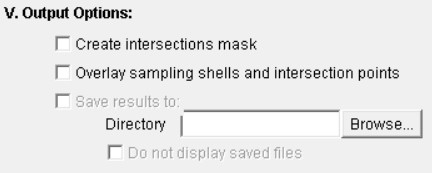
Output Options可以设置导出结果,包括Intersections mask与显示在分析图像上显示交点与同心圆:
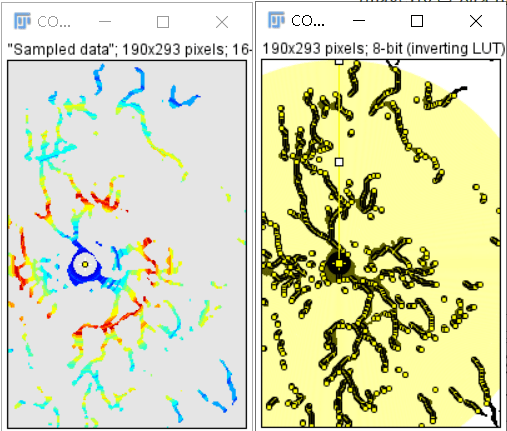
得到如下结果:
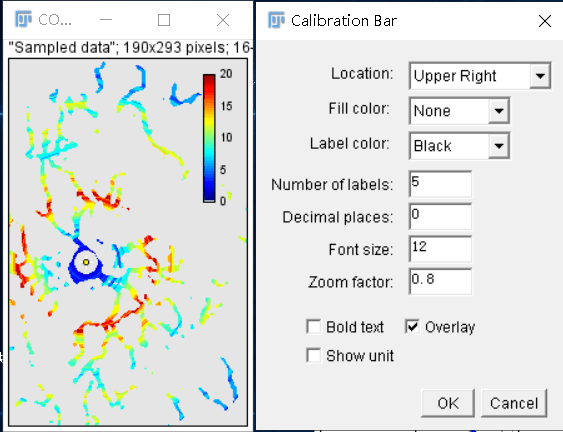
左侧的Intersections mask为伪彩图片,颜色深浅代表交点数。点击Analyze -> Tools -> Calibration Bar可以添加Color bar,设置bar位置(Location)、颜色、字体大小等,点击OK可见最多交点为20个:
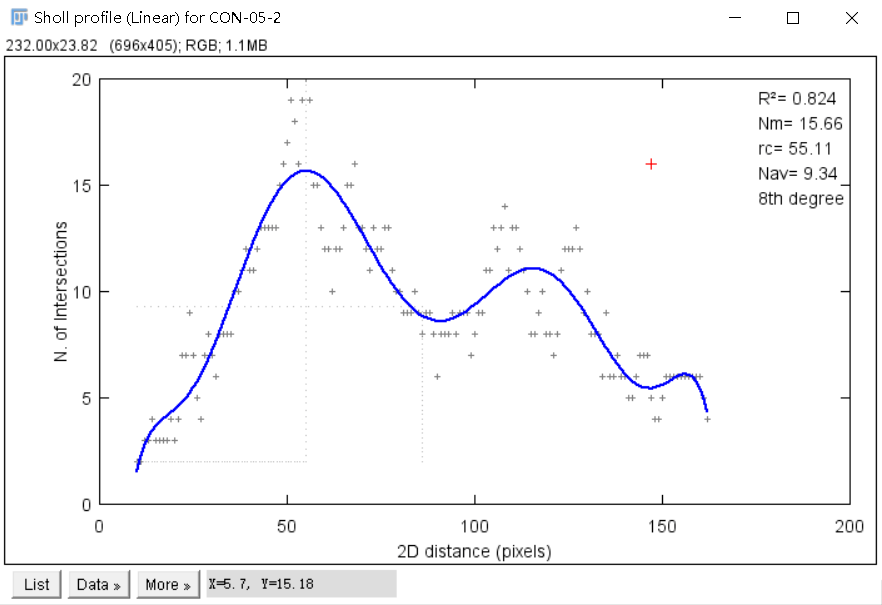
Sholl Analysis还可以得到SCI论文中常见的交点统计图,点击List可查看并保存数据:
上图每个小十字架为对应分析半径的实际交点数目,蓝色曲线为对2D distance与交点数目进行多项式拟合的曲线。R2为拟合曲线与散点的相关性为0.824,右上角其他数据解释如下:
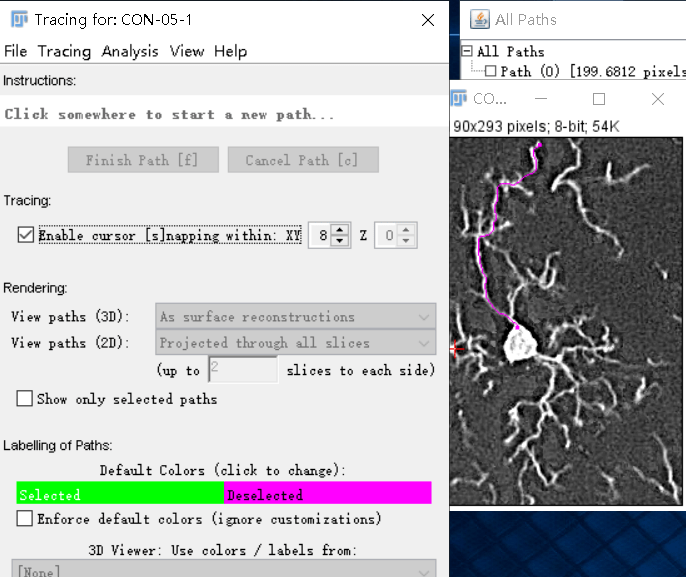
指标 含义 Max 实际交点数最大的位置。 Nm 临界值,即拟合曲线最高点对应的交点数,对应的Sholl半径为临界半径rc。 Nav 指所有交点的平均值。曲线中心的x为分析形状的质心所在位置。 RImax 实际交点数最大值与Starting radius交点数目/分枝数的比值。 RINm 交点临界值交点数与Starting radius交点数目/分枝数的比值。 扩展: 除对图片进行二值化后进行Sholl分析外,还可对追踪重建的神经元进行Sholl Analysis,此方法同时可以得到突起的长度。使用功能为Fiji下Plugins -> Segmention -> Simple Neurite Tracer。
此外,NeuronJ插件也可以用来追踪突起长度。NeuronJ下载链接为:https://imagescience.org/meijering/software/neuronj/。将下载文件放至Plugins文件夹中,重启软件后在ImageJ软件Plugins菜单中即可找到NeuronJ插件。
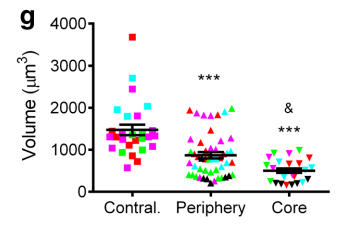
2. 三维分析指标 2.1 Volume(体积),对应二维指标为MG的面积(Area),吞噬状态的MG体积减小。
ImageJ打开层扫Stack图片,Plugins-> 3D Viewer -> Display as Volume,即可查看的三维效果图:
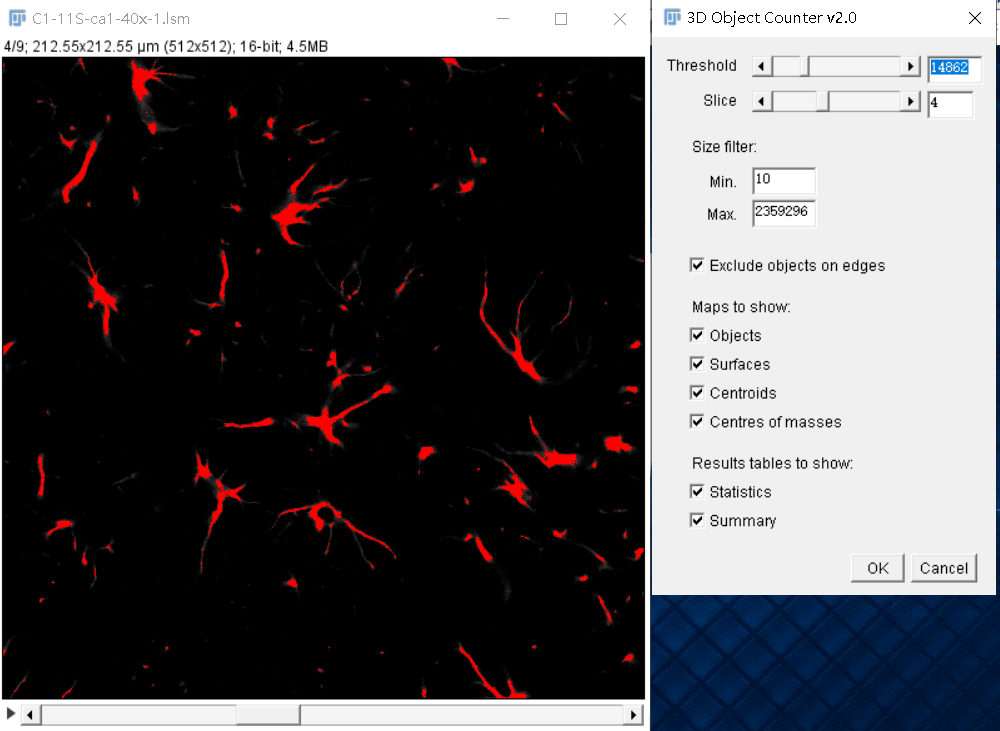
后续可以使用Analyze -> 3D Objects Counter计算体积。Threshold调节阈值选中不同Slice中的目标,红色代表选中。Slice代表当前切片,Size filter根据最大值与最小值筛选目标:
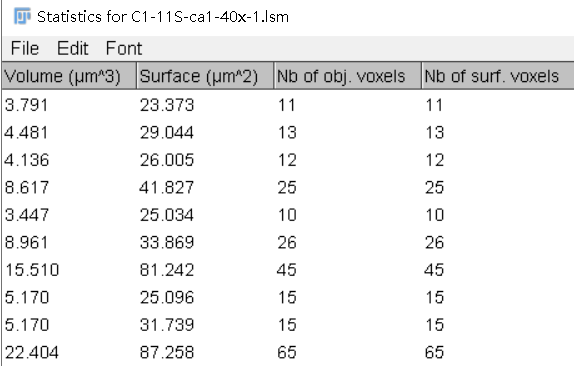
点击OK得到体积、表面积等指标:
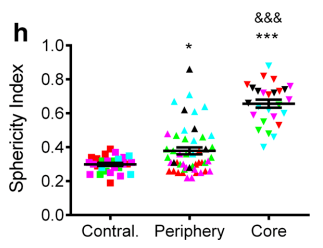
2.2 Sphericity index(球度),在3D图像创建后用Imaris软件可以实现计算。
形态学指标能够很好的反映MG功能状态的改变,指标众多,挑选最为自己所用的指标进行分析统计,有助于加强文章的说服力,希望对大家有所帮助,祝大家早日发文章! 参考文献: 【1】 Lawson LJ, Perry VH, Dri P, Gordon S.Heterogeneity in the distribution and morphology of microglia in the normaladult mouse brain. Neuroscience. 1990;39(1):151-70. doi:10.1016/0306-4522(90)90229-w. PMID: 2089275. 【2】 Nayak D, Roth TL, McGavern DB. Microglia development and function. Annu Rev Immunol. 2014;32:367-402. doi: 10.1146/annurev-immunol-032713-120240. Epub 2014 Jan 22. PMID: 24471431; PMCID: PMC5001846. 【3】 Otxoa-de-Amezaga A, Miró-Mur F, Pedragosa J, Gallizioli M, Justicia C, Gaja-Capdevila N, Ruíz-Jaen F, Salas-Perdomo A, Bosch A, Calvo M, Márquez-Kisinousky L, Denes A, Gunzer M, Planas AM. Microglial cell loss after ischemic stroke favors brain neutrophil accumulation. Acta Neuropathol. 2019 Feb;137(2):321-341. doi: 10.1007/s00401-018-1954-4. Epub 2018 Dec 22. PMID: 30580383; PMCID: PMC6513908. 【4】 Young K, Morrison H. Quantifying Microglia Morphology from Photomicrographs of Immunohistochemistry Prepared Tissue Using ImageJ. J Vis Exp. 2018 Jun 5;(136):57648. doi: 10.3791/57648. PMID: 29939190; PMCID: PMC6103256. — END— 点下“在看”,多根头发返回搜狐,查看更多 |
【本文地址】