| 除了相关系数,物种网络构建还有哪些方法?(一) | 您所在的位置:网站首页 › 生物丰度指数计算教程 › 除了相关系数,物种网络构建还有哪些方法?(一) |
除了相关系数,物种网络构建还有哪些方法?(一)
|
(2)工具:R语言psych包、Omicshare tools”组内相关性分析”工具(图1)。
图1 OS小工具 2. MENA法 (1) 特色:全称Molecular Ecological Network Analysis。由于传统相关系数计算完成后,一般只能根据主观经验结合相关系数和P值进行数据筛选。 而MENA的重要特色在于: 阈值:计算完相关系数(提供pearson等选择)后,通过一系列阈值调整,获得随机矩阵(Random Matrix Theroy)进行评估,最终选择最佳筛选阈值,构建网络图; 模块:基于构建好的网络图开展网络结构分析,如将物种划分成不同的模块,并统计物种在模块内、模块间的连通性特征等(图2)。
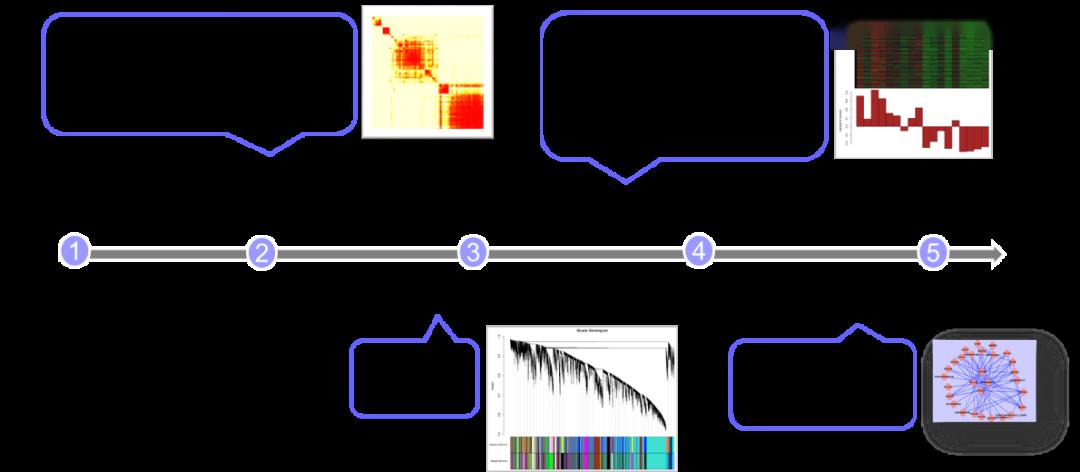
图2 MENA分析流程 (2) 工具:公开免费线上处理(http://ieg4.rccc.ou.edu/mena/) 3. WGCNA法 (1) 特色:全称Weighted gene correlation network analysis,最早是用于基因间表达调控网络的分析,近来逐渐应用于物种关系分析。基于物种的相对丰度表进行计算。最大的亮点在于对pearson等相关系数的变形转化。 分析流程如图3,获得相关系数矩阵后,取次方操作,以扩大强相关弱相关的间隔,然后融入直接相关和间接相关,得出新的相关性描述,即weight值(也称TOM值),一般取值[0,1],所以可以理解为WGCNA是升级版的pearson/spearman等相关系数分析。最后基于TOM值将物种划分为不同的模块。一个模块为一簇强相关的物种。
图3 WGCNA分析流程 PS:具体原理可学习基迪奥云平台Omicshare class中的WGCNA课程。 课程链接:https://www.omicshare.com/class/home/index/series?id=3
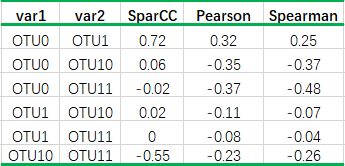
(2)工具:R语言WGCNA包。 4. SparCC法 (1) 特色:基于物种的绝对丰度表(tag counts)进行计算。由于传统方法基于相对丰度计算时,经常出现负相关偏多,即正相关被抑制的现象,所以SparCC方法进行了优化。 首先将丰度进行取对数转化,以有效消除 “正相关性被限制”的问题;然后基于pearson线性相关原理,结合方差计算相关系数,取值[-1,1]。最后基于置换检验获得相关性P值。 文献[1]中相同数据SparCC、pearson、spearman计算结果截取如表2,可以看出SparCC的计算结果与另两种差异比较大,强相关和弱相关更突出,且无“正相关被限制”的现象,说明对数化处理和方差引入对数据相关性评估有非常积极的影响。 表2 相关系数结果对比
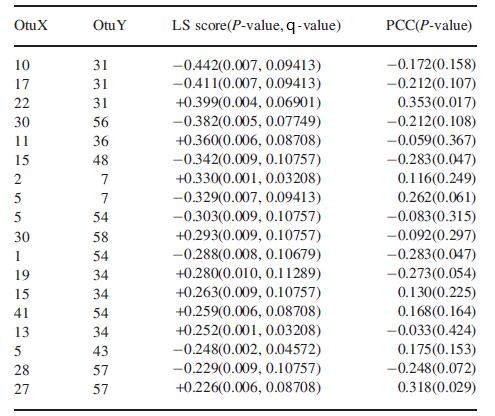
(2) 工具:Python(https://bitbucket.org/yonatanf/sparcc) 5. LSA法 (1) 特色:全称local similarity analysis。仅针对时间梯度样本中的微生物网络开展分析,相对于普通pearson相关性(表3),LSA方法对物种间相关性的捕捉更敏感准确 [2] ,可广泛用于生长、季节、处理周期等时间尺度的物种网络分析。 表3 LSA与pearson系数(PCC)结果对比
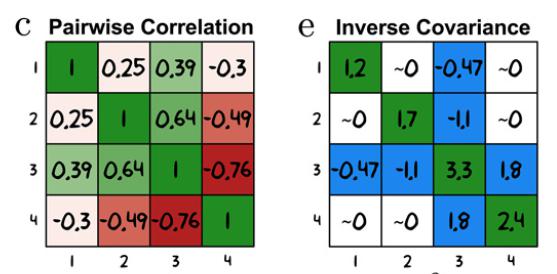
基于物种绝对丰度表格,首先进行F-函数转化,以区分生物学重复和时间梯度的数据特征。然后结合时间梯度、pearson相关等计算出物种间的LSA得分,取值[-1,1],再进行置换检验计算P值,最终输出显著相关的用于绘制网络图的数据。 (2) 工具:python(如https://bitbucket.org/charade/elsa/src/master/) 6.SPIEC-EASI法 (1) 特色:全称SParse InversE Covariance Estimation for Ecological AssociationInference,基于物种绝对丰度表分析。 1)首先将丰度数据进行中心化-取对数转化,处理优势同SparCC; 2)提供neighborhood和inverse covariance(协方差的逆矩阵,也称为精度矩阵[precision matrix或部分相关矩阵[partial correlationmatrix] )两种模型构建网络,并结合Stability-Based module进行评估; 3)输出协方差逆矩阵作为关系数据,取值[-∞,+∞]。如图4,由此可见,该算法是与pearson完全不同的一种新颖关系构建方法。这种处理模式的好处在于网络的简约性,避免了传统方法中间接相关对网络结构的影响。
图4 pearson相关系数与SPIEC-EASI结果对比 [3] 7.CoNet法 (1) 特色:全称Co-occurence network,基于物种绝对丰度表分析。最大的特色在于提供了多种网络关系构建方法,并将每种结果进行筛选合并。作为cytoscape软件的插件,合并的结果还可直接在软件中可视化展示(图5)。
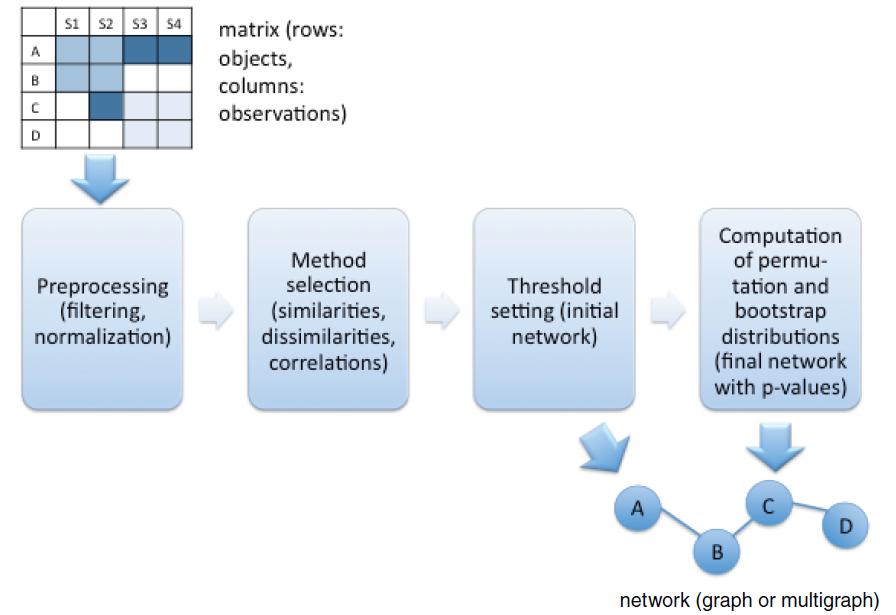
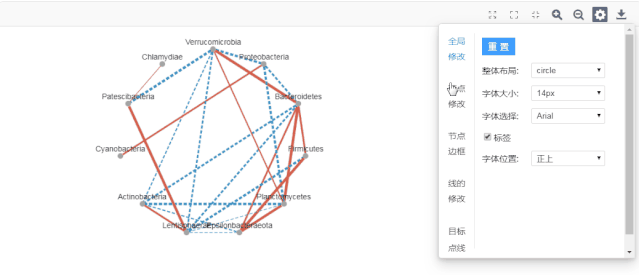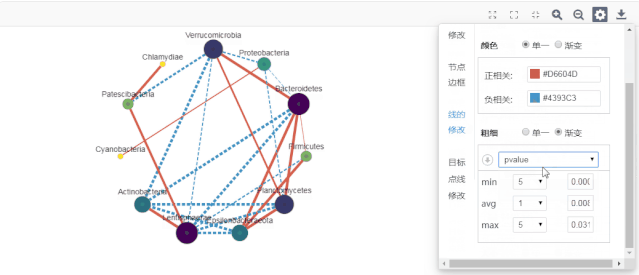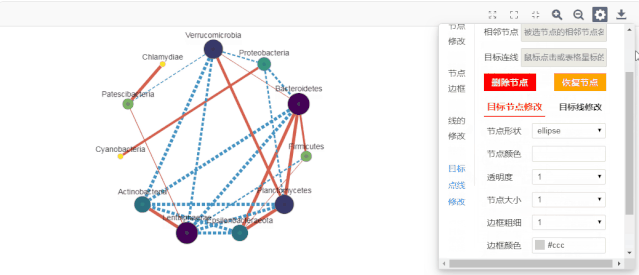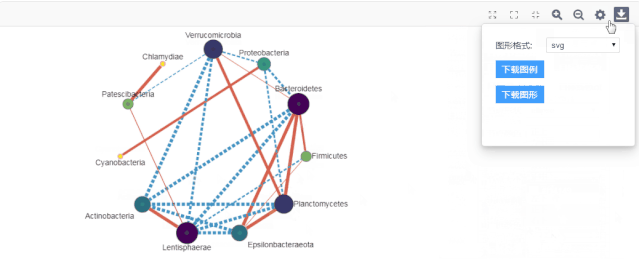
图5 CoNet分析流程 (2)工具:Cytoscape软件CoNet插件 物种关系可视化 大家根据模型特点,选择某种方法即可得到网络关系数据。但是从数据到文章中高大上的网络图,还需要使用Cytoscape等软件进行可视化展示。 具体使用方法和技巧,可学习基迪奥云平台Omincshare class中的Cytoscape课程。 课程链接:https://www.omicshare.com/class/home/index/series?id=3 Omicsmart物种网络图 如果觉得计算又复杂, Cytoscape太难,可以来集测序数据上传、数据挖掘、图形美化于一身的Omicsmart微生物平台。 最近更新的Omicsmart metagenome version 4.0版本中的物种组成板块贴心地提供了各分类水平物种间网络图分析,我们基于pearson计算结果,配备了高大上的网络图绘制功能,如图6,既可进行网络图的精简筛选,还可进行丰富的颜色、大小的渐变等美化,足以满足一幅SCI级图形的各种需要,而且可以一键生成图例哦。
图6 Omicsamrt物种相关性网络图分析 平台地址:https://www.omicsmart.com/ 对Omicsmart感兴趣的小伙伴赶紧扫码报名哦,填写您的测序、分析意向,我们将从填写信息中抽取20位,赠送价值500元的Omicsmart VIP会员30天使用权限。Omicsmart的VIP会员拥有200G的超大存储空间,单个样本流程分析价格低至70元(市场价约350元)! 物种关系研究为何有如此魅力,引来一批又一批科学家进行各种方法探索呢?关于物种关系分析的具体意义和应用价值,我们将在后续文章中分享,敬请关注。 参考文献: [2]Ruan Q, Dutta D, Schwalbach MS, SteeleJA, Fuhrman JA, Sun F. Local similarity analysis reveals unique associationsamong marine bacterioplankton species and environmental factors.Bioinformatics. 2006;22(20):2532–2538. doi:10.1093/bioinformatics/btl417返回搜狐,查看更多 |
【本文地址】