| 干货∣如何得到染色体级别的植物基因组序列? | 您所在的位置:网站首页 › 微分基因怎么样 › 干货∣如何得到染色体级别的植物基因组序列? |
干货∣如何得到染色体级别的植物基因组序列?
|
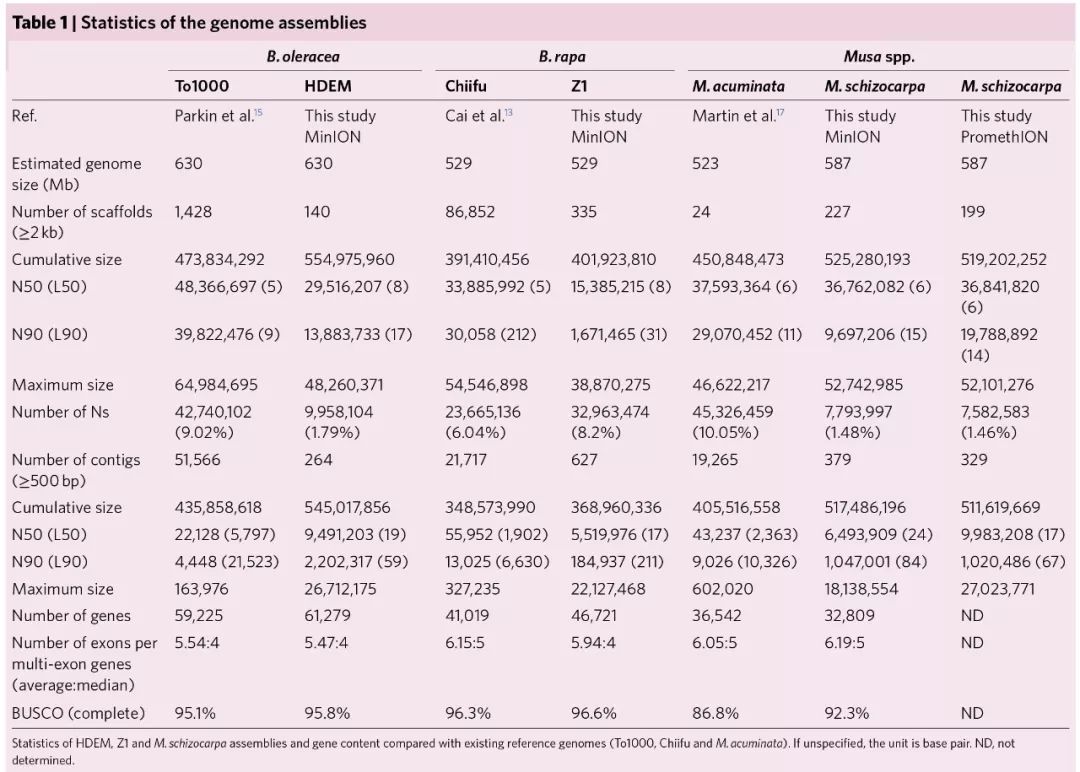
本次组装contig N50与之前相比,提升了100倍和450倍;从注释结果来看,B. rapa,B. oleracea和M. schizocarpa分别注释出46,721,61,279,32,809个基因,与之前发表的结果相似。下表是本次组装结果与以往公布结果的详细比较。
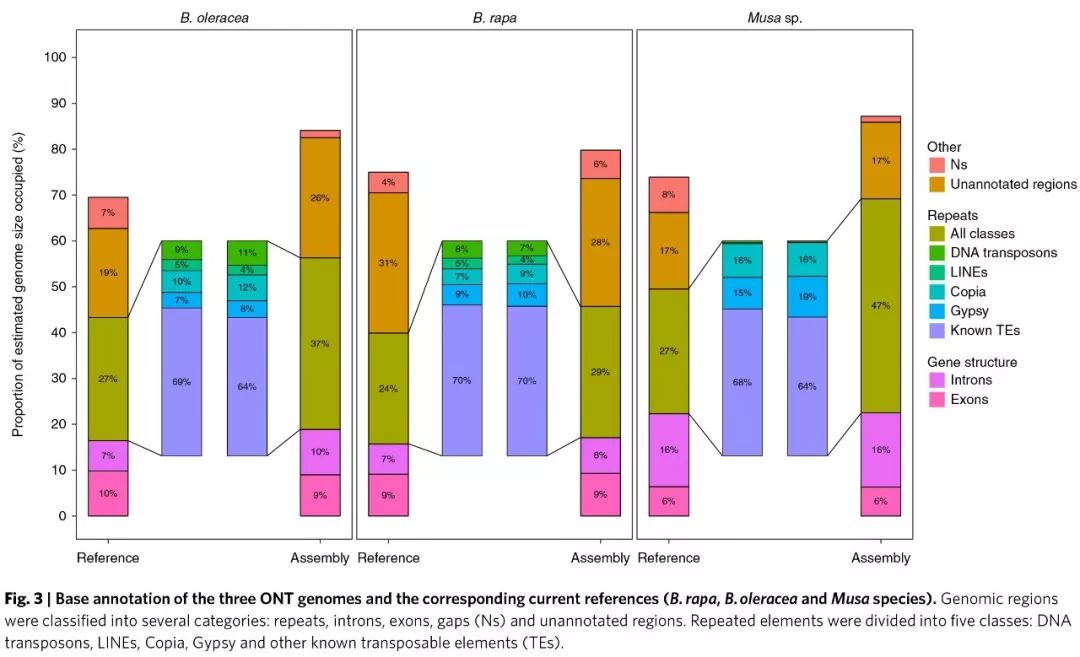
值得一提的是,虽然注释的功能编码基因个数与之前发表的结果较一致,但是采用长片段测序明显提升了重复序列的组装结果,检测到的重复序列比例较高,且转座元件的平均长度要长;而且通常在转座子富集区域的基因难以定位到染色体上,但是本研究组装中能将超过98%的基因锚定到染色体上。由此可见,长reads对于提升转座元件富集区域的组装质量是非常关键的。
同时分析了FLC 基因的拷贝数(该基因与春化及开花时间有关,该基因家族的拷贝数变化能够影响开花时间),在B.oleracea(broccoli, HDEM) 和B.rapa Z1 中分别发现了7 个和4 个FLC 基因。表明长读长更有利于重复区域的组装。 抗病基因R-genes 一般是成簇出现的,较难正确地组装;the M. acuminata 和the M. schizocarpa 基因组中3 个同源R-gene 簇中不确定碱基的比例分别为6.5% 和0% ,再次显示了长读长对复杂区域组装的重要性。 进一步对199 份B.rapa 和119 份B.oleracea 材料进行了重测序,将测序结果与本次组装的序列及之前的序列进行比对,发现除了Chinese cabbage ,Chinese kale 外,其它的与本次组装的参考序列比对率更高;但是uniquely mapped reads 的比例要低,进一步说明新组装的序列重复区域组装的效果更好,新组装的序列更适合在后续芸薹属重测序中作为参考序列。
PacBio测序、Nanopore测序都能获得长的reads,这两种测序技术对组装有何影响呢?文章比较了用PacBio测序的6个物种与本文的3个物种之间的组装结果(基因组大小在130-630Mb之间),发现使用ONT获得的大于50kb的reads比例更高;而PacBio的测序深度更高一些(在125X-283X之间),表明PacBio需要更高的测序深度以获得足够的长reads来提升组装的连续性。在这9个物种中,连续性第二好的,长读长reads的深度只有36X,但是reads长度是最长的,说明相比于测序深度,更长的reads对提升组装结果更有效;30X的long reads能够满足组装的需求。 不过,小编认为这里的比较如果在同一物种上进行平行比较会更有说服力,不过不管怎样,从文章的比较结果来看,reads长度是决定组装结果好坏的关键因素。
PacBio三代测序仪 随着Nanopore、PacBio测序技术的发展、升级及各种新组装软件的出现,必定会在极大降低研究费用的同时大大提升基因组组装效果,那些以往难以取得较好结果的复杂基因组,将会迎来研究契机,当然,那些相对简单的基因组组装结果也会更好。 微分基因通过搭建世界领先的高通量测序平台、基因芯片平台、大数据分析平台,在全国范围内与众多学术机构、研发企业以及健康管理平台建立了深入的合作关系,为一系列基因组科学研究、健康管理项目等提供专业基因测序技术服务。 如果您有物种想进行基因组测序,现在可以准备起来啦。 参考文献: 1.Arabidopsis Genome Initiative Analysis of the genome sequence of the flowering plant Arabidopsis thaliana. Nature 408, 796–815 (2000). 2.International Rice Genome Sequencing Project The map-based sequence of the rice genome. Nature 436, 793–800 (2005). 3.Du, H. et al. Sequencing and de novo assembly of a near complete indica rice genome. Nat. Commun. 8, 15324 (2017). 4.Edger, P. P. et al. Single-molecule sequencing and optical mapping yields an improved genome of woodland strawberry (Fragaria vesca) with chromosome-scale contiguity. Gigascience 7, 1–7 (2018). 5.Dassanayake, M. et al. The genome of the extremophile crucifer The llungiella parvula. Nat. Genet. 43, 913–918 (2011). 6.International Brachypodium Initiative Genome sequencing and analysis of the model grass Brachypodium distachyon. Nature 463,763-768(2010) 7.Raymond, O. et al. The Rosa genome provides new insights into the domestication of modern roses. Nat. Genet. 50, 772–777 (2018). 8.Caroline B. et al. Chromosome-scale assemblies of plant genomes using nanopore long reads and optical maps. Nature Plant. 4(11):879–887 (2018).返回搜狐,查看更多 |
【本文地址】