| 说一说转录组链特异性文库的那些事 | 您所在的位置:网站首页 › 建文库的目的 › 说一说转录组链特异性文库的那些事 |
说一说转录组链特异性文库的那些事
|
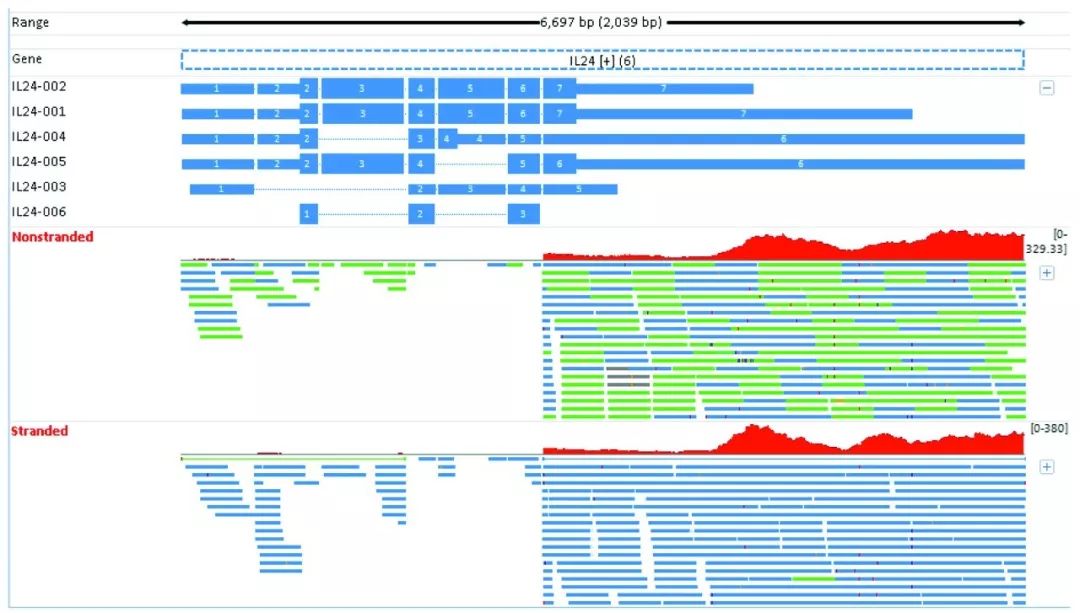
图1 IL24基因链特异性文库与非链特异性文库定量结果 研究已证实IL24位于“+”链上,因此真正源自IL24基因的序列reads是反向互补并映射到“-”链上。因此,在链特异性文库的RNA-seq中,仅计数映射到“-”链的reads。可以看出,在链特异性文库几乎所有的reads都被映射到“+”链(如图2)。因此,不计算这些reads数,也说明了为什么在图1链特异性文库的IL24基因没有表达。链特异性文库比非链特异性文库基因的表达水平更可靠。
图2 IL24基因定量结果可视化图 (蓝色是比对到正链,绿色是负链) 02、有利于发掘潜在顺式天然反义转录本 天然反义转录物(Natural antisense trans,NATs)通常指天然情况下生物体内生成的反RNAs,它们可以与其互补的RNAs通过碱基配对,形成正义-反义RNAs双链(dsRNAs)。根据来源,NATs可以为两类:顺式NATs(cis-NATs)和反式NATs(trans-NATs)。Cis-NATs来自基因组同一位点相反链上的转录,互补区在两个基因的重叠部分,重叠长且完全互补,通常以“一对一”的方式调节有义链的表达。Trans-NATs来自基因组不同位点的转录,重叠区域短且不完全重叠,可以调节多个有义链的表达。 链特异性文库可以提高反义链上非编码转录本的检出率,同时也便于确定哪些位于基因间的非编码转录本的方向。
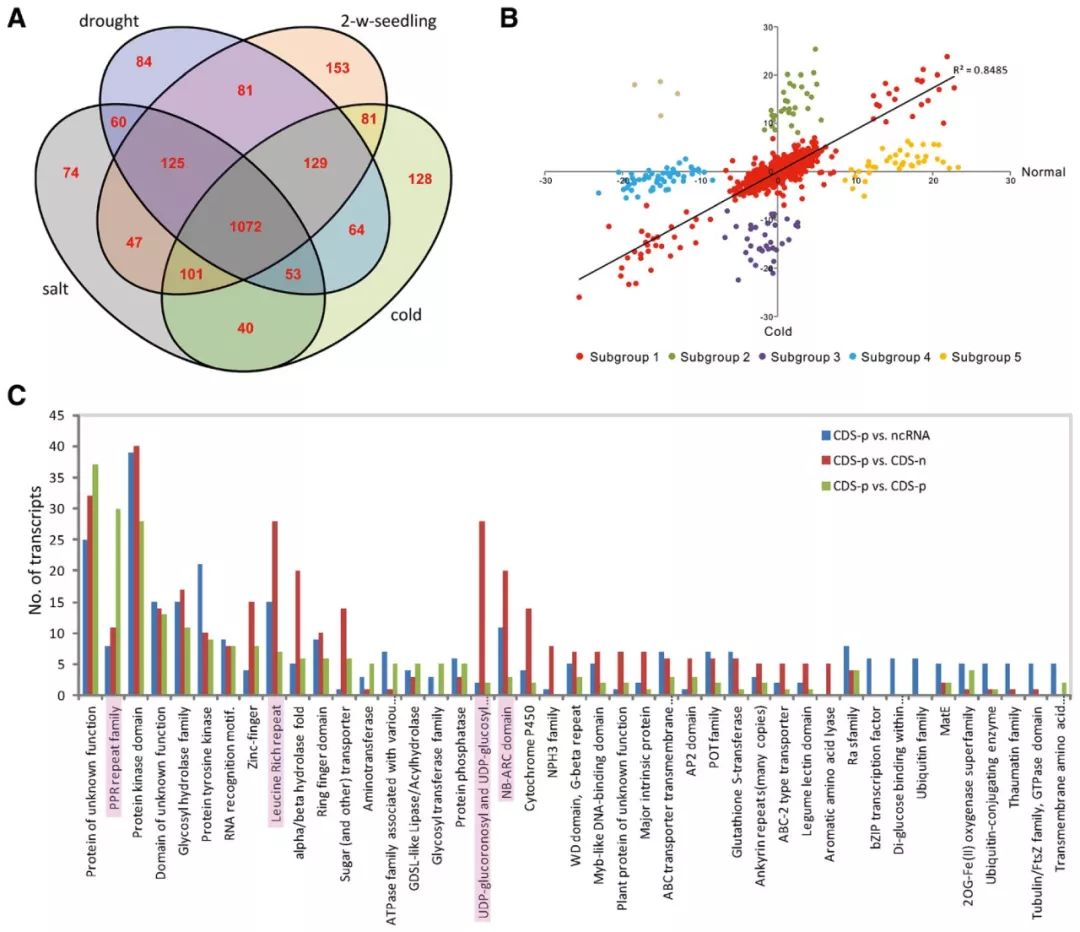
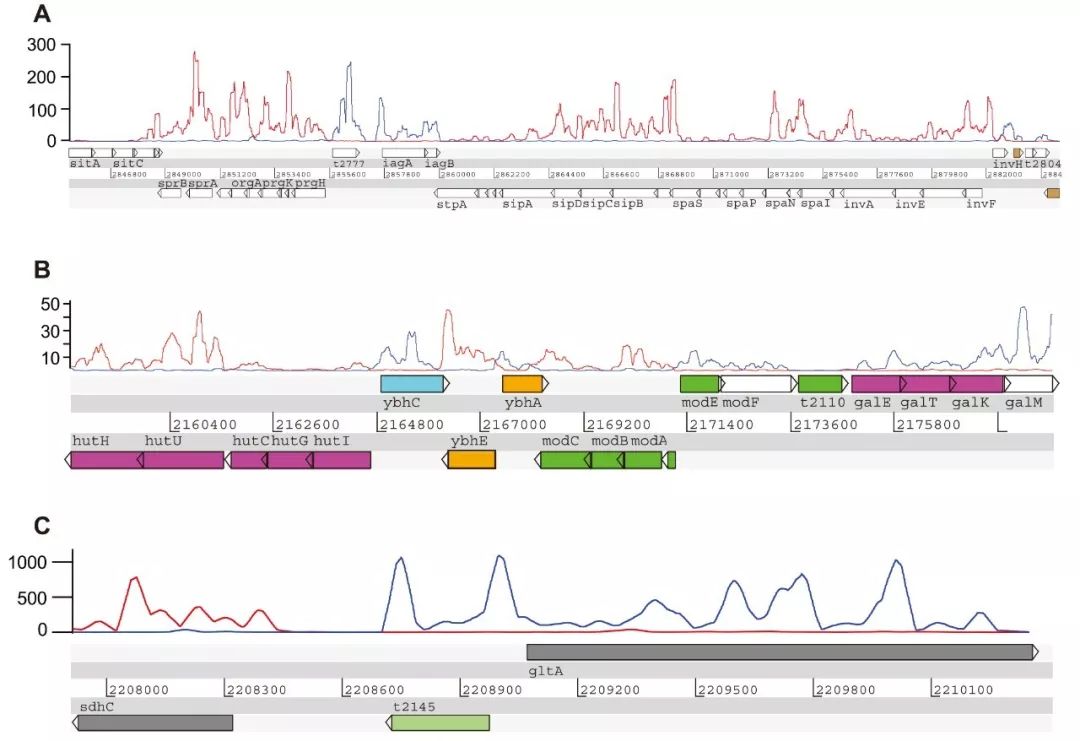
图3 水稻中鉴定到的一对一cis-NATs 为了全面鉴定水稻cis-NATs,文章采用链特异性文库对模式植物水稻(Oryza sativa L.)中具有明确方向的cDNA进行深度测序,收集在正常和三种非生物胁迫条件下(盐,冷和干旱处理)生长的水稻幼苗构建链特异性文库,以评估转录方向清晰的转录本。检测结果表明,大约89.4-95.5%的reads序列与正确的转录方向一致,与链特异性文库数据相反,之前的普通转录组文库数据中,只有一半的reads序列读取与相同转录方向的基因一致,证实了链特异性文库数据的可靠性,链特异性文库有利于发掘大量潜在顺式天然反义转录本。 03、对可变剪切事件的检测更准确 因为链特异性文库可以排除反义转录的影响,使得可变剪接事件的识别存在假阳性而降低。如发掘新的circRNA,circRNA由反向剪切形成,链特异性的测序方式有助于提高circRNA检测的准确率。 04、无参转录组组装更真实 在进行无参转录本组装时,明确转录本的方向性很有必要,普通转录组组装出来的unigene既包括编码转录本,也包括一些非编码转录本(比如lncRNA),但是如果不区分正反链,那么有互补配对关系的编码与非编码转录本会被组装成一条转录本,链特异性文库更有利于无参转录组组装。 05、有利于原核生物操纵子(operon)的分析 原核生物的基因为多顺反子结构,如果不区分反义转录本上的基因,其对应位置的基因表达量会计算不准确,操纵子及基因结构的预测也不准确。链特异性测序能够更准确地对操纵子进行分析与鉴定。
图4 Salmonella enterica serovar Typhi (S. Typhi)操纵子分析 说了这么多关于链特异性文库的事,相信大家在选择文库类型的时候也不会再纠结,链特异性文库做起来,来看一看微分的转录组产品。 微分转录组产品优势 1、建库成功率高>98%,测序质量好Q30>85% 2 、构建链特异性文库,减少比对错误,保证结果的准确性 3 、分析全面:标准分析+定制化分析 4 、一站式服务:上游测序+下游QPCR验证 参考文献: 1.Mills J D,Kawahara Y, Janitz M. Strand-specific RNA-seq provides greater resolution oftranome profiling[J]. Current genomics, 2013, 14(3): 173. 2.Park J W,Chariker J, Xing Y. Strand specific RNA-seq data for higherspecificity[C]//Proceedings of the 2015 Conference on research in adaptive andconvergent systems. ACM, 2015: 123-124. 3.Zhao S, Zhang Y, Gordon W, etal. Comparison of stranded and non-stranded RNA-seq tranome profiling andinvestigation of gene overlap[J]. BMC genomics, 2015, 16(1): 675 4.Lu T , Zhu C , Lu G , et al. Strand-specific RNA-seq reveals widespread occurrence of novel cis-natural antisense trans in rice[J]. BMC Genomics, 2012, 13(1):721. 5.Perkins T T , Kingsley R A , Fookes M C , et al. A Strand-Specific RNA–Seq Analysis of the Tranome of the Typhoid Bacillus Salmonella Typhi[J]. PLOS GENETICS, 2009, 5(7):0-0.返回搜狐,查看更多 |
【本文地址】