| 必须收藏!比较基因组学分析必备神器 | 您所在的位置:网站首页 › 基因组的同源性 › 必须收藏!比较基因组学分析必备神器 |
必须收藏!比较基因组学分析必备神器
|
全基因组直系同源基因簇(Orthologousclusters)的分析是比较基因组学研究中的一项重要内容,直系同源基因簇之间的聚类及网络构建有助于解释跨多物种的蛋白质的功能和进化关系,今天小编给大家分享由西南大学生物学研究中心王翊教授开发一款用于多物种全基因组直系同源基因簇比较和注释的在线工具—OrthoVenn2,非常简单好用! OrthoVenn2简介OrthoVenn2 是由西南大学生物学研究中心王翊教授开发,较之前版本OrthoVenn v 1.0版本,OrthoVenn2的性能、数据库、分析及结果展示方面都有很大的提升;OrthoVenn2基于OrthoMCL启发式匹配算法,用DIAMOND进行比对,比传统的BLAST算法速度提高了1万倍,OrthoVenn2基因组蛋白序列来源于Ensembl数据库,通用性更强,单次在线分析的上限物种数量提高了1倍达12个。OrthoVenn2还提供了本地版本下载,可以处理更大数据量的分析,无物种数限制,分析环境封装于docker镜像中,个人安装docker后直接下载OrthoVenn2镜像即可使用,还未安装docker或者不了解docker的朋友请点击链接学习:生信学习第一步!免费领取一套docker虚拟机!OrthoVenn2访问网址:https://orthovenn2.bioinfotoolkits.net/home。
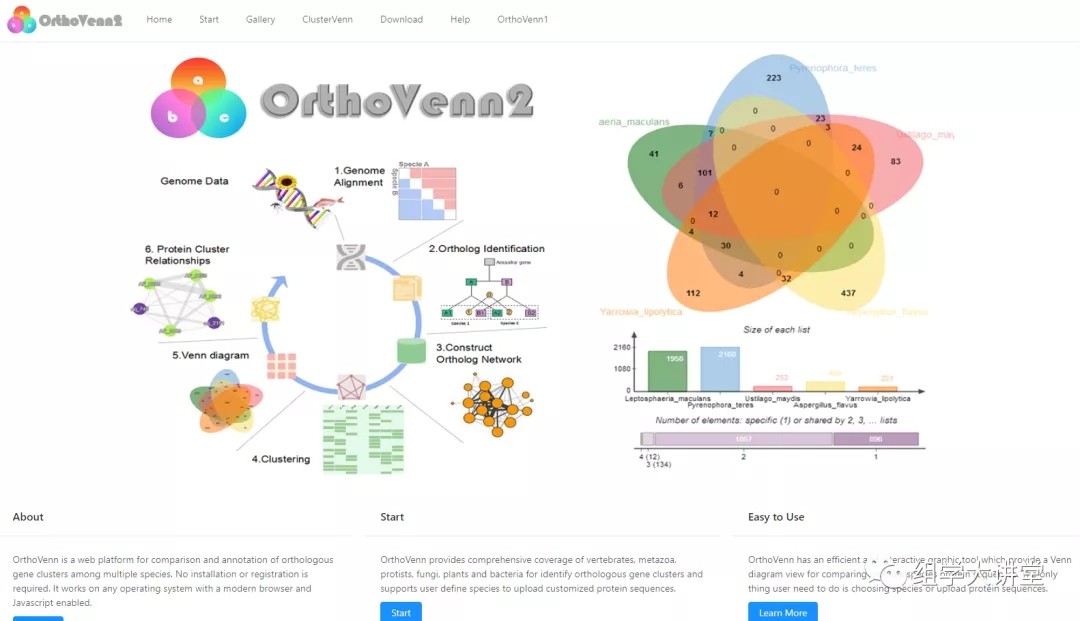
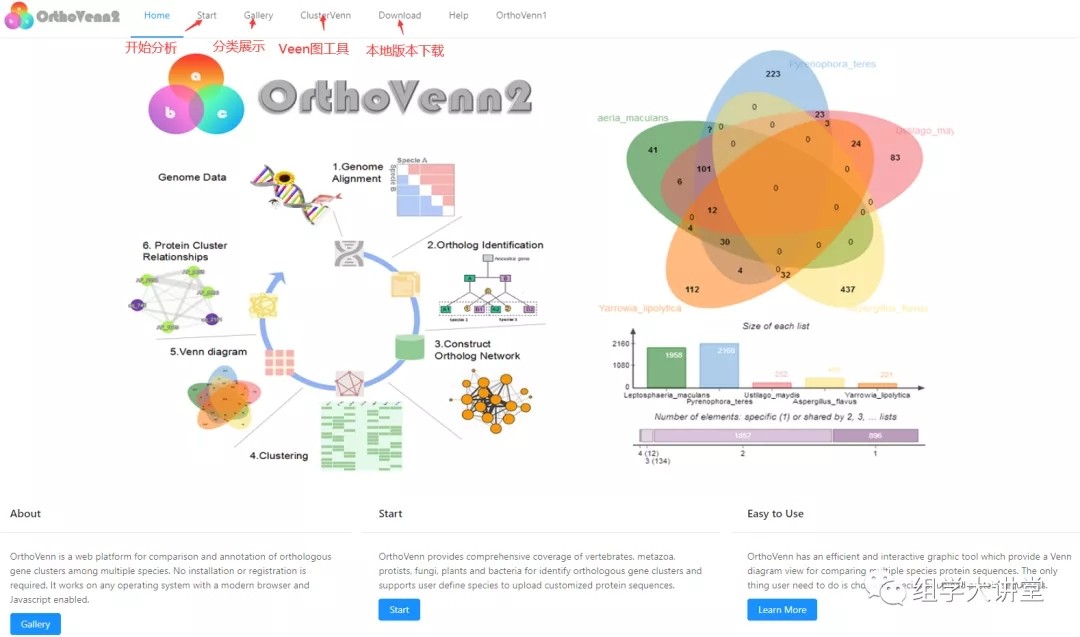
OrthoVenn2的操作十分简单,打开主页后,各功能项介绍如下图。
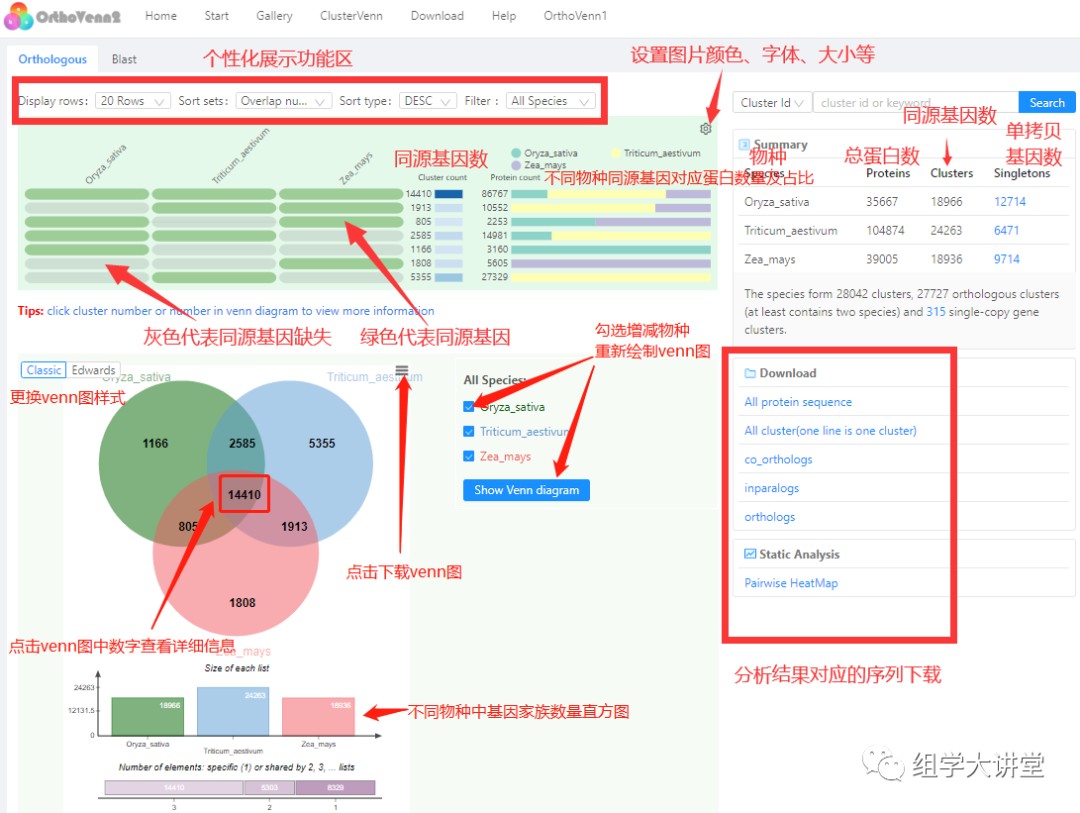
OrthoVenn2的结果展示很是丰富,较OrthoVenn1版增加了不同物种同源基因簇的存在或者缺失情况的直观显示及统计信息(绿色表示存在,灰色表示缺失)还可以通过个性化展示功能区进行调整展示内容,以及设置图片颜色、字体等;页面右侧则统计了各物种基因组蛋白数、基因家族数目、单拷贝基因序列数目,其下方还可直接下载序列聚类信息表以及同源基因簇的相应的蛋白序列。
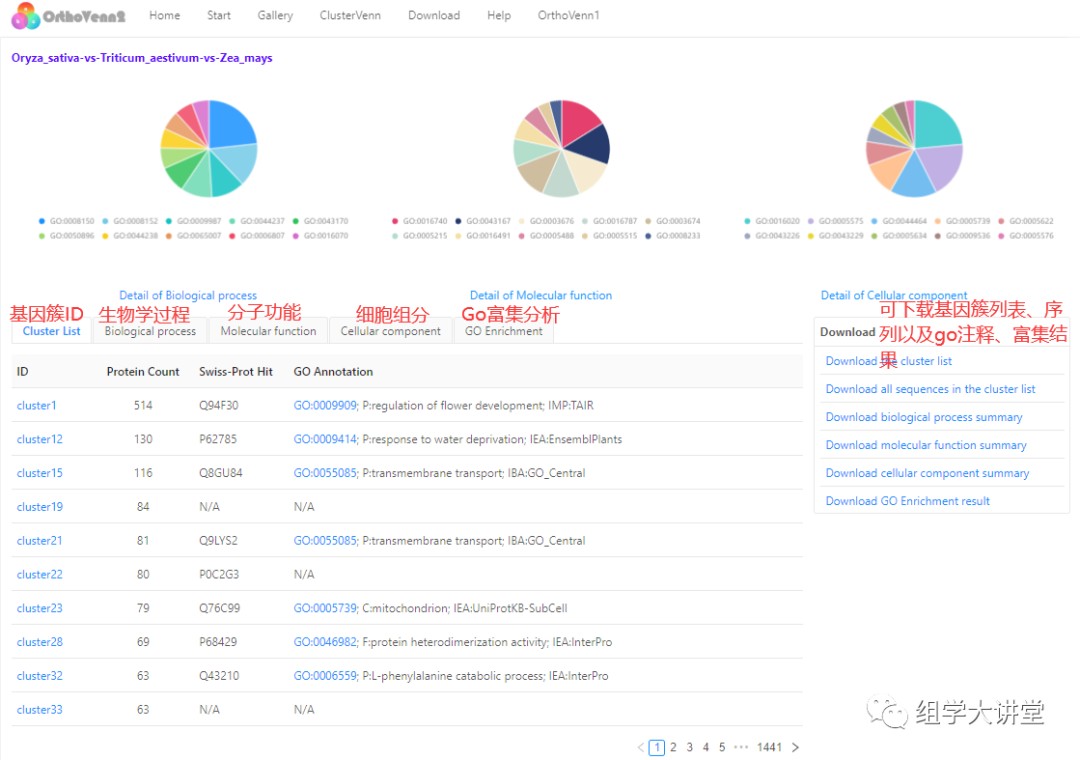
引申到跨物种比较基因组学中,该Venn图展示的是分别分布于核心基因组、非必须基因组以及特异基因中基因家族,如红框中的数字14410是3物种共有的核心基因家族;该Venn图共两种风格,还可以通过勾选增减物种数量重新绘制,Venn图下载见箭头所指,提供的图片格式包括PNG、PDF、SVG。Venn图中数字均可点击,打开后页面如下图,上方三个饼状图展示的是对应同源基因簇的GO功能类别的比例,包括生物过程(biological processes)、分子功能(molecular functions)和细胞成分(cellular components)3个层次的信息。饼图下方的列表中有基因簇蛋白数量、Swiss-Prot及GO(Gene Ontology)功能注释及富集分析的结果;右侧还可以下载基因家族聚类信息表以及该共有区域的相应序列,具体见下图标注信息。
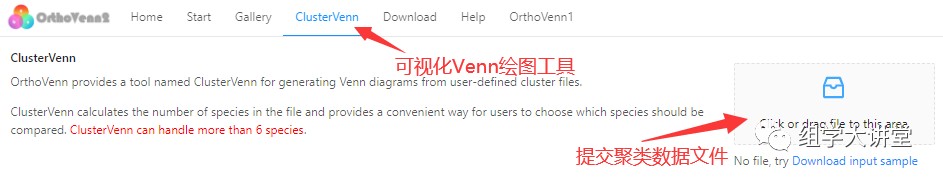
点开上图的基因簇编号 Cluster-,可以看到该基因簇包含蛋白ID、基因家族的相似性网络图,该网络图中两条序列(节点)连线越粗,表示两基因相似度越高,并且网络图中连线和节点都可以拖动,可以根据自己审美及展示需求进行调整。左下方链接还可以进一步查看蛋白质序列、多重序列比对、motif分析以及进化树,详见上图标注信息。 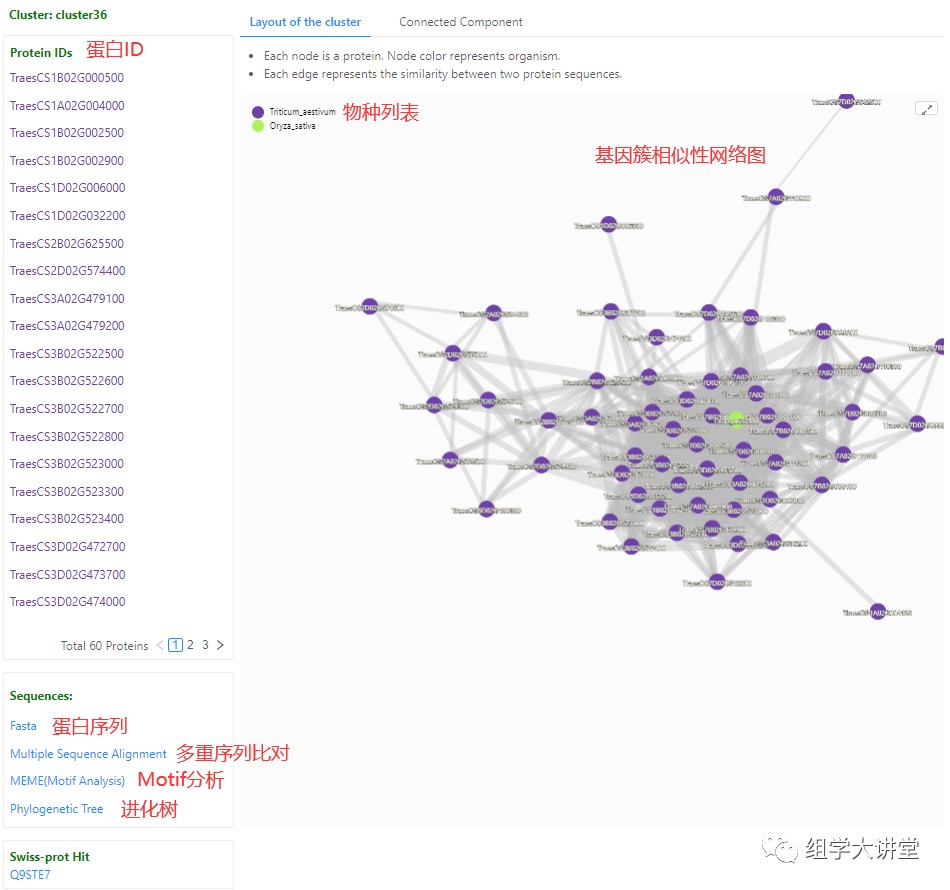 OrthoVenn2还有一个可视化工具ClusterVenn,支持本地上传的聚类数据文件来生成韦恩图。 OrthoVenn2还有一个可视化工具ClusterVenn,支持本地上传的聚类数据文件来生成韦恩图。
OrthoVenn2支持在线或本地使用,无需注册,分析内容丰富、功能强大,是一款不可或缺的比较基因组学研究工具,分享给大家,希望能对您可研有所帮助!最后,提醒大家,原创不易,使用该工具发文章时记得引用文献出处,参考文献见下方。参考文献:Ling Xu, Zhaobin Dong, Lu Fang, Yongjiang Luo, Zhaoyuan Wei, HailongGuo, Guoqing Zhang, Yong Q Gu, Devin Coleman-Derr, Qingyou Xia, Yi Wang. OrthoVenn2: a web server for whole-genome comparison and annotation oforthologous clusters across multiple species, Nucleic Acids Research, Volume 47, Issue W1, 02 July 2019, Pages W52–W58, https://doi.org/10.1093/nar/gkz333. 1. 文章越来越难发?是你没发现新思路,基因家族分析发2-4分文章简单快速,学习链接:基因家族分析实操课程 2. 转录组数据理解不深入?图表看不懂?点击链接学习深入解读数据结果文件,学习链接:转录组(有参)结果解读;转录组(无参)结果解读 3. 转录组数据深入挖掘技能-WGCNA,提升你的文章档次,学习链接:WGCNA-加权基因共表达网络分析 4. 转录组数据怎么挖掘?多学点数据处理技能:学习链接:转录组标准分析后的数据挖掘 5. 微生物16S/ITS/18S分析原理及结果解读 6. 学生物的必学生信技能:linux系统入门 7. 学生物的必学生信技能:Perl语言入门到精通 8. 学生物的必学生信技能:perl语言高级编程 9. 更多学习内容:linux、perl、R语言画图,更多免费课程请扫描下方二维码:
|
【本文地址】
 点击Start即进入分析页面,如下图:可分析物种分为脊椎动物、植物、多细胞动物、原生生物、真菌、细菌六大类,点击下图圆形彩色物种目录即可进入下一级物种目录,选择物种后,右侧会展示所选物种列表;另一方面,该网站支持本地基因组蛋白质序列上传(fasta格式)。
点击Start即进入分析页面,如下图:可分析物种分为脊椎动物、植物、多细胞动物、原生生物、真菌、细菌六大类,点击下图圆形彩色物种目录即可进入下一级物种目录,选择物种后,右侧会展示所选物种列表;另一方面,该网站支持本地基因组蛋白质序列上传(fasta格式)。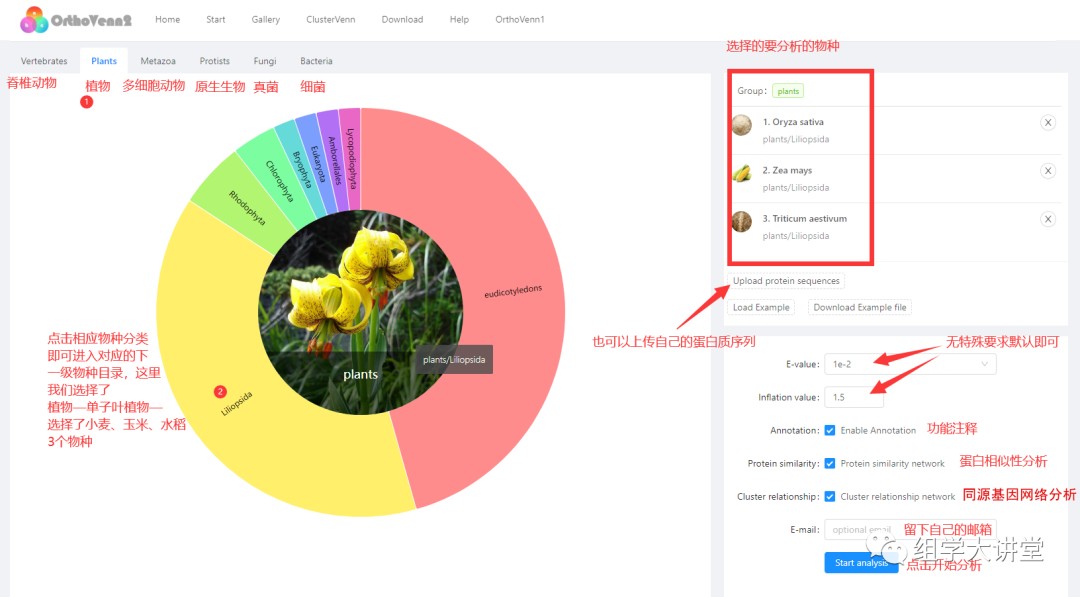 任务提交后,系统会自动生成任务号,分析速度还是蛮快的,小编选了小麦、玉米、水稻三个物种,仅30分钟就分析完成,最终结果会以链接形式发送到预留的邮箱中,结果形式如下图:
任务提交后,系统会自动生成任务号,分析速度还是蛮快的,小编选了小麦、玉米、水稻三个物种,仅30分钟就分析完成,最终结果会以链接形式发送到预留的邮箱中,结果形式如下图: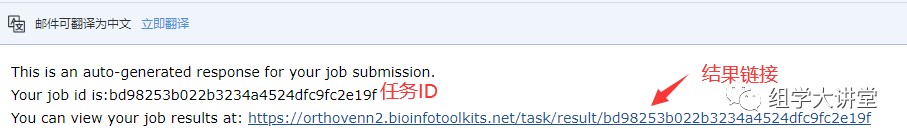
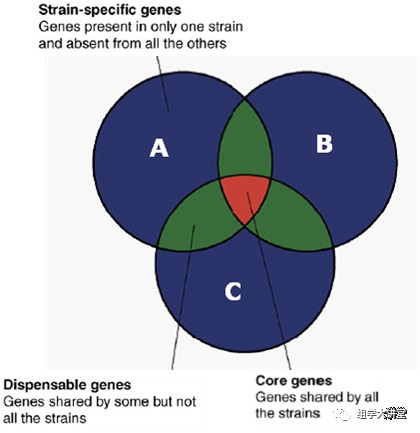
 最醒目的Venn图部分展示的是不同物种全基因组基因家族的交集信息,理解该Venn图,我们首先需要理解以下概念:我们常听说泛基因组(pan genome),是指某一物种全部基因的总称,但由于物种内也存在分化,所以并不是这个物种中所有个体的基因组成都是一样的,所以,泛基因组可分为三个部分:1. 核心基因组(core genome),即在所有个体中都存在的基因,该类基因非常保守;2. 非必须基因组(dispensable genome)是指存在于部分个体中的基因,与物种的分化有关,利于个体竞争;3. 特有基因(specific genes)是指只存在于某一个体中的基因,常与该个体的独特表型相关,如对特定环境的适应性或独特的抗病性等。
最醒目的Venn图部分展示的是不同物种全基因组基因家族的交集信息,理解该Venn图,我们首先需要理解以下概念:我们常听说泛基因组(pan genome),是指某一物种全部基因的总称,但由于物种内也存在分化,所以并不是这个物种中所有个体的基因组成都是一样的,所以,泛基因组可分为三个部分:1. 核心基因组(core genome),即在所有个体中都存在的基因,该类基因非常保守;2. 非必须基因组(dispensable genome)是指存在于部分个体中的基因,与物种的分化有关,利于个体竞争;3. 特有基因(specific genes)是指只存在于某一个体中的基因,常与该个体的独特表型相关,如对特定环境的适应性或独特的抗病性等。