|
文章目录
介绍查询某个物种的全部核酸序列和蛋白序列查看某个物种的其他信息(蛋白结构,基因,测序数据,相关文献等)Taxonomy 的相关数据下载1. gi_taxid 标识的数据2. taxcat 标识的数据以尼安德特人(taxid:63221)为例
3. taxdump 标识的数据
介绍
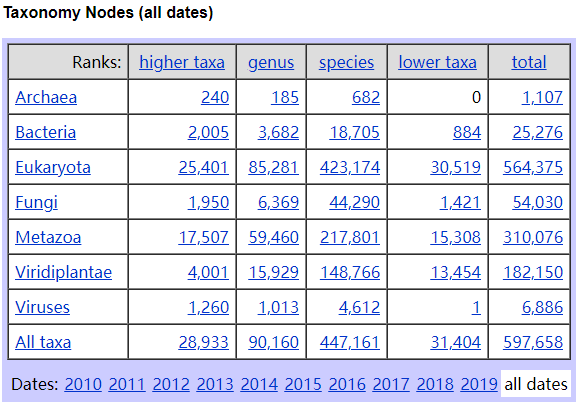
Taxonomy : NCBI公共序列数据库中所有生物的策划分类和命名法。目前包含地球上大概10%的物种。 我们现在查询到底包含有有多少物种,进入统计页面:。可以看到不同的分类下的分布情况,总体包含有597658条物种信息。
查询某个物种的全部核酸序列和蛋白序列
进入 NCBI 首页 点击Taxonomy,进入物种分类数据库
进入 Taxonomy 首页,输入human,点击Search
浏览该物种下的核酸序列或蛋白序列,直接点击Nucleotide或者Protein 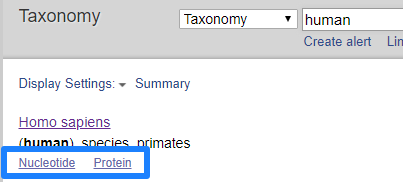
浏览核酸序列列表,数量远远超过了所预想的数量,因为这里包含的是与 Nucleotide 相关的该物种的信息
选择左栏的Viruses切换显示物种,可以看到有好多病毒的整合位点信息。你也可以点击左栏来筛选其他你想要的信息,比如mRNA。 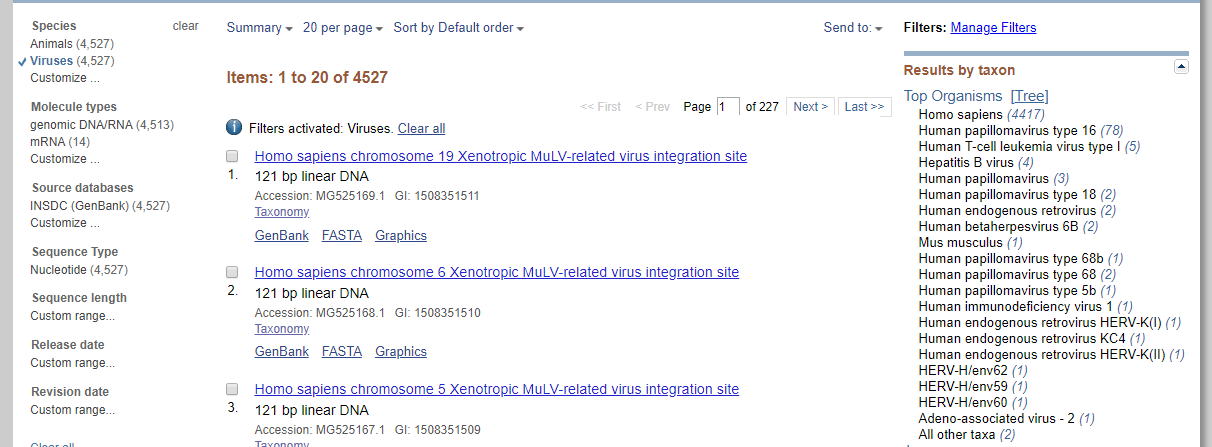
查看某个物种的其他信息(蛋白结构,基因,测序数据,相关文献等)
https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?mode=Root
进入首页,我们以人类为例:输入human,点击Go 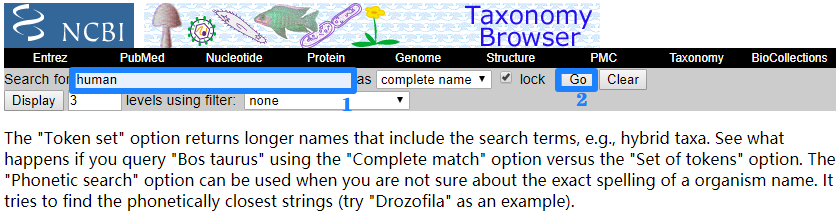
点击Homo sapiens
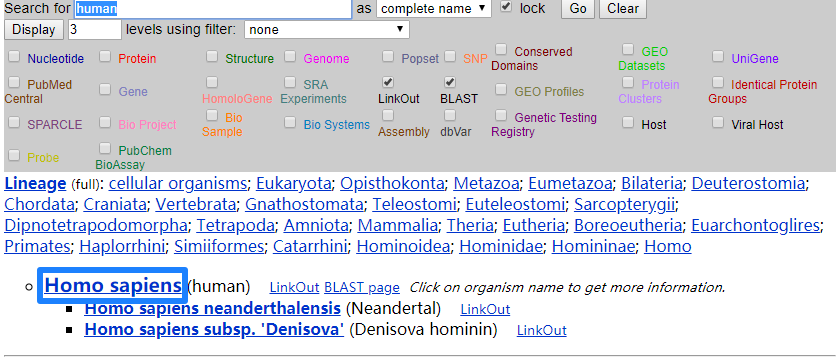
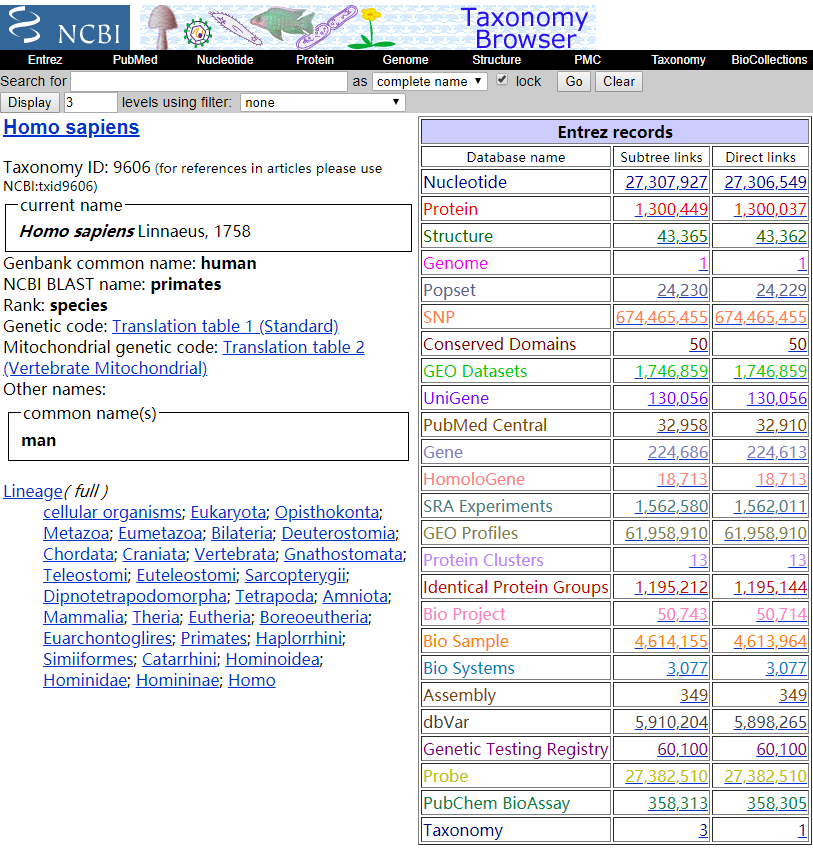
大家会看到在NCBI中关于人类的目前几乎全部的生物数据。左栏显示人类拉丁名Homo sapiens,Taxonomy编号为txid9606,基因密码子表,线粒体密码子表等。
右栏展示与人相关的数据,常用的包括
Nucleotide: 核酸序列Protein: 蛋白序列Structure: 蛋白结构(大部分来源于PDB数据库)SNP: 单位点突变数据GEO Datasets/SRA Experiments/GEO Profiles: 用于储存公共测序数据,这个包含之前的芯片数据,也有目前大部分的高通量测序PubMed Central: 文献Gene: 基因信息
Taxonomy 编号在查询和标注信息时候常常用到,比如,在Nucleotide中查询现代智人的时候:

Taxonomy 的相关数据下载
ftp://ftp.ncbi.nih.gov/pub/taxonomy/
1. gi_taxid 标识的数据
NCBI早在2016年已经宣布逐渐停用,这部分信息不再关注
2. taxcat 标识的数据
ncbi提供有不同格式的压缩包,解压后都只有一个categories.dmp文件。打开该文件,包含三列信息,三列代表的不同的分类层次。
第一列:代表分类的顶级类别(top-level category),字母分别代表不同分类名(古菌,细菌,真核生物,病毒和类病毒,未分类,其他)
A = Archaea B = Bacteria E = Eukaryota V = Viruses and Viroids U = Unclassified O = Other
第二列:相应的物种级别(species-level)的taxid 第三列:taxid本身
以尼安德特人(taxid:63221)为例
查看categories.dmp文件(下面命令代表去categories文件中查找63221并显示):
cat categories.dmp | grep 63221
结果如下,第一行即为63221(taxid)代表尼安德特人:
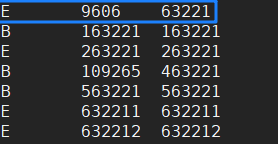
我们现在可以描述尼安德特人(taxid:63221)属于真核生物(E)里的智人(taxid:9606)类的一个分支。
3. taxdump 标识的数据
同样提供不同格式的压缩包,解压gunzip -c taxdump.tar.gz | tar xf -后包含7个文件:
citations.dmp:与某个物种(taxid表示)的文献信息: 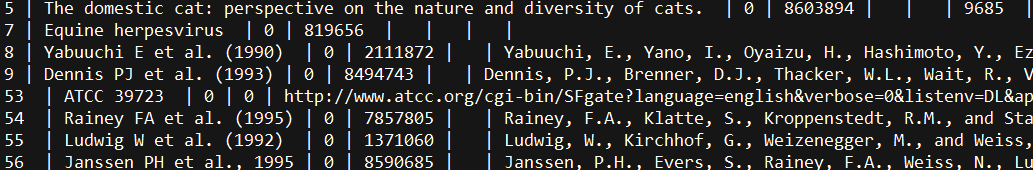
it_id :the unique id of citationcit_key:citation keymedline_id:unique id in MedLine database (0 if not in MedLine)pubmed_id:unique id in PubMed database (0 if not in PubMed)url:URL associated with citationtext :any text (usually article name and authors) :The following characters are escaped in this text by a backslash: :newline (appear as “\n”), :tab character ("\t"), :double quotes (’"’), :backslash character ("\").taxid_list:list of node ids separated by a single space names.dmp:存储 taxid 对应的物种名信息 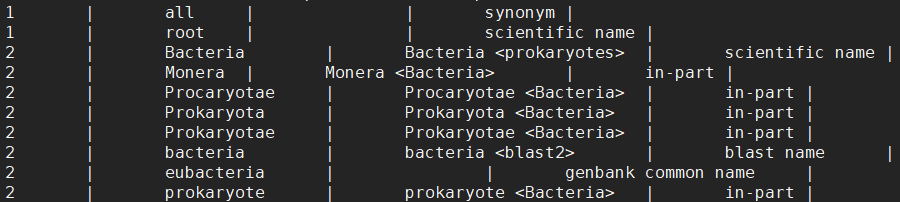
tax_id:the id of node associated with this namename_txt:name itselfunique name:the unique variant of this name if name not uniquename class:(synonym, common name, …) nodes.dmp:存储 taxid对应的多级节点信息 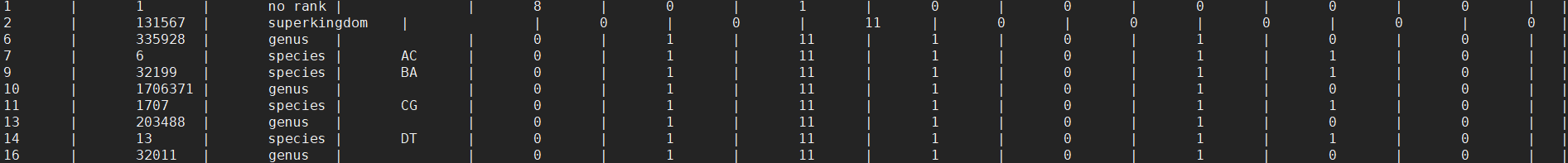
tax_id:node id in GenBank taxonomy databaseparent tax_id:parent node id in GenBank taxonomy databaserank:rank of this node (superkingdom, kingdom, …)embl code:locus-name prefix; not uniquedivision id:see division.dmp fileinherited div flag (1 or 0): 1 if node inherits division from parentgenetic code id:see gencode.dmp fileinherited GC flag (1 or 0): if node inherits genetic code from parentmitochondrial genetic code id: – see gencode.dmp fileinherited MGC flag (1 or 0): – 1 if node inherits mitochondrial gencodeGenBank hidden flag (1 or 0) : – 1 if name is suppressed in GenBank entryhidden subtree root flag (1 or 0) : – 1 if this subtree has no sequence data yetcomments:free-text comments and citations delnodes.dmp:已经删除不用的节点信息 
division.dmp: 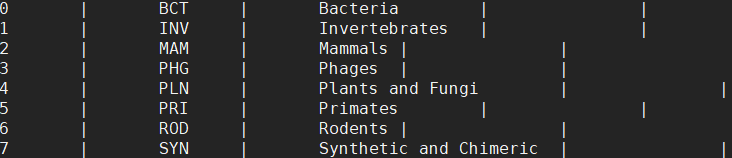
division id:taxonomy database division iddivision cde:GenBank division code (three characters)division name:e.g. BCT, PLN, VRT, MAM, PRI…comments gencode.dmp:密码子表信息 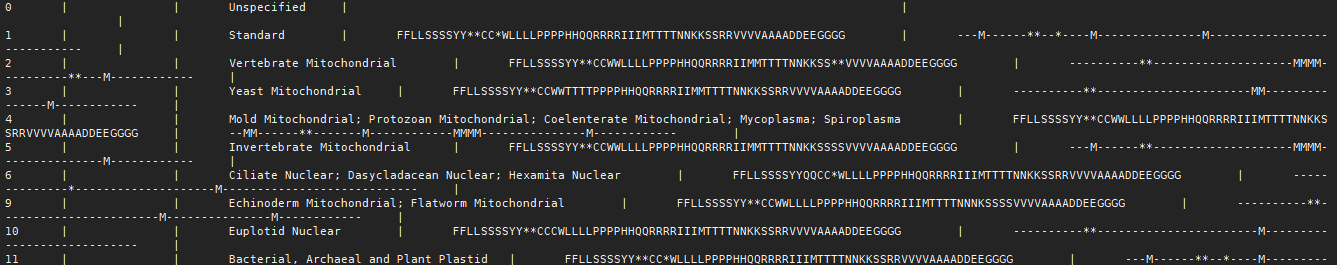
genetic code id:GenBank genetic code idabbreviation:genetic code name abbreviationname:genetic code namecde:translation table for this genetic codestarts:start codons for this genetic code merged.dmp:记录新taxid替换旧taxid的信息 
old_tax_id:id of nodes which has been mergednew_tax_id:id of nodes which is result of merging
|