| Scikit | 您所在的位置:网站首页 › sklearn归一化还原 › Scikit |
Scikit
|
本系列文章介绍人工智能的基础概念和常用公式。由于协及内容所需的数学知识要求,建议初二以上同学学习。 运行本系统程序,请在电脑安装好Python、matplotlib和scikit-learn库。相关安装方法可自行在百度查找。
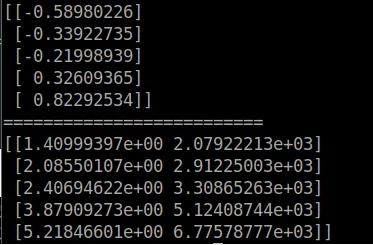
PCA,主成份分析法。它是一种维数约减算法,即将高维数据转换成低维数据的算法。它是用来对数据进行压缩的算法,在可控的失真范围内提高运算速度。 高维数据转换成低维数据可以理解为一种高维向低维投影的过程。比如三维环境中的物品投射到二维屏幕的过程。这就需要一个投射转换的视图矩阵。这个矩阵在PCA算法里叫主成份特征矩阵。 PCA的转换过程 样本数据归一化,方便降维。计算主成份特征矩阵。利用主成份特征矩阵对样本数据进行降维。 示例说明至于降维后的数据用什么方法进行机器学习,由使用者决定。PCA只负责降维,精简数据。下面我们来看一个用PCA降维的例子。 有5个记录,每个记录有2个特征值(二维数据)。 A = [ [1,2500],[2,3000],[3,2700],[4,5000],[5,7000] ]不同特征值差别太大,对其进行归一化。利用numpy库上的svd函数计算主成份特征矩阵。利用特征矩阵计算降维后数据。对降维后数据进行恢复,比较与原始数据的差异。 示例程序1 import numpy as np A = [ [1,2500],[2,3000],[3,2700],[4,5000],[5,7000] ] A = np.array(A) #归一化处理 mean = np.mean(A,axis=0) norm = A - mean #数据缩放 scope = np.max(norm,axis=0) - np.min(norm,axis=0) norm = norm / scope #计算主成份特征矩阵 U,S,V = np.linalg.svd(np.dot(norm.T,norm)) #由于是二维降一维,所以主用第一列数据 U_reduce = U[:,0].reshape(2,1) ret = np.dot(norm,U_reduce) #输出降维后数据 print(ret) print("==========================") #降维后数据恢复 a = np.dot(ret,U_reduce.T) a = np.multiply(a,scope)+mean print(a)
Scikit里面也有PCA降维的工具包,不过它只负责PCA部份处理,数据的归一化、缩放的预处理需要用户自已完成。下面我们来看下例子。 import numpy as np from sklearn.decomposition import PCA from sklearn.pipeline import Pipeline from sklearn.preprocessing import MinMaxScaler def std_PCA(**argv): #归一化、数据缩放 scaler = MinMaxScaler() pca = PCA(**argv) pipeline = Pipeline([('scaler',scaler),('pca',pca)]) return pipeline A = [ [1,2500],[2,3000],[3,2700],[4,5000],[5,7000] ] A = np.array(A) #初始化PCA函数,降成一维 pca = std_PCA(n_components=1) #开始降维 ret = pca.fit_transform(A) #输出降维后数据 print(ret) print("==========================") #降维后数据恢复 print(pca.inverse_transform(ret))
从运算结果我们可以看出,对数据的还原结果是一样的,只是数据降维后运算符不同了。这不是错误,只是降维后大家用的坐标系不同。
|
【本文地址】
公司简介
联系我们