| R语言(五) | 您所在的位置:网站首页 › loglog图r语言 › R语言(五) |
R语言(五)
|
目录 一、数据 二、logistic回归 1.拟合 2.预测 三、probit回归 四、经典判别分析(线性、混合线性、灵活线性) 五、交叉验证与比较 一、数据脊柱数据(Column_2C.csv、Column_3C.csv)有两个版本,区别在于分为两类还是3类。数据可以从网站http://archive.ics.uci.edu/ml/datasets/Vertebral+Column下载,不过是.dat文件,需要进行相应的转换 参考我这篇博客Python将.dat文件转换成.csv文件。或者直接下载我上传的文件,是已经对格式和数据经过处理,可以直接进行分析的csv文件。 数据具有6个自变量(生物力学特征V1-V6,都是数量),和类别(V7) 两个数据内容分别为 类别数量(人)代码C2正常100NO不正常210ABC3正常100NO椎间盘突出60DH腰椎滑脱150SL 二、logistic回归 1.拟合 #logistic #拟合 w2=read.csv("column_2C.csv") a=glm(V7~.,w2,family="binomial") #默认logit连接函数 b=step(a) #做逐步回归筛选变量 summary(b) #结果汇总
在逐步回归过程中,变量3和4被淘汰,输出的AIC为188.95 2.预测通过逐步回归或其他方法挑选的模型可能有助于解释模型,但不一定对预测有帮助。因此在我们分析的步骤中,还是利用逐步回归之前的a拟合模型 R自动把p(概率)作为排序靠后的类别的概率,于是运行代码levels(w2$V7)时,得到“AB”“NO”,p指的是NO的概率(R按照字典排序) 按照总误判率来寻找p值得阈值的函数如下: BI=function(D,w,ff,fm="binomial"){ a=glm(ff,w,family=fm) z=predict(a,w,type="response") ee=NULL for(p in seq(.1,.9,.01)){ u=rep(levels(w[,D])[2],nrow(w)) u[!(z>p)]=levels(w[,D])[1] e=sum(u!=w[,D])/nrow(w) #e=sum(u=="NO"&w[,D]=="AB")/sum(w[,D]=="AB") ee=rbind(ee,c(p,e)) } I=which(ee[,2]==min(ee[,2])) P=ee[min(I),1] return(ee[min(I),]) }其中D代表因变量是第几个变量,w为使用数据,ff是拟合公式,fm是模型。在此函数中,p值从0.1到0.9按0.01的间隔搜索。调用函数 p.2C=BI(7,w2,V7~.)得到阈值0.63,误判率0.1322。运行以下代码查看判别结果 #判别结果 D=7;p=p.2C[1];a=glm(V7~.,w2,family = "binomial") z=(predict(a,w2,type="response")>p) u=rep(levels(w2[,D])[2],nrow(w2)) u[!(z>p)]=levels(w2[,D])[1] zz=table(w2[,7],u); #混淆矩阵 sum(diag(zz))/sum(zz) #正确率
还可以修改函数以改进效果,可以改变e的判别方式 三、probit回归由于该数据的变量V6有出错信息,但我们又不愿意去掉该变量,因此代码中改用probit选项,此时不能计算AIC,也不能用逐步回归选择变量,但还是用BI()函数选择阈值 D=7;w=w2;ff=V7~.;fm=quasibinomial(link="probit") pp.2C=BI(D,w2,ff,fm) D=7;p=pp.2C[1];a=glm(V7~.,w2,family=fm) summary(a) z=(predict(a,w,type="response")>p) u=rep(levels(w[,D])[2],nrow(w)) u[!(z>p)]=levels(w[,D])[1] zz=table(w2[,7],u); #混淆矩阵 sum(diag(zz))/sum(zz) #正确率得到的probit回归的估计系数及近似正态z检验的结果和分类展示如下,可知训练出来的模型:
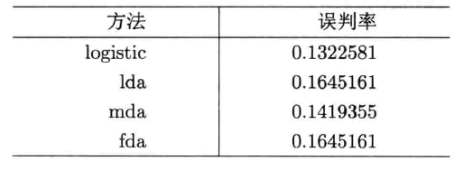
三者代码基本相似,只是部分地方有微小差别 #线性判别分析 library(MASS) w2=read.csv("column_2C.csv") a=lda(V7~.,data=w2) b=predict(a,w2)$class zz=table(w2[,7],b) #混淆矩阵 sum(diag(zz))/sum(zz) #正确率 #混合线性判别分析 library(mda) w2=read.csv("column_2C.csv") set.seed(1010) a=mda(V7~.,data=w2) b=predict(a,w2) zz=table(w2[,7],b) #混淆矩阵 sum(diag(zz))/sum(zz) #正确率 #灵活线性判别分析 library(splines) library(Matrix) library(fda) w2=read.csv("column_2C.csv") set.seed(1010) a=fda(V7~.,data=w2) b=predict(a,w2) zz=table(w2[,7],b) #混淆矩阵 sum(diag(zz))/sum(zz) #正确率 五、交叉验证与比较把数据随机分成Z份(此处Z=10),使用以下函数划分数据: #产生数据集 Fold=function(Z=10,w,D,seed=7777){ n=nrow(w);d=1:n;dd=list() e=levels(w[,D]) t=length(e) set.seed(seed) for(i in 1:t){ d0=d[w[,D]==e[i]] j=length(d0) ZT=rep(1:Z,ceiling(j/Z))[1:j] id=cbind(sample(ZT,length(ZT)),d0) dd[[i]]=id } #每个dd[[i]]是随机1:Z及i类的下标集组成的矩阵 mm=list() for(i in 1:Z){ u=NULL for(j in 1:t)u=c(u,dd[[j]][dd[[j]][,1]==i,2]) mm[[i]]=u #mm[[i]]为第i个下标集 } return(mm) #输出Z个下标集 }运行logistic、lda、mda、fda判别分析的交叉验证,代码如下: BI=function(D,w,ff,fm="binomial"){ a=glm(ff,w,family=fm) z=predict(a,w,type="response") ee=NULL for(p in seq(.1,.9,.01)){ u=rep(levels(w[,D])[2],nrow(w)) u[!(z>p)]=levels(w[,D])[1] e=sum(u!=w[,D])/nrow(w) #e=sum(u=="NO"&w[,D]=="AB")/sum(w[,D]=="AB") ee=rbind(ee,c(p,e)) } I=which(ee[,2]==min(ee[,2])) P=ee[min(I),1] return(ee[min(I),]) } #读数据 w=read.csv("column_3C.csv") D=7;Z=10 mm=Fold(Z,w,D,7777) ff=paste(names(w)[D],"~.",sep="") ff=as.formula(ff) #logistic回归用最优阈值 p=BI(D,w,ff)[1] ERROR=matrix(0,Z,4) J=1 for(i in 1:Z){ m=mm[[i]] a=glm(V7~.,w,family="binomial") z=(predict(a,w[m,],type="response")>p) #测试集预测 u=rep(levels(w[m,D])[2],length(m)) u[!z]=levels(w[m,D])[1] ERROR[i,J]=sum(w[m,D]!=u)/length(m) } J=J+1 for(i in 1:Z){ m=mm[[i]] a=lda(ff,w[-m,]) ERROR[i,J]=sum(w[m,D]!=predict(a,w[m,])$class)/length(m) } J=J+1 for(i in 1:Z){ m=mm[[i]] set.seed(1010) a=mda(ff,w[-m,]) ERROR[i,J]=sum(w[m,D]!=predict(a,w[m,]))/length(m) } J=J+1 for(i in 1:Z){ m=mm[[i]] a=fda(ff,w[-m,]) ERROR[i,J]=sum(w[m,D]!=predict(a,w[m,]))/length(m) } ERROR=data.frame(ERROR) names(ERROR)=c("logistic","lda","mda","fda") #ERROR ME=apply(ERROR,2,mean)得到各种方法的误判率和平均误判率
三分类数据同上,只需改变读取的数据集。同时三分类不能使用logistic方法 |
【本文地址】

 混淆矩阵如图,正确率为0.871
混淆矩阵如图,正确率为0.871