| 分子动力学模拟VMD基础教程有哪些? | 您所在的位置:网站首页 › lammps后处理 › 分子动力学模拟VMD基础教程有哪些? |
分子动力学模拟VMD基础教程有哪些?
|
分子动力学模拟VMD基础教程有哪些?windows下分子动力学模拟可视化工具VMD的安装与基本使用方法 VMD是一款非常小而精致的可视化工具,也是业界非常常用且功能强大的分子动力学可视化软件。本期内容一木就为大家介绍这款软件的下载安装和基本使用方法。01下载与安装下载VMD需要先注册账号,使用自己邮箱注册即可,注册完成后登陆即可免费下载。VMD下载地址如下:https://www.ks.uiuc.edu/Development/Download/download.cgi?PackageName=VMD  其他平台: 02基本操作 ①界面介绍 打开VMD后,会打开三个窗口,分别为:主窗口(VMD Main),显示窗口(VMD OpenGLDisplay),和命令窗口。 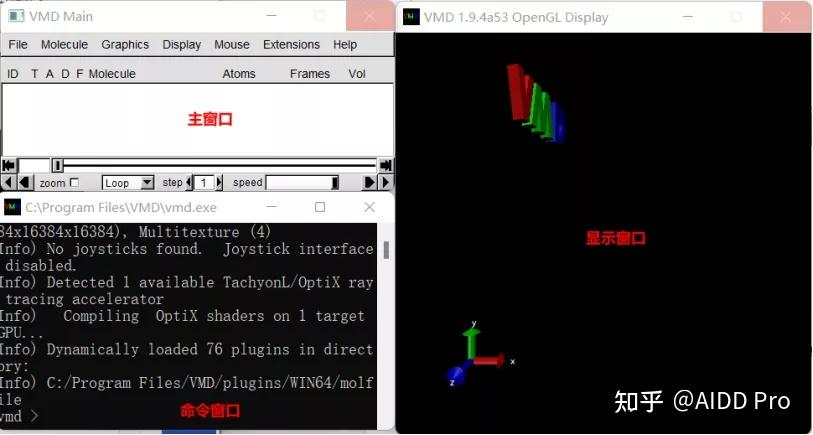 下面我们导入一个蛋白结构,在VMD主窗口的菜单栏点击File->New Molecule打开Molecule File Browser窗口;点击Browse选择要导入的文件,注意目录名、文件名都不应出现中文;Detemine file type可设置文件类型,一般可自动读取。  在Linux系统下,可在终端直接输入命令来导入文件,如: vmd complex.pdb不要忘记点击Load按钮,此时,导入的结构以默认的显示方案显示在显示窗口中。  ②改变视角 现在将鼠标移至显示窗口中,按住鼠标左键拖动,可进行旋转;按住右键左右拖动,蛋白质结构会垂直于屏幕旋转;滑动鼠标滚轮,可将蛋白放大或缩小。  VMD中有多种鼠标模式,默认的鼠标模式是Rotate Mode(旋转模式,R),以上即为R模式的操作方式。在主窗口菜单栏的Mouse菜单,可将鼠标模式改为Translate Mode(移动模式,T)或Scale Mode(缩放模式,S)  T模式下,按住鼠标左键可移动结构,按下鼠标中键来改变剪切板  S模式下,按住鼠标左键水平移动来缩小或放大分子,这种缩放不同于R模式下滑动鼠标中键的缩放,大家可自行感受。  此外,Center(中心模式,C)十分有用,可确定分子绕之旋转的支点。C模式下,鼠标在显示窗口内为十字。将十字放在想要作为旋转中心的原子上点一下,再按住鼠标左键旋转,就会以新定义的中心进行旋转。  主窗口中菜单栏点击Display->Reset View,或者在显示窗口内点击“=”键,可将当前显示的对象居中。 ③修改显示样式 下面我们对对象的显示模式进行修改。 在主窗口中菜单栏点击Graphics->Representations,打开GraphicalRepresentations窗口; 在Selected Molecule的下拉菜单中可选择不同的对象,即指定对哪个对象设定显示方式; 点击Create Rep可创建新的表示,可创建多个,效果可叠加,以此可完成复杂的绘图; 点击Delete Rep可删除表示;  下图部分为各种表示的当前状态,由三个元素构成。Style为样式显示方案;Color为颜色显示方案;Selection为内容显示方案。如此时蛋白结构以线状显示,根据名称进行着色,所有的原子都显示。  下图中可以设置当前选择的原子,Coloring Method的下拉菜单可选择着色方案;Drawing Method的下拉菜单可选择显示样式;Material的下拉菜单可选择显示材质。  下面来看一些不同的显示方案。  每一种Drawing Method都可以再进一步设置显示效果。比如对于CPK,可以调整原子球的大小(Sphere Scale)、改变化学键的粗细(Bond Radius)、以及设置更高或更低的分辨率(Sphere/Bond Resolution)。  同样的方式,大家可自行去探索不同的着色方案和不同材质的显示效果。 ④选择对象方式 在Graphical Representations窗口中选择Selections标签,在Singlewords部分中可以看到可以输入的选项列表。  如在Selected Atoms的文本输入框输入“helix”,可在显示窗口中只显示螺旋结构。  也可使用逻辑词“and/or/not”组合用于选择时的文本输入。如输入“not helix”来选择除了螺旋外的其它部分。  Selections标签的Keyword栏可根据蛋白质某些部分的特定值来进行选择。同样的可使用Keyword和Value组成的词组输入Selected Atoms的文本输入框。  如输入“resname LYS”可选择蛋白结构中所有的赖氨酸。  此外,within命令会经常用来范围选择,如输入“waterwithin 3 of protein” 来选择距离蛋白质3埃之内的所有的水分子。输入“within 5 of protein”来选择距离蛋白质5埃之内的所有原子。 ⑤更改背景色 在主窗口菜单栏点击Graphics->Colors打开Color Controls窗口,可对颜色进行自由设定。Categories选择Display;Names选择Background;Colors选择White即可将背景色改为白色。需要修改其它对象的颜色也是类似的。  ⑥隐藏坐标轴 在主窗口菜单栏点击Display->Axes->off即可隐藏坐标轴。  VMD(Visual Molecular Dynamics)教程(1) VMD(Visual Molecular Dynamics)教程(1)原文: VMD是专门为建模、可视化和分析生物大分子(如蛋白质、核酸等)。可读取PDB文件并显示,并提供多种分析、可视化选择、渲染方法,还可以对分子动力学(MD)模拟的轨迹进行处理和分析。 1.载入一个分子 以1ubq.pdb在做演示,点击(a),就会出现窗口(b)点击Browse可选择本地文件,点击Load载入 图1 载入一个分子 图1 载入一个分子2.显示分子 VMD允许旋转、移动、缩放等操作,来显示分子3D结构鼠标操作:长按左键进行上下左右旋转,右键可绕中心平面内旋转,滑轮可进行缩放(见图2)在VMD Main→Mouse也可以转换操作方式(见图3) 图2 图2 图3 图33.图像的展示 3.1 分子不同的显示方式 点击VMD Main→Graphics→Representations..显示出Graphical Representations窗口(见图4)(a)展示窗口,在(b)Draw Style下,可选择Coloring Method、Drawing Method、Material,这里一部分先关注Drawing模式在Selected Atoms里输入protein,Coloring.选择Name,Drawing选择Lines,点击Apply,看下什么样(图5)?右下方有Thickness还可以改变粗细,在Drawing还可以选择VDM(van der Waals)显示,看看是什么样?右下方是否有变化?在Drawing下选择Licorice、Tube、NewCartoon试试(图6)3.2 给分子不同的着色 在Coloring→Name,看下分子怎么着色(注:主菜单下Graphics→Colors可以更改各类原子着色)选择ResType,可以为非极性、碱性、酸性等不同类氨基酸着手,看下α-螺旋构成的残基选择Secondary Structure,可以将分子以二级结构显示尝试其他着手方案看看,选择不同的Matrial(Coloring旁边),看您能发现什么? 图 4 图 4 图 5 图 5 图 6 图 6这节的内容就到此吧,下次讲展示您想看的具体细微结构和多重构象。 NOTES:1.VMD下载地址,笔者下载的1.9.3 Windows OpenGL,最好安装在全英文路径下,如果遇到要注册,按照程序注册即可; 2.参考的教程和文件在这里可以下载到; 3.网上也有中文版教程,很容易找到(但建议阅读英文版教程); 参考: VMD(Visual Molecular Dynamics)教程(2)原文: 在教程(1)中,相信大家已经学会基本的载入、不同显示方式,今天分享VMD重要功能,选择不同的内容,展示多重图像等。 1.选择不同的结构、原子、残基等 对于酶,蛋白的结构,常常只关心活性周围的结构,这时你需要只想观察一定范围的内容。 在图(1)中,进入Selections,在(b)中提供了您可以选择的关键词,在(c)里提供更加详细的选择内容,比如多少号残基可以使用布尔逻辑算符(Boolean operators)进行组合,比如想观察螺旋上的赖氨酸,应该在Selected Atoms写:helix and resname LYS,然后点击Apply,看看效果又如您想选择蛋白质中所有赖氨酸5埃内物质,输入:protein and within 5 of resname LYS(假如是选择某个特定残基应该输入什么呢?)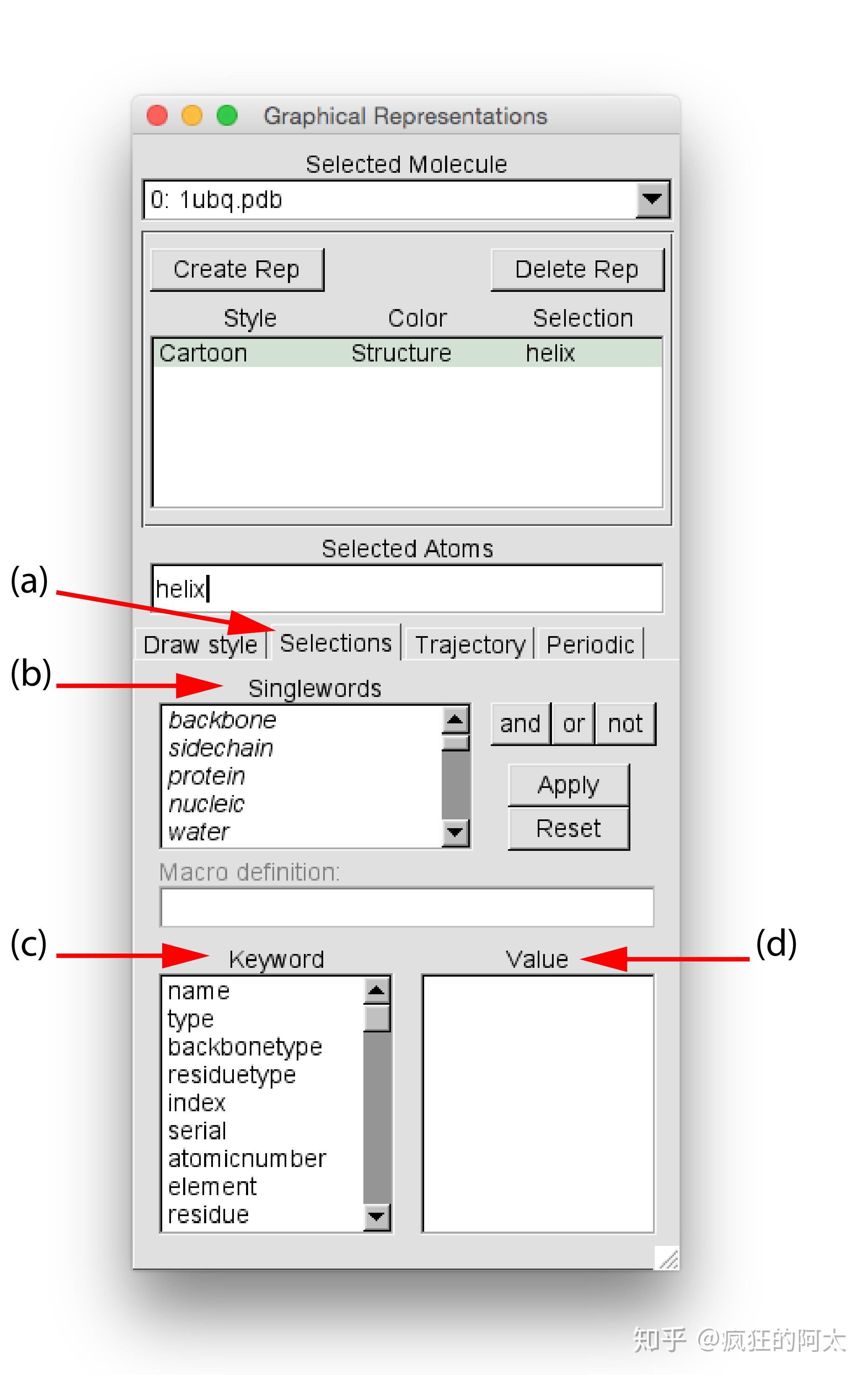 图1 选择不同内容界面 图1 选择不同内容界面2.创造多重图像 VMD可以很方便、直观的显示多重构象,方便分析不同残基间的关联性 看图(2),a,b,c分别代表添加新图像、删除图像、图像列表,根据1的方法,当您选择好内容后,点击Apply→Create Rep,即可出现想要的效果,当然,你也可以随时在Selections里修改同理,您可以创造多个图像,比如A链的组氨酸,B链的赖氨酸等;想要删除某个图像就选择它,然后Delete Rep,假如您只想隐藏,可以双击它,变红就隐藏了,想删除就选(b) 图(2) 图(2)3.用序列面板辅助选择 有时您知道活性中心的位置,而且知道具体起作用的氨基酸残基,这时,直接选择具体的氨基酸显的尤为重要。 在主菜单下,Extensions→Analysis→Sequence Viewer下可以打开(如图3)你可以在(e)中单击选择氨基酸,并且可以使用shift进行连选,右键到某个氨基酸又可以取消,其他两列B value和struct有兴趣的同学可以Google(文末有参考信息)接着打开Graphical Representations窗口,看看您选择的氨基酸在这里显示了吗?这样操作有什么好处 图(3) 图(3)4.额外的Tips 在主菜单的Mouse下,您可以选择Label→Atoms,点选具体的原子,这对确定结构结构很有用在Graphics→Colors,有很多选项改变颜色,包括背景(在Display下)假如你不想让Axes显示在屏幕中,可在Display→Axes关掉参考资料:1.https://www.ks.uiuc.edu/Research/vmd/ 2.STRIDE:http://webclu.bio.wzw.tum.de/stride/ 3.B-factor Definition in WIKIPEDIA:The Debye–Waller factor(DWF), named after Peter Debye and Ivar Waller, is used in condensed matter physics to describe the attenuation of x-ray scattering or coherent neutron scatteringcaused by thermal motion.[1][2]It has also been called the B factor or the temperature factor. Often, "Debye–Waller factor" is used as a generic term that comprises the Lamb–Mössbauer factor of incoherent neutron scattering and Mössbauer spectroscopy [1]doi:10.1002/andp.19133480105 [2]doi:10.1007/BF01328696 官方tutorial:VMD使用目录 VMD使用 打开data文件 周期性显示问题 原子选择 移动原子 导出图片 导出data文件 VMD使用 打开data文件 在Extensions/Tk Console中,先cd到data文件所在地址,键入:topo readlammpsdata filename.data 周期性显示问题 显示盒子边界: pbc box 模型in-cell显示: pbc wrap -all 取消in-cell显示: pbc unwrap -all 原子选择 选择同一类型的原子: atomselect top "type 1" 移动原子 对于选择的原子z方向整体移动10个单位: 导出图片 导出*.bmp文件:render snapshot filename.bmp 导出data文件 topo writelammpsdata filename.data 原文链接:VMD使用_vmd怎么导出图片_YuanbaoQiang的博客-CSDN博客 |VMD图像保存实用教程VMD是一款免费的分子可视化软件,通过该程序可以对GROMACS、Amber、NAMD、Lammps等程序得到的轨迹文件进行处理,实现显示大型分子系统、制作动画、分析轨迹等操作。本文将介绍如何通过VMD渲染得到高分辨率的图片。 01生成输入文件 首先,进入File-Render-Tachyon,点Start Rendering,即可在VMD的目录下生成Tachyon渲染器的输入文件vmdscene.dat。   02绘制图片 接着,在VMD安装目录下建立一个render.bat文件,用于将vmdscene.dat文件中的数据绘制成图片,具体操作如下: *render.bat文件获取方式见文末 tachyon_WIN32 vmdscene.dat -format BMP -o VMD.bmp -aasamples 24 -mediumshade -trans_vmd -res 2000 1500 -numthreads 4  点击此.dat文件运行即可得到渲染后的VMD.bmp。 *o设定输出文件的名字以及格式; *aasamples调节生成图片的锯齿化程度,越大锯齿越不明显; *res设定图像尺寸,越大图像越清晰,耗时越高; *numthreads 设定渲染时的线程数,可根据本机CPU的物理核心数进行调整。 参考链接:http://bbs.keinsci.com/thread-9178-1-1.html 以上是如何将获取的dat文件渲染出图片的教程,其实还可以通过直接将上述代码改变一下直接放入render command中,实现点击start rendering后直接渲染出图的功能。(默认的参数渲染的图真的太粗糙了) "C:\Program Files (x86)\University of Illinois\VMD\\tachyon_WIN32.exe" -aasamples 24 %s -format BMP -res 3840 2160 -mediumshade -trans_vmd -o %s.bmp -numthreads 4 这里提醒一下,那个线程数没事可以不要设置,在windows10上面测试,发现设置了不如没设置的渲染快,而且中途会卡主程序几秒。 有的同学使用上述直接渲染的方法,渲染完找不到图了,那可能是没有在设置文件名时候点击Browse..设置存放路径。此时渲染的图片会被存放到 C:\Users\你的电脑用户名\AppData\Local\VirtualStore\Program Files (x86)\University of Illinois\VMD另外附上自己常用的Linux-VMD 的render command"/usr/local/lib/vmd/tachyon_LINUXAMD64" -aasamples 24 %s -res 3840 1750 -format TARGA -o %s.tga -numthreads 6 这里你可能要根据需要改一下tachyon_LINUXAMD64的路径。 Questions?
|
【本文地址】