| 老司机带你解锁ImageJ批量处理图片 | 您所在的位置:网站首页 › imagej版本号 › 老司机带你解锁ImageJ批量处理图片 |
老司机带你解锁ImageJ批量处理图片
|
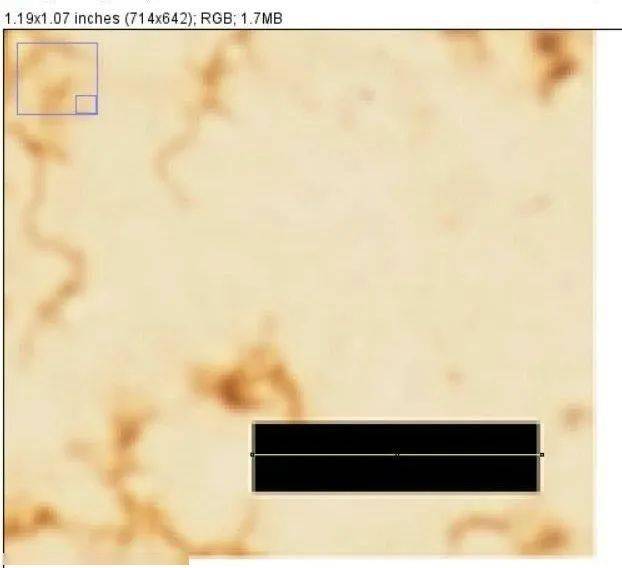
接下来给大家回顾ImageJ添加标尺的常规方法与省时高效的批量添加标尺的方法! ImageJ添加标尺的基本步骤: 1、打开一张有标尺的图片,使用 MagnifyingGlass(放大镜)工具放大标尺,使用 直线工具Shift沿着标尺画一条直线:
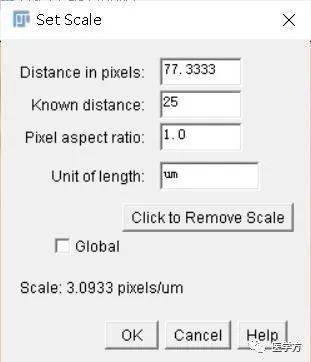
2、Analyze -> Set Scale进入标尺设置界面:
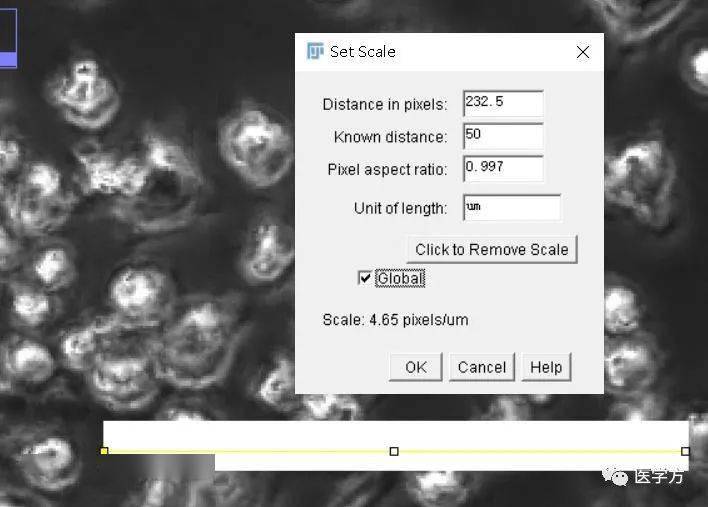
直线的距离为77.3333pixels,已知距离是25μm,Unit of length为μm,Global对所有后续打开图片有效,否则只对当前图片有效,Scale为3.0933 pixels/μm,点击OK可见图片右上角显示图片的长宽单位为μm:
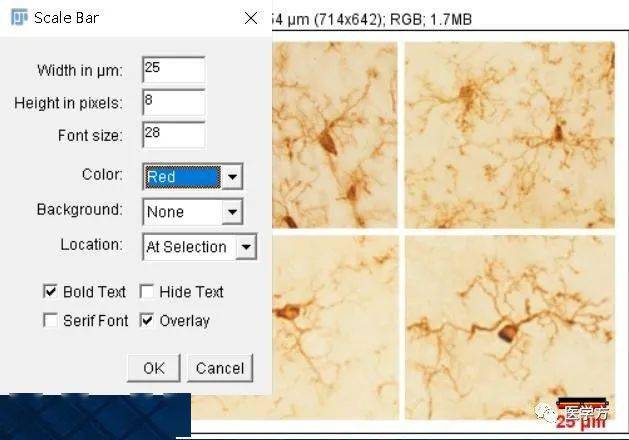
3、Analyze -> Tools ->Scale Bar给无标尺的图片添加标尺:
依次设置标尺大小、外观、字体、颜色、位置等。点击OK就能方便的添加标尺了,如上图添加了一个红色的25μm的标尺。 应用实例二 如果实验中忘记给图片添加标尺,可使用ImageJ进行批量添加, 步骤如下: 1、本次使用软件为Fiji(ImageJ的版本之一)。现有一系列无标尺的同尺寸图片,打开相同条件下获得的有标尺的图片,根据基本方法校正标尺,得到该标尺为4.65 pixels/μm:
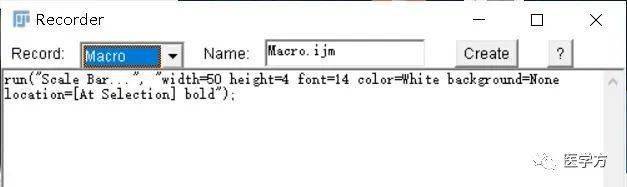
2、Plugins -> Macros -> Record…录制宏,Analyze -> Tools-> Scale Bar添加标尺,点击Create创建宏:
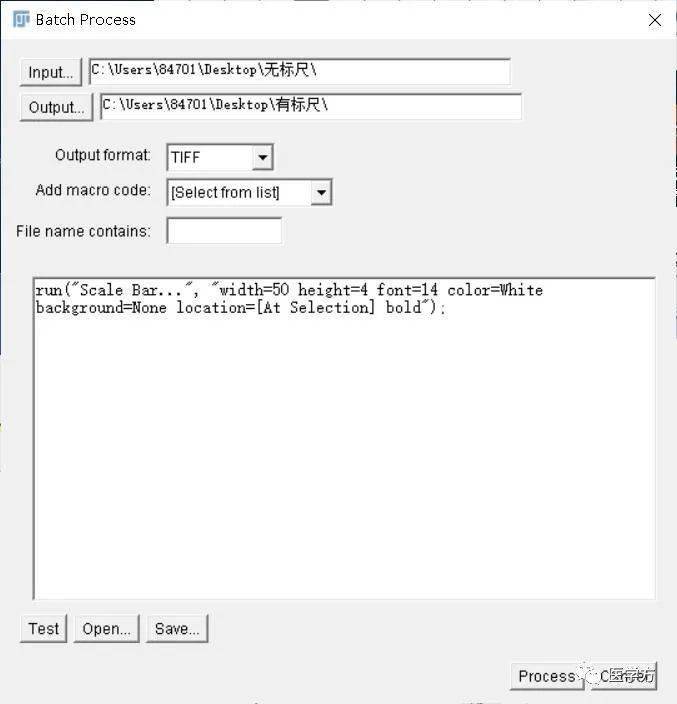
3、将一系列无标尺的图片放至无标尺文件夹,新建一个空的命名为有标尺的文件夹。Process -> Batch -> Macro进入Batch Process窗口。选择输入文件夹为无标尺文件夹,选择输出文件夹为有标尺文件夹。输出图片格式为TIFF,将录制好的宏复制粘贴至下方文本框中,点击Process进行批量添加操作:
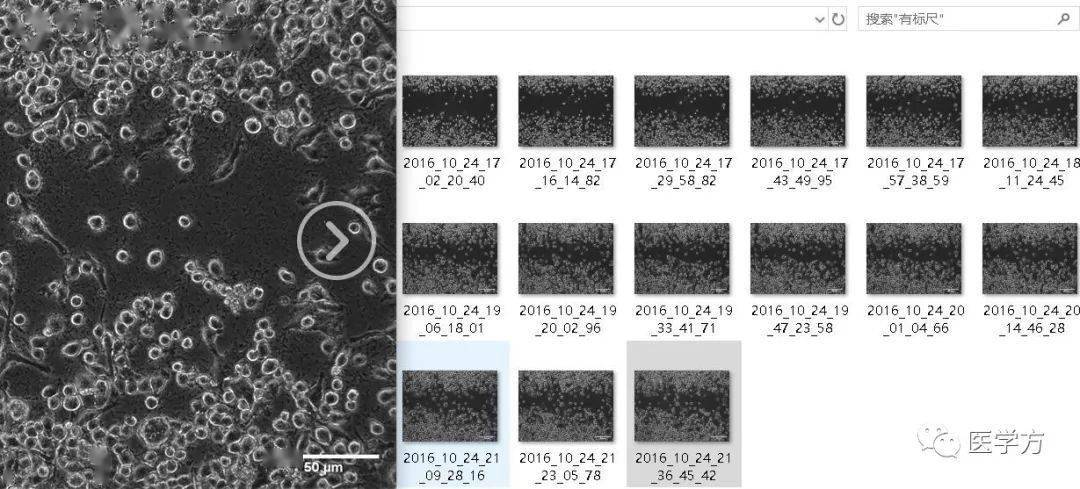
打开有标尺文件夹,可见每张图片均已添加了一个50μm的标尺:
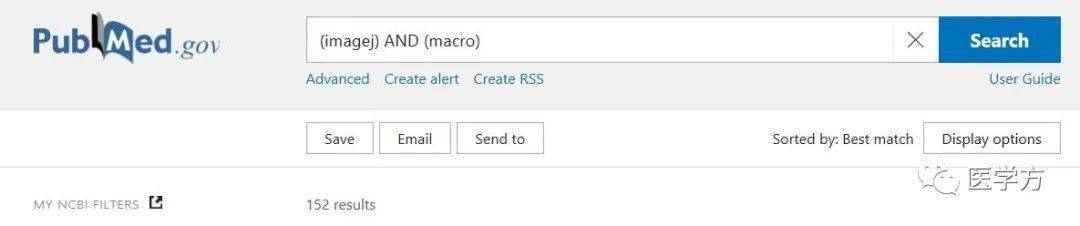
本方法使用 宏批量的给无标尺的图片添加了标尺,大大提高了效率,节省了时间。 举一反三 宏广泛应用于快速、批量的图片处理。例如在Pubmed搜索(imagej) AND (macro)可得到152篇检索结果:
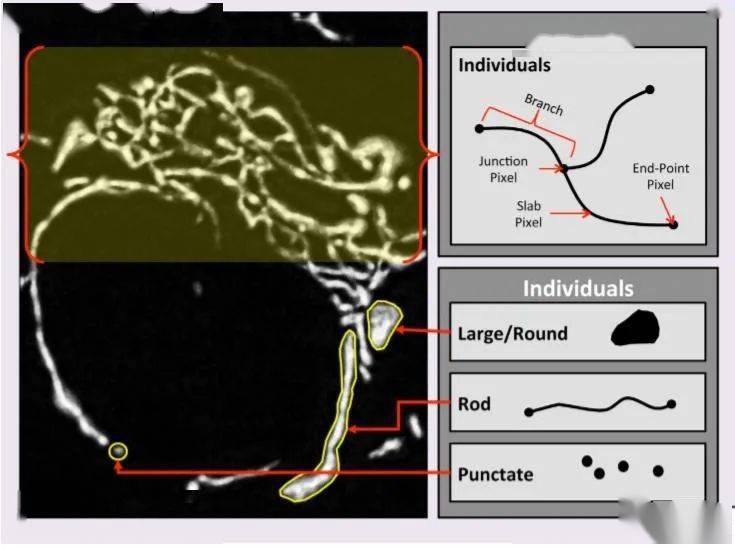
我们来看看有哪些有意思的宏。记得曾经有人留言需要分析线粒体形态的ImageJ教程,半夏曾经分享过一个名为 MiNA的宏【2】,有兴趣的小伙伴可在往期查找。
此外与检索关键词最匹配的结果是一个计算核浆比的宏,2018年5月发表在BiotechnologyJournal(最新影响因子3.912)上:
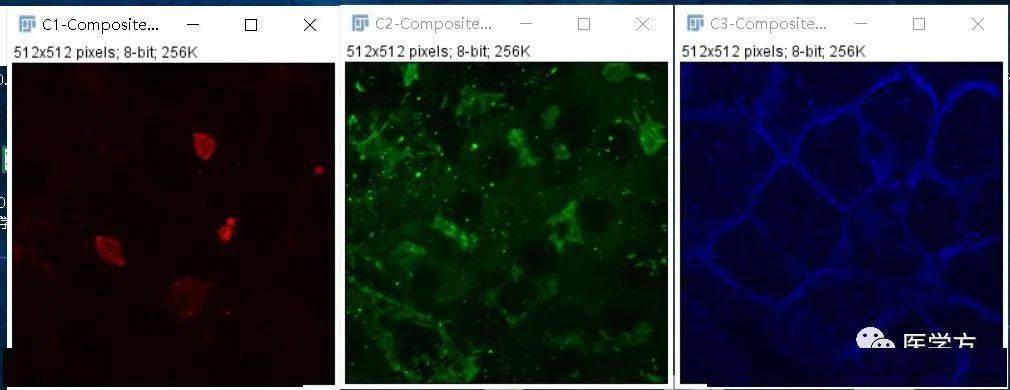
写一个实用的宏可发表SCI论文,对我们自己而言可以录制宏进行批处理提高工作效率。 1、例如批量进行R、G、B荧光通道拆分。 拆分通道基本操作为Image -> Color -> Split Channels。
后续录制宏后使用Batch Process可进行批量操作。 2、快速划痕面积分析。 在往期内容中半夏曾给大家分享过一篇名为“重磅分享:30秒搞定划痕实验面积分析!”https://mp.weixin.qq.com/s/wm5MWSNvAxllLWF45A5wjg 。该方法可以快速、批量、准确的分析划痕面积,本质上也是宏的使用。有兴趣的小伙伴可以学习学习! 学习扩展 如何使用PS添加标尺? 步骤: 1、PS打开一张带有标尺的免疫组化图片:
2、放大标尺:
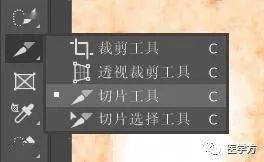
选择切片工具沿着标尺画矩形,此时矩形的宽度为标尺宽度:
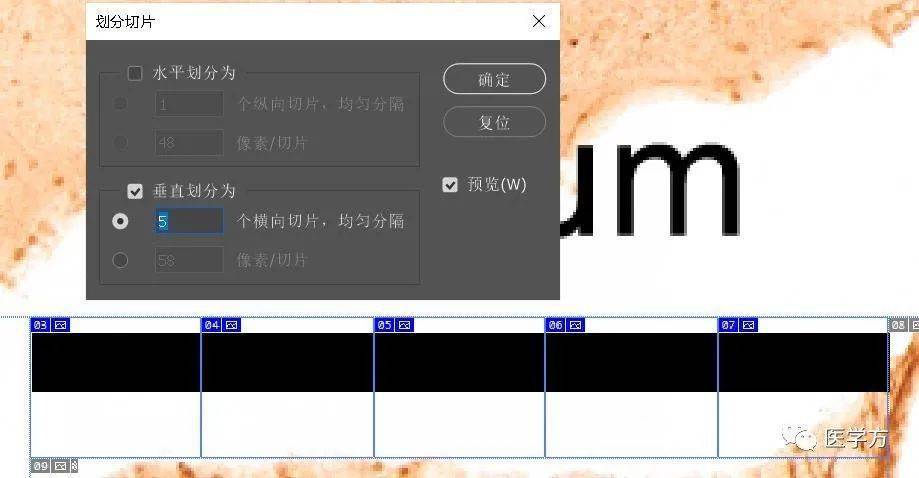
右键,划分切片,选择将垂直划分为5等分,那么每个等分即为100μm。在同样条件下的无标尺的图片根据划分的结果绘制矩形即可:
今日“屠龙刀”已在手,各位小伙伴有没有跃跃欲试的冲动。今天给大家分享ImageJ的图片批量处理就到此为止了,希望对大家有所帮助! 参考文献: ZengP, Shi Y, Wang XM, et al. Emodin Rescued Hyperhomocysteinemia-Induced Dementiaand Alzheimer's Disease-Like Features in Rats. Int J Neuropsychopharmacol.2019;22(1):57-70. doi:10.1093/ijnp/pyy090
END 征 稿 启 事 “医学方”始终致力于服务“医学人”,将最前沿、最有价值的临床、科研原创文章推送给各位临床医师、科研人员。返回搜狐,查看更多 |
【本文地址】