群体遗传学习笔记 |
您所在的位置:网站首页 › 基因组测序技术的一般步骤和方法是什么 › 群体遗传学习笔记 |
群体遗传学习笔记
|
群体遗传学习笔记-测序技术学习
重测序技术简介 全基因组重测序(Resequencing)是对已知参考基因组序列的物种进行不同个体间的基因组测序,并在此基础上对个体或群体进行差异性分析。通过全基因组重测序,将不同梯度插入片段(Insert-Size)的测序文库结合短序列(Short-Reads)、双末端(Paired-End),可以找到大量的单核苷酸多态性位点(SNP)、拷贝数变异(Copy Number Variation,CNV)、插入缺失(InDel,Insertion/Deletion)、结构变异(Structure Variation,SV) 等变异信息,应用范围涉及临床医药研究、群体遗传学研究、关联分析、进化分析等众多领域。 原理将特定组织或者细胞中的DNA进行随机打碎,构建片段为350bp或者500bp的文库,通过Illumina Hiseq对文库进行高通量测序,从而获得某一个个体所有DNA序列的信息。 全基因组数据分析的必要条件 所测物种的序列是有参考基因组的所测序个体与参考基因组之间遗传差异性不大 (read 比对不上,很难找到SNP 等突变信息) 评价测序量的指标测序深度是评价测序量的最重要指标。测序深度(Sequencing Depth):测序得到的碱基总量(bp)与基因组大小(Genome)的比值。测序覆盖比例(Sequencing Coverage),指的是基因组上至少被检测到1次的区域,占整个基因组的比例。 例如,在某1个样本测序的项目中,基因组平均测序深度为60X。基因组大部分区域的测序深度在60X左右,但同时依然有一小部分区域的测序深度低于3X(极低覆盖或没有覆盖)。 当然,这是理想条件下。在实际情况下的覆盖度,会低于理想值。主要是由于GC含量偏好,基因组完整性,个体差异,重复序列影响等。
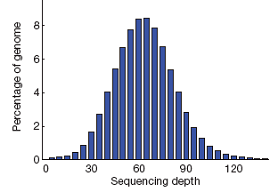
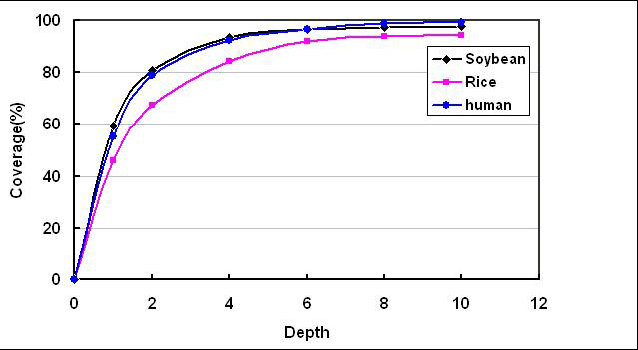
此处输入图片的描述 如下图,在某1个样本测序的项目中,基因组平均测序深度为60X。基因组大部分区域的测序深度在60X左右,但同时依然有一小部分区域的测序深度低于3X(极低覆盖或没有覆盖)。 当然,这是理想条件下。在实际情况下的覆盖度,会低于理想值。主要是由于GC含量偏好,基因组完整性,个体差异,重复序列影响等。 测序深度与基因组覆盖度之间是一个正相关的关系,测序带来的错误率或假阳性结果会随着测序深度的提升而下降。重测序的个体,如果采用的是Paired-End,当测序深度达到10x时基因组的覆盖度已接近饱和,基因组覆盖度和测序错误率控制均得以保证。
此处输入图片的描述
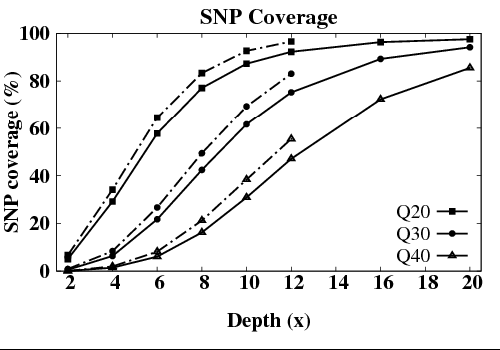
但SNP的检测率却没有达到饱和。这是由于当深度达到10X的时候,虽然基因组大部分区域已被覆盖,但在覆盖到的区域中,依然有相当多的区域深度小于34X。SNP检测的最低深度标准通常为34X。如果没有达到这个水准,则判断其不可靠,而在分析结果中不予接受。为了进一步减少低测序深度区域的比例,则需要进一步提高测序深度。只有测序深度达到30X的时候,SNP检测才会达到饱和
此处输入图片的描述
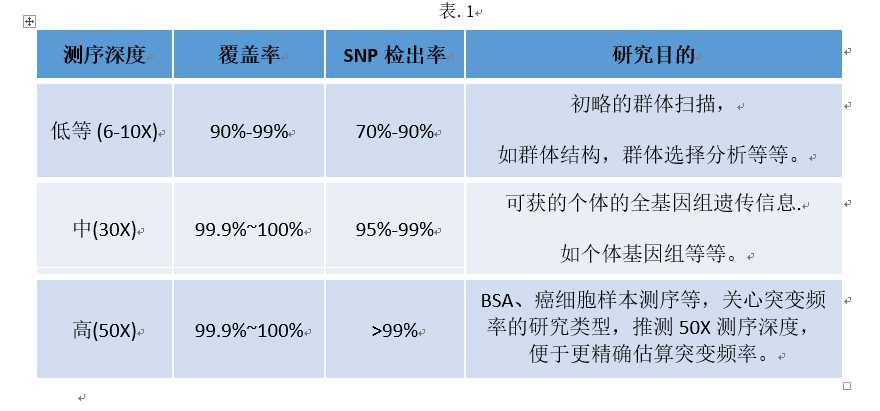
因此,可以根据我们的研究目的来选择相应的测序深度。
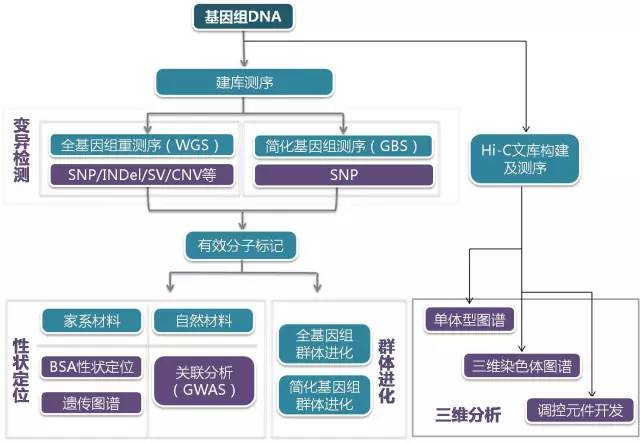
此处输入图片的描述 重测序应用目前重测序技术已广泛应用于农学、医学等各个研究领域,包括性状相关候选基因筛选、动植物育种、单基因病筛查、癌症筛查等,快速准确,对育种和临床诊断有很好的指导作用。下面举例一些重测序常用的应用范围和主要思路。
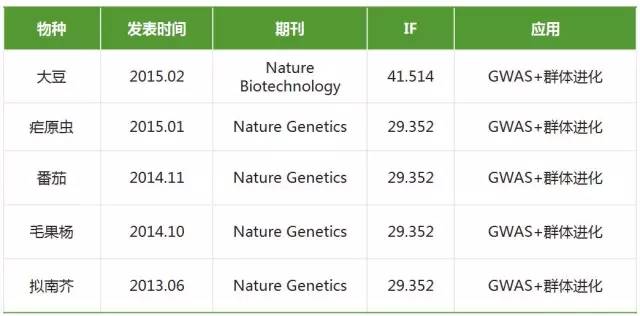
此处输入图片的描述 个体重测序,突变体检查 (对每个个体的位点进行扫描)混池重测序,群体进化分析 (通过SNP进行后续分析)BSA,遗传图谱构建Hic-主要是做人类比较有用,这里不多说了。 动植物重测序文章思路 GWAS与群体进化相结合随着GWAS统计方法的不断完善,GWAS能够适用于大部分物种,将GWAS与群体进化结合分析为性状关键基因定位提供了一个新的思路。
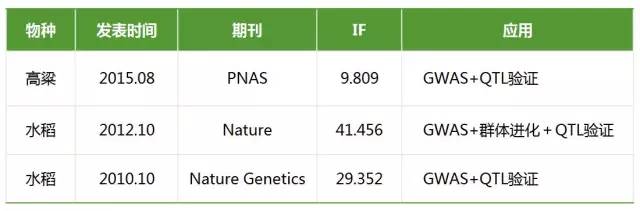
此处输入图片的描述 GWAS与QTL定位相结合连锁分析和关联分析在数量性状研究上都具有重要作用,它们在QTL定位的精度和广度、提供的信息量、统计分析方法等方面具有明显的互补性。
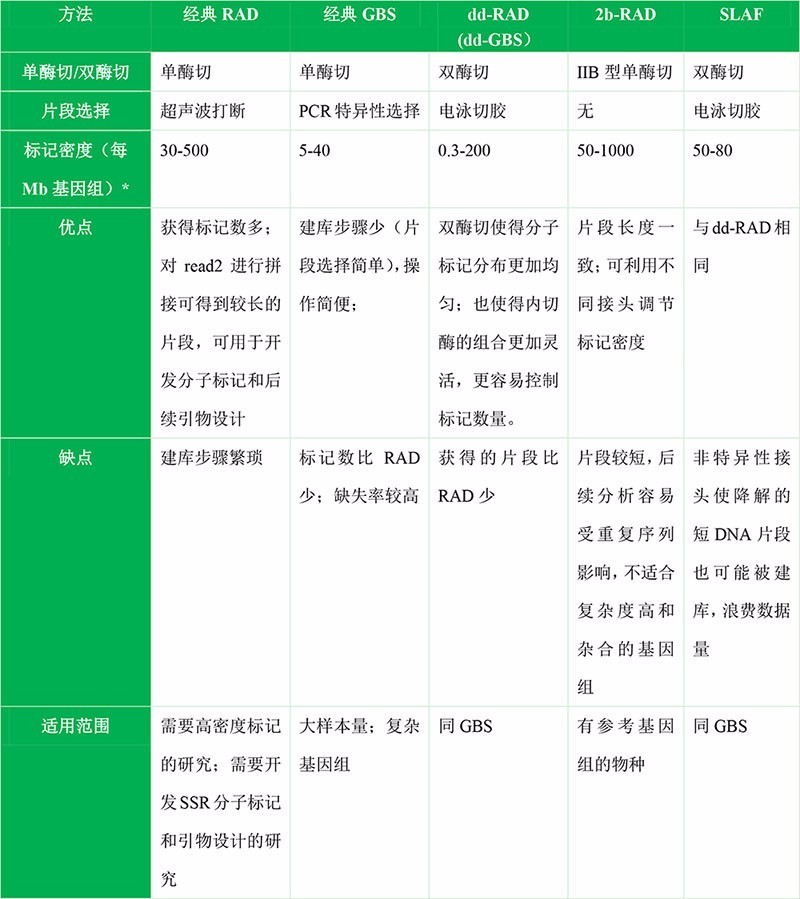
此处输入图片的描述 简化基因组测序简单来说是对与限制性核酸内切酶识别位点相关的DNA进行高通量测序。 其实那么多种简化基因组方法的区别就在于单酶切还是双酶切、是否有随机打断、使用不同的内切酶、是否加barcode、接头设计等这些细节,本质还是一样的,就是对基因组进行酶切并对酶切片段进行测序。在这些方法中,RAD和GBS是使用最广泛的两种方法,2b-RAD,dd-RAD,SLAF等都是在这些方法的基础上在不同细节处改良。 RAD(Restriction site Associated DNA):是与限制性核酸内切酶识别位点相关的DNA。RAD方法对基因组DNA进行单酶切,然后对酶切片段超声波随机打断,因此测序得到的read1是位置对齐的,而read2是参差不齐的,因此可用于denovo聚类拼接,获得较长的contig,有利于开发SSR等分子标记。 GBS(Genotyping-By-Sequencing):是指通过测序进行基因分型。2011年由Elshire, R. J.提出2。GBS方法对基因组DNA进行单酶切,不需要超声波随机打断,而是利用PCR进行片段大小选择;并且对不同的样品加上不同的barcode,可对多达96个样品进行pooling建库,简化了建库步骤,因此比RAD成本更低。现在的GBS经过改良,普遍使用双酶切了,双酶切能够得到在基因组上分布更均一的酶切片段。双酶切的GBS,有时候又被称为dd-RAD。 dd-RAD(double-digest RAD,也可以称为dd-GBS):是双酶切的RAD,并且通过切胶来进行片段选择,2012年由Brant K.提出3。其实dd-RAD已经放弃了经典RAD的超声波片段化的策略,dd-RAD的建库流程和经典的GBS更为相似,所以下文我们也将之称为dd-GBS。 dd-RAD最大的优势在于,由于使用了两种内切酶处理,最终获得片段在基因组上的分布更加均一,从而提高了数据的有效性。由于目前的GBS方法普遍使用双酶切,并且用电泳切胶来取代PCR扩增来选择片段大小,因此dd-RAD几乎等同于目前的GBS方法了 不同方法之间的比较:
简化基因组技术由于降低了基因组的复杂度、比全基因组重测序成本低,因此广泛应用于遗传图谱构建与QTL定位、群体进化分析、群体遗传分析、全基因组关联分析等研究领域。那么,这么多种方法,应该如何选择呢?概括来说可以从以下几方面考虑: 1. 所需标记数 不同的研究目的,所需的标记数量并不完全一样。通常,需要在全基因组范围内进行功能区间扫描和功能基因挖掘的研究,如全基因组关联分析和选择压力分析,就需要上万个高密度的分子标记,而系统发育关系、地理群体结构、基因流、系谱检测、连锁分析等研究的分子标记密度则不需要那么高,一般只需要几百到几千个分子标记足以完成分析。 对于基因定位的研究,不同的研究材料、作图群体,也会影响到需要的标记数目。例如利用自然群体进行全基因组关联分析,所需的标记数与物种的LD衰减距离相关,物种LD衰减得越快,所需的标记数就越多。又例如利用作图群体进行连锁作图QTL定位,所需的标记数与作图群体类型和群体大小有关。群体经历的世代越多(如RIL群体),群体越大,则重组事件越多,理论上提高标记密度可以有效提高遗传图谱的质量,所以所需的标记数越多。 因此,可先评估研究所需的标记数,再选择适合的简化基因组技术。RAD技术因为对所有酶切位点都检测,因此标记数要比GBS多,适合于需要标记密度高的研究,如选择压力分析。dd-GBS类的技术虽然收集的片段偏少,但标记分布更加均一,所以数据有效性更高;并且建库成本比RAD低,更适合大样品量的研究。 2.有无参考基因组 如果所研究物种没有参考基因组,那么RAD技术更合适,因为RAD技术可以利用不对齐的read2进行denovo拼接,再与read1拼接,可以得到长达400~500bp的片段,有利于SSR分子标记开发以及后续的引物设计。而2b-RAD由于片段过短,容易受重复序列干扰,且后期不利于设计引物验证测序得到的SNP。因此,2b-RAD不建议用在没有参考基因组的物种,和大的复杂的基因组上。 3.研究经费 简化基因组测序与全基因组重测序相比,由于只对酶切片段进行测序,因此在测序费用上大大下降。而由于目前的各种简化基因组技术都会使用barcode对多个样品进行混合建库,因此各方法间的建库成本差异已经不大。但RAD文库构建过程中有超声波打断步骤、dd-RAD需要使用Pippin Prep等仪器,因此成本还是要比GBS等高。在实际情况中,可根据具体样品数和研究经费选择合适的技术方法。 总的来说,RAD和GBS技术是使用最广泛的两种简化基因组技术,其他的技术方法都是在这两者的基础上的改进或细化。 基因分型芯片基因分型芯片:利用已知的SNP位点侧翼的序列设计探针。探针固定在芯片上后,待测定样本的DNA与芯片杂交并扫描杂交荧光信号,从而鉴定这些探针位点(SNP位点)的基因型。最有代表性的品牌是illumina和affymetrix。 与简化组测序技术相比较从以上比较,我们可以认为两种技术都是高性价比的大规模基因分型的方法。但最大的不同的是: 芯片基因分型本质上是对已知SNP多态位点的扫描,来确定样本在这个位点的基因型。(其实很多做人类医疗测序的都用的是芯片,人类的SNPs多态性信息已经比较完善了)那么,我们需要预先知道这个物种的基因组SNPs多态性信息(一般来源大规模重测序),然后筛选SNP设计芯片,才能进行后续的基因分型。打个比方:就是已经知道这个位置有个“坑”了,只是看看坑里到底是沙子还是水。所以芯片只能“分型”,不能“发现”。简化基因组测序本质上还是测序,所以哪怕这个物种没有任何已知的SNPs信息,也能使用简化基因组测序进行检测。测序兼顾了“发现”和“分型”的功能。 两个技术的适用范围1. 没有标准化芯片的非模式物种 遇到非主流的无标准化芯片的物种,测序无疑是最佳选择。兼顾了SNP的发现和基因型分型两个功能。 2. 有标准化芯片的模式物种 如果你研究的物种是人、猪、牛等这些物种,那么芯片公司的提供的芯片还是不错的选择的。毕竟这些芯片位点都是优化过的,基本是比较均匀地覆盖了相应物种的整个基因组。芯片的数据相对简单,后期数据的基本处理更简单。而测序数据,由于数据量大,后期数据的预处理复杂且需要较多计算资源。 那么,模式生物中是否抱定标准化芯片呢?也未必。主要还是两点: 1)芯片密度是否满足你的需求? 一些成熟的模式种,例如人,芯片密度都已经达到了兆级别。但对于某些农业种,芯片密度则还停留在较低的水平。例如:illumina玉米和绵羊的芯片,都停留在50k的密度水平,很久没有优化了。不过也可以理解他们的逻辑,反正芯片和测序仪都是他们家生产的。芯片密度不够?测序啊。哪怕是使用只对基因组一部分进行测序的简化基因组测序,也可以轻松获得几百k数量级的SNP标记。所以,对于芯片密度不够的情况下,测序是芯片很好的替代品。 2)对一些稀有位点的检测 由于设计芯片只使用群体中具有普遍性的多态位点,即这些位点都是在群体中高频出现的多态性位点。所以,对一般人群/种群进行普遍性的筛查的时候,没有太大的问题。但如果,我们研究的群体十分特殊(如,研究材料是比较偏的亚种),或研究目标就是低频甚至罕见位点的时候(癌症、家族遗传病),芯片就无能为力了——因为芯片上就没有这些位点的探针啊。 例如,你研究的是藏猪,那么猪的porcine 60k芯片效果就不会太好。因为芯片上的60k位点都是从常见的品种中筛查得到的,这些位点在藏猪这样的特殊亚种中可能多态性较差。而藏猪群体中普遍的多态性位点,标准化芯片上却没有。那么,这个时候简化基因组测序的效果会优于芯片。测序嘛,我测的是序列,管你SNPs稀有不稀有通通一网打尽。 总之,基因分型芯片和简化基因组测序,各有优缺点。在具体项目中,应该根据具体情况做选择。随着测序价格不断降低,测序的确会不断侵蚀芯片的市场空间。但芯片依然有其稳定、易标准化、效率高、成本容易控制等优点,在某些需要标准化的领域(例如:医疗诊断领域)有巨大的应用空间。 |
【本文地址】
今日新闻 |
点击排行 |
|
推荐新闻 |
图片新闻 |
|
专题文章 |