2 |
您所在的位置:网站首页 › 基因index › 2 |
2
|
RNA-seq分析: 通路富集分析(GSEA)
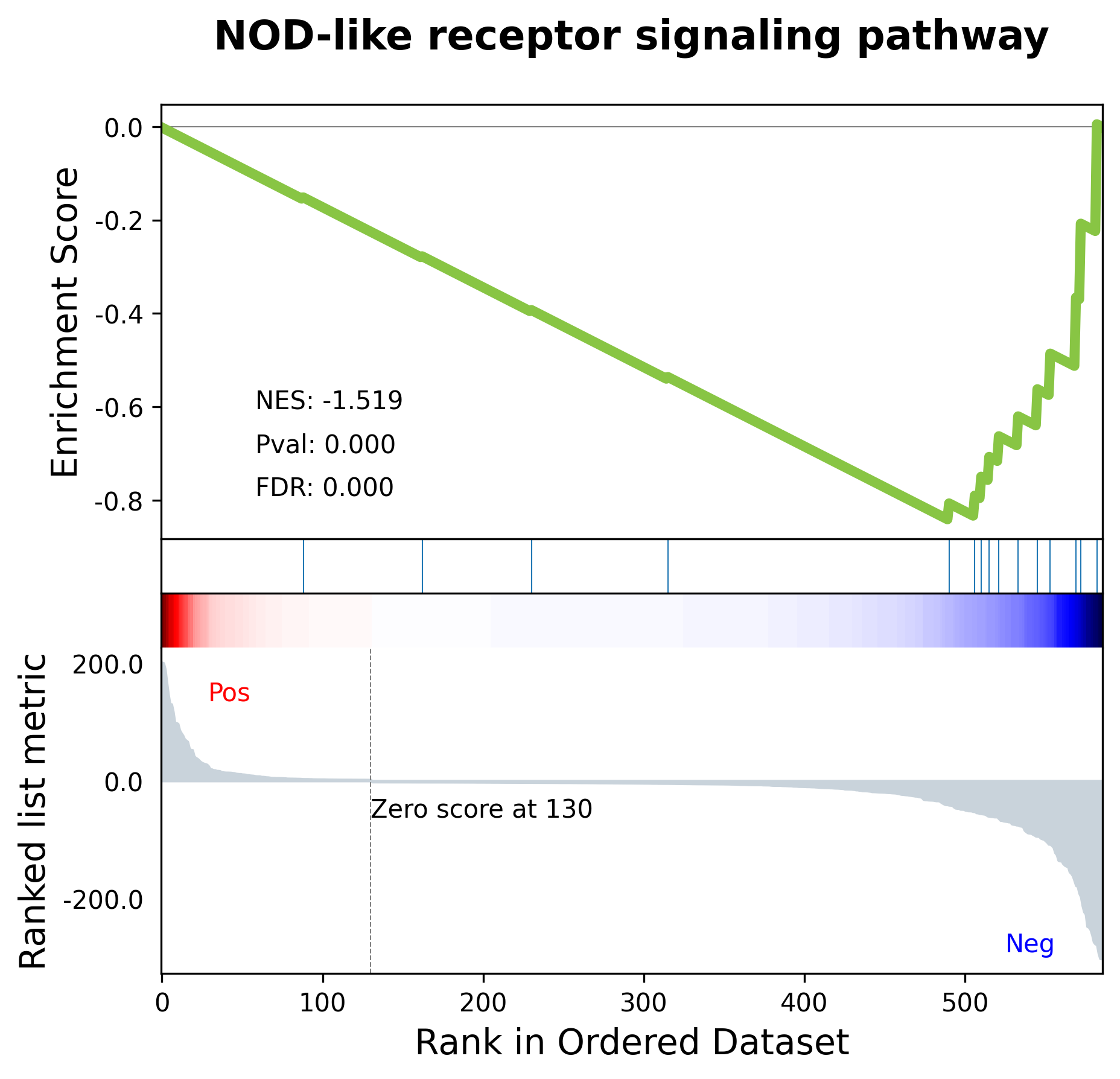
在得到差异表达分析的结果后,我们往往还会对差异表达基因相关的功能感兴趣,在这里,我们介绍GSEA方法,这是一种比较好用的通路富集方法,可以根据基因表达的倍数情况,来分析基因相关的通路。 一般来说,做GSEA分析其实是可以不用做提取差异表达基因分析的结果的,直接用DESeq2的结果就可以了,但是我这里更关注差异表达基因相关的功能,所以就用了差异表达基因来做GSEA分析 1. 数据预处理 1.1 加载包 import gseapy as gp 1.2 提取数据由于我们需要同时考虑log2FoldChange与p-value,所以我们这里用一个新的公式来计算基因倍数 metric=-sign(log2FoldChange)/-lg10(pvalue) gseada=data1.loc[data1['sig']!='normal'] #倍数变化规则 gseada['fcsign']=-np.sign(gseada['log2FoldChange']) gseada['logp']=-np.log10(gseada['pvalue']) gseada['metric']=gseada['logp']/gseada['fcsign'] gseada.head()得到结果如下 baseMean log2FoldChange lfcSE stat pvalue padj sig log(padj) fcsign logp metric ID3 5442.355360 -5.710849 0.145082 -39.362892 0.000000e+00 0.000000e+00 down inf 1.0 inf inf TXNIP 3147.115496 4.041551 0.103068 39.212444 0.000000e+00 0.000000e+00 up inf -1.0 inf -inf MMP1 4344.077177 4.476829 0.108971 41.082873 0.000000e+00 0.000000e+00 up inf -1.0 inf -inf ID1 5526.691818 -5.280627 0.112286 -47.028177 0.000000e+00 0.000000e+00 down inf 1.0 inf inf PTGIS 4187.607799 3.441545 0.092530 37.193624 8.651991e-303 3.367009e-299 up 298.472756 -1.0 302.062884 -302.062884 1.3 数据排序 gseada=gseada.sort_values(by=['metric'],ascending=False) gseada.head() baseMean log2FoldChange lfcSE stat pvalue padj sig log(padj) fcsign logp metric ID3 5442.355360 -5.710849 0.145082 -39.362892 0.000000e+00 0.000000e+00 down inf 1.0 inf inf ID1 5526.691818 -5.280627 0.112286 -47.028177 0.000000e+00 0.000000e+00 down inf 1.0 inf inf OXTR 4105.199915 -2.959533 0.100562 -29.429803 2.283138e-190 2.468072e-187 down 186.607642 1.0 189.641468 189.641468 SAMD11 1574.582498 -6.502040 0.235542 -27.604533 9.816869e-168 8.682574e-165 down 164.061352 1.0 167.008027 167.008027 1.4 rnk矩阵提取我们发现数据中有inf,这对后续的分析有一定的影响,所以我们对inf进行赋值。 rnk=pd.DataFrame() rnk['gene_name']=gseada.index rnk['rnk']=gseada['metric'].values k=1 total=0 for i in range(len(rnk)): if rnk.loc[i,'rnk']==np.inf: total+=1 #200跟274根据你的数据进行更改,保证inf比你数据最大的大,-inf比数据最小的小就好 for i in range(len(rnk)): if rnk.loc[i,'rnk']==np.inf: rnk.loc[i,'rnk']=200+(total-k) k+=1 elif rnk.loc[i,'rnk']==-np.inf: rnk.loc[i,'rnk']=-(274+k) k+=1 #rnk=rnk.replace(np.inf,300) #rnk=rnk.replace(-np.inf,-300) rnk.head() gene_name rnk 0 ID3 201.000000 1 ID1 200.000000 2 OXTR 189.641468 3 SAMD11 167.008027 4 ATOH8 146.165895 2. GSEA通路富集 2.1 基因index转大写由于gseapy的包只能识别大写的基因名,所以我们需要先把所有的基因名转换成大写的。 for i in range(len(rnk)): rnk.loc[i,'gene_name']=rnk.loc[i,'gene_name'].upper() rnk.head() 2.2 GSEA通路富集这里简单介绍以下函数的参数 rnk:输入排序矩阵 gene_sets:需要富集到的通路数据集 processes:并行使用的线程数 permutation_num:检验的速度 outdir:结果输出目录 #我们可以通过以下函数观察都有哪些数据集可被用来富集 names = gp.get_library_name() # default: Human names[:10]在这里,我们选择的是KEGG_2019_Human数据集,因为NEGF是人源的细胞系 pre_res = gp.prerank(rnk=rnk, gene_sets='KEGG_2019_Mouse', processes=4, permutation_num=100, # reduce number to speed up testing outdir='NEGF_gsea/prerank_report_kegg', format='png', seed=6)输出结果被保存到了NEGF_gsea目录下 我们可以通过index查看 pre_res.res2d.sort_index() es nes pval fdr geneset_size matched_size genes ledge_genes Term Chemokine signaling pathway -0.767073 -1.330912 0.045977 0.091794 190 16 CXCL11;CCL11;CX3CL1;HCK;CXCL10;CCL20;CCL8;VAV3... CXCL3;CXCL5;CXCL2;CXCL8;CXCL1;CXCL6 Cytokine-cytokine receptor interaction -0.702018 -1.351795 0.055556 0.094755 294 29 IL18;IL1RN;CXCL11;CCL11;CX3CL1;CXCL10;IL18R1;C... LIF;CXCL3;CXCL5;CXCL2;ACKR4;IL6;IL1B;IL32;CXCL... IL-17 signaling pathway -0.801394 -1.427337 0.000000 0.029611 93 18 CCL11;CXCL10;CCL20;MMP9;CSF2;CSF3;CCL7;CXCL3;C... CXCL3;CXCL5;CXCL2;IL6;IL1B;PTGS2;TNFAIP3;CXCL8... NOD-like receptor signaling pathway -0.840998 -1.518649 0.000000 0.000000 178 15 IL18;IRF7;NOD2;GBP4;CXCL3;CXCL2;IL6;IL1B;BIRC3... CXCL3;CXCL2;IL6;IL1B;BIRC3;OAS1;TNFAIP3;OAS2;C...Term就代表了富集到的通路 2.3 单通路可视化 from gseapy.plot import gseaplot # to save your figure, make sure that ofname is not None gseaplot(rank_metric=pre_res.ranking, term=terms[0], **pre_res.results[terms[0]])我们想观察每一个通路具体的富集情况,可以通过gseaplot绘制相关函数
我们还想观察富集到的所有通路结果,这里提供了一种气泡图的方式进行观察 sc_data=pd.read_csv(r'NEGF_gsea\prerank_report_kegg\gseapy.prerank.gene_sets.report.csv') sc_data=sc_data.loc[sc_data['pval'] |
【本文地址】
公司简介
联系我们
今日新闻 |
点击排行 |
|
推荐新闻 |
图片新闻 |
|
专题文章 |