| RNA | 您所在的位置:网站首页 › 语言表达是什么标准 › RNA |
RNA
|
学习目标
了解如何在归一化过程中列出不同的
uninteresting factors(无关因素)
了解常用的归一化方法,已经如何使用
了解如何创建
DESeqDataSet 对象及其结构
了解如何使用
DESeq2 进行归一化
1. 归一化
差异表达分析工作流程的第一步是计数归一化,这是对样本之间的基因表达进行准确比较所必需的。 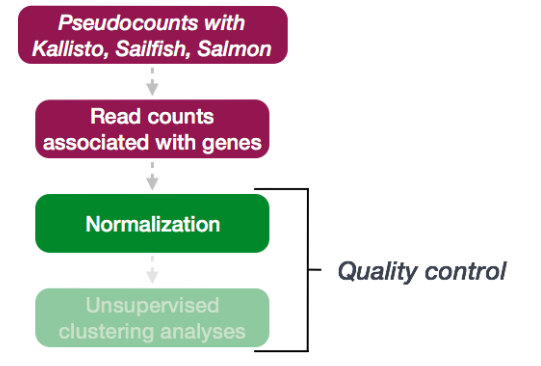 Normalization
Normalization
每个基因的映射读数计数是 RNA 表达以及许多其他因素的结果。归一化是调整原始计数值以解决“无关”因素的过程。以这种方式,表达水平在样本之间或样本内更具可比性。 在归一化过程中经常考虑的“无关”因素: 1.1. 测序深度考虑测序深度对于比较样本之间的基因表达是必要的。在下面的示例中,每个基因在样本 A 中的表达似乎是样本 B 的两倍。然而,这是样本 A 的测序深度加倍的结果。 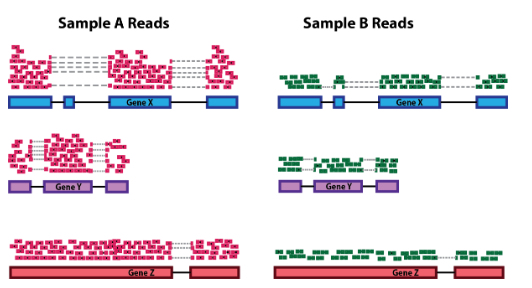 sequencing depth
1.2. 基因长度
sequencing depth
1.2. 基因长度
计算基因长度对于比较同一样本中不同基因之间的表达是必要的。在下面的示例中,基因 X 和基因 Y 具有相似的表达水平,但映射到基因 X 的读数数量将比映射到基因 Y 的读数多得多,因为基因 X 更长。 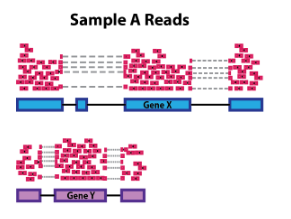 Gene length
1.3. RNA组成
Gene length
1.3. RNA组成
样本之间一些高度差异表达的基因、样本之间表达的基因数量的差异或污染的存在可能会扭曲某些类型的归一化方法。建议考虑 RNA 组成以准确比较样本之间的表达,这在进行差异表达分析时尤为重要。 在下面的示例中,假设样本 A 和样本 B 之间的测序深度相似,并且除了基因差异表达之外的每个基因在样本之间呈现相似的表达水平。样本 B 中的计数会受到 差异表达基因的极大影响,它占据了大部分计数。因此,样本 B 的其他基因的表达似乎低于样本 A 中的相同基因。 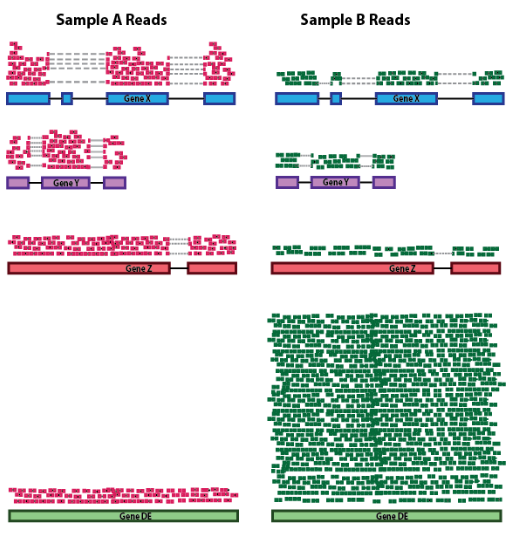 RNA composition
RNA composition
归一化不仅对于差异表达分析必不可少,对于探索数据分析、数据可视化以及探索或比较样本之间或样本内的计数也是必要的。 2. 归一化方法几种常见的归一化方法: 方法描述考虑因素使用场景CPM (counts per million)按照reads总数缩放计数测序深度同一样本组重复之间的基因计数比较;不适用于样本内比较或差异表达分析TPM (transcripts per kilobase million)每百万读取reads比对的转录本长度 (kb) 计数测序深度与基因长度样本内或同一样本组样本之间的基因计数比较;不适用于差异表达分析RPKM/FPKM (reads/fragments per kilobase of exon per million reads/fragments mapped)类似于TPM测序深度与基因长度样本中基因之间的基因计数比较;不适用于样本比较或差异表达分析DESeq2’s median of ratios计数除以特定于样本的大小因子,该因子由基因计数相对于每个基因的几何平均值的中位数比率确定测序深度和RNA组成样品之间的基因计数比较和差异表达分析;不适用于样本内比较EdgeR’s trimmed mean of M values (TMM)使用样本之间对数表达比率的加权修剪平均值测序深度和RNA组成样品之间的基因计数比较和差异表达分析;不适用于样本内比较 RPKM/FPKM:不推荐用于样本间比较虽然 TPM 和 RPKM/FPKM 归一化方法都考虑了测序深度和基因长度,但不推荐使用 RPKM/FPKM。原因是RPKM/FPKM方法输出的归一化计数值在样本之间没有可比性。 使用 RPKM/FPKM 归一化,每个样本的 RPKM/FPKM 归一化计数总数会有所不同。因此,您不能在样本之间平均比较每个基因的归一化计数。 RPKM-归一化计数表: genesampleAsampleBXCR15.55.5WASHC173.421.8………Total RPKM-normalized counts1,000,0001,500,000例如,在上表中,样本 A 的 XCR1 (5.5/1,000,000) 计数比例高于样本 B (5.5/1,500,000),即使 RPKM 计数值相同。因此,我们不能直接比较样本 A 和样本 B 之间 XCR1(或任何其他基因)的计数,因为样本之间的归一化计数总数不同。 DESeq2-归一化计数:比率方法的中值(Median of ratios method)由于用于差异表达分析的工具正在比较样本组之间相同基因的计数,因此该工具不需要考虑基因长度。然而,确实需要考虑测序深度和 RNA 组成。为了标准化测序深度和 RNA 组成,DESeq2 使用比率中值方法。在用户端只有一个步骤,但在后端涉及多个步骤,如下所述。 创建一个伪参考样本(逐行几何平均值)对于每个基因,都会创建一个伪参考样本,该样本等于所有样本的几何平均值。 genesampleAsampleBpseudo-reference sampleEF2A1489906sqrt(1489 * 906) = 1161.5ABCD12213sqrt(22 * 13) = 17.7………… 计算每个样本与参考的比率对于每个样本中的每个基因,计算比率(样本/参考)(如下所示)。由于大多数基因没有差异表达,因此每个样本中的大多数基因在样本中的比例应该相似。 genesampleAsampleBpseudo-reference sampleratio of sampleA/refratio of sampleB/refEF2A14899061161.51489/1161.5 = 1.28906/1161.5 = 0.78ABCD1221316.922/16.9 = 1.3013/16.9 = 0.77MEFV793410570.2793/570.2 = 1.39410/570.2 = 0.72BAG1764256.576/56.5 = 1.3542/56.5 = 0.74MOV105211196883.7521/883.7 = 0.5901196/883.7 = 1.35………… 计算每个样本的归一化因子(大小因子)给定样本的所有比率的中值(上表中的列)被视为该样本的归一化因子(大小因子),计算如下。请注意,差异表达的基因不应影响中值: normalization_factor_sampleA |
【本文地址】
| 今日新闻 |
| 推荐新闻 |
| 专题文章 |