| 实现影像组学全流程 | 您所在的位置:网站首页 › 影像组学radiomics库 › 实现影像组学全流程 |
实现影像组学全流程
|
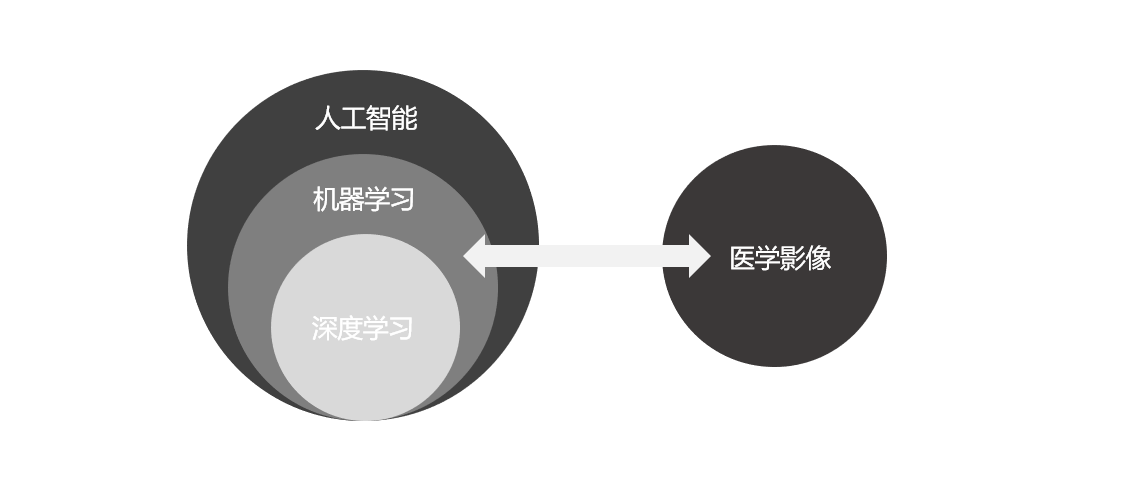
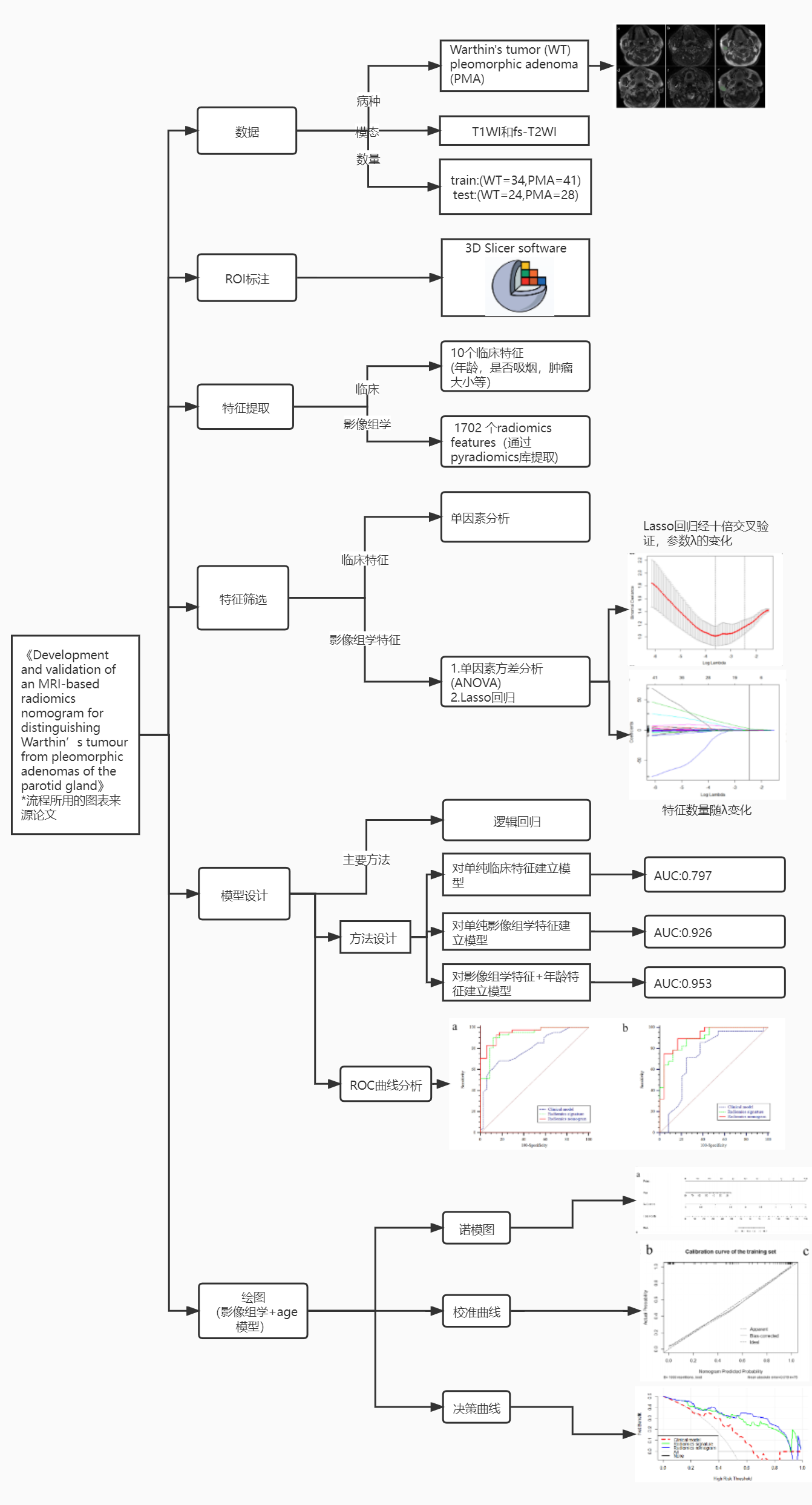
对一篇影像组学的的论文(《Development and validation of an MRI-based radiomics nomogram for distinguishing Warthin’s tumour from pleomorphic adenomas of the parotid gland》)中方法进行复现。完整地跑通影像组学全流程,对临床+影像组学特征进行建模并绘制Lasso回归图和ROC曲线、诺模图、校准曲线、决策曲线等。 需要完整代码到这里https://aistudio.baidu.com/aistudio/projectdetail/3270880 fork项目之后下载glioma.7z压缩包,然后用RStudio的RMarkdown进行运行 1.项目介绍2012年荷兰学者提出影像组学(radiomics)概念:借助计算机,从医学影像中挖掘海量定量影像特征,使用统计学/机器学习方法,筛选出最有价值的影像学特征,用来解析临床信息。 可以说影像组学是人工智能在医学领域的一种特定研究方式。
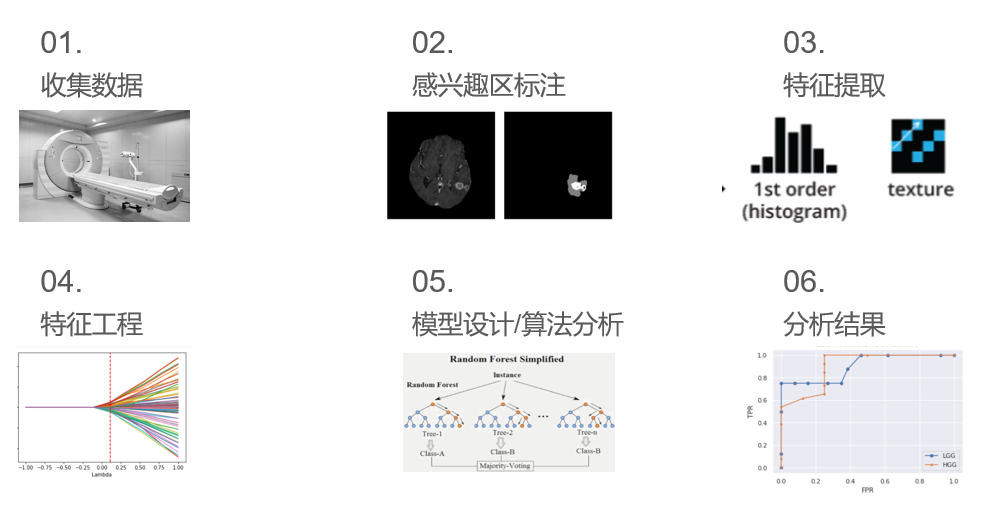
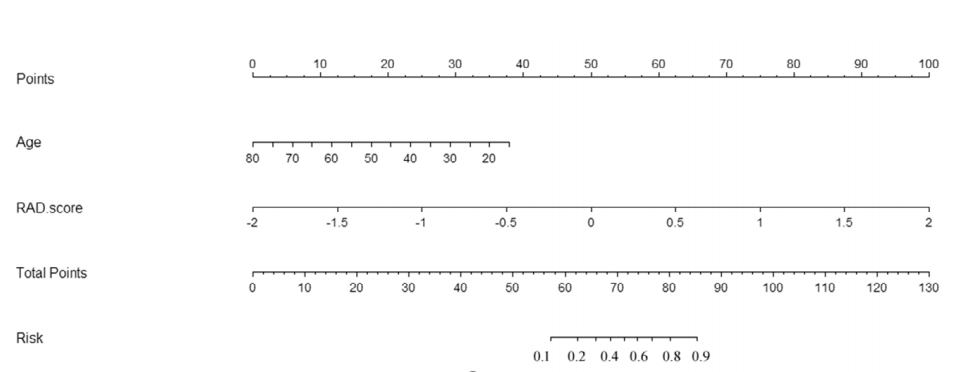
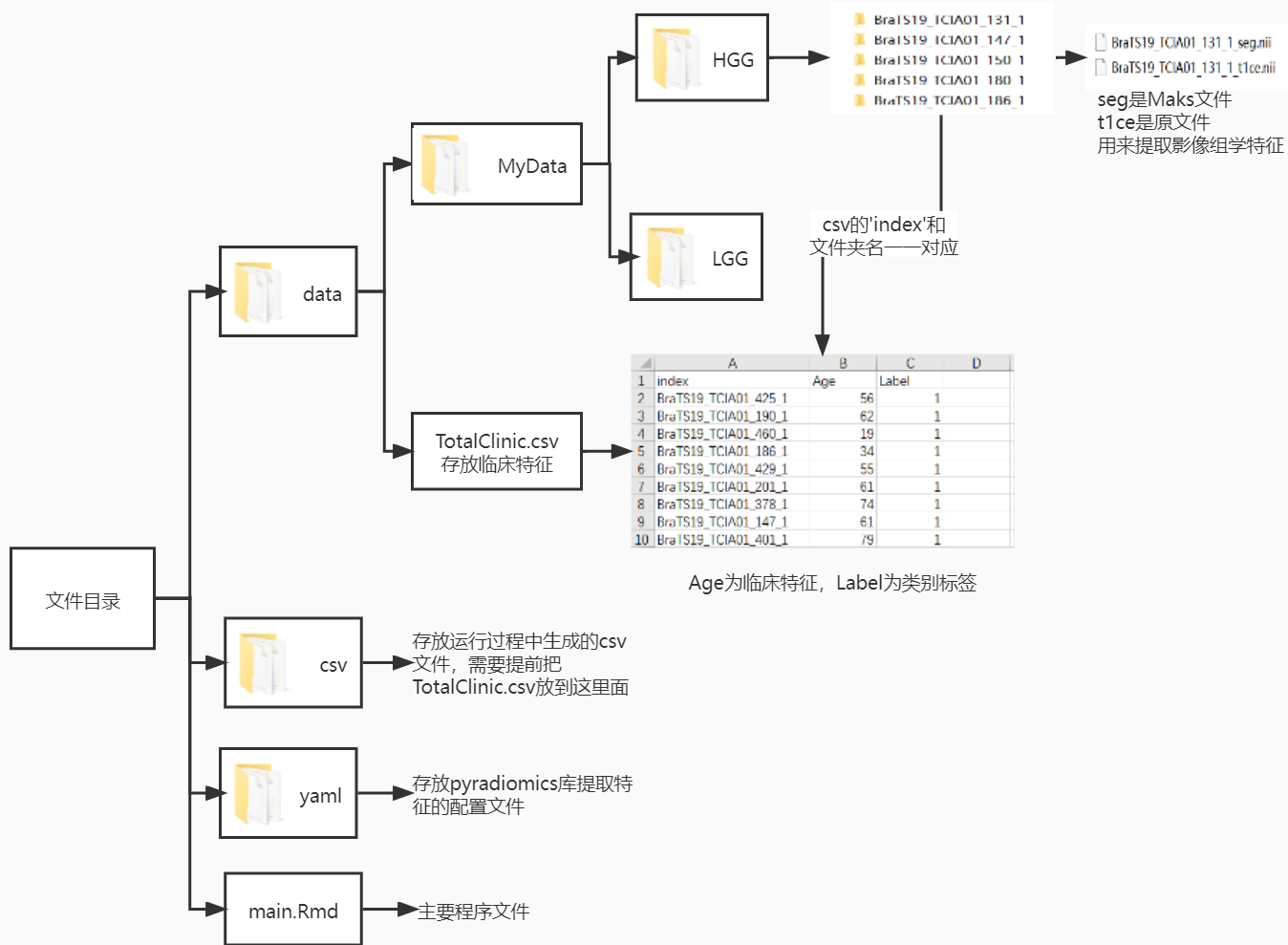
做影像组学一般会经过以下步骤1.收集数据,2.标注数据,3.特征提取,4.特征工程,5.模型设计,6.结果分析和绘图。 而对于大多数医学专业的朋友来说,毕竟学医的对写代码不一定擅长,或者第一次接触影像组学。因此想打算写一个影像组学相关的项目,对某篇组学论文方法进行复现。通用这个项目的代码希望可以稍微帮助到他们,可以让他们把时间更多花在疾病的分析上,而不是解决某个bug花大量的时间。 1.2 项目大概情况这个项目主要是对《Development and validation of an MRI-based radiomics nomogram for distinguishing Warthin’s tumour from pleomorphic adenomas of the parotid gland》这篇影像组学论文中的方法进行复现,完整的跑一个影像组学流程。包括: 1.对临床特征进行建模 2.提取影像组学特征,通过LassoCv进行特征筛选再建模 3.结合影像组学特征+age临床特征进行建模 4.绘制三个模型的ROC曲线进行对比 5.对组合模型绘制诺模图+决策曲线+校准曲线 1.3运行环境由于有些图例,例如诺模图(列线图)需要用到R语言来绘制,但是提取特征用到python语言的Pyradiomics库,所以这个项目是一个混合编程语言的项目。刚好RMarkdown可以同时运行Python和R语言,所以请有需要运行项目的请离线安装RMarkdown来运行。 虽然不能通过在线BML运行项目,但是可以展示复现论文方法的代码,主要可以容易复制粘贴代码~~~ 。 注:对于如何安装python和安装R语言和如何使用RMarkdown不是这篇项目的范围,自己通过百度自学学习。 注:这个项目也是我在学习影像组学中做下的笔记,如有错误请纠正和谅解 2.关于数据虽然是论文复现,但是论文中涉及的腮腺MR数据是不公开的,无法和论文实验数据一致,不过这个项目主要是为了实现论文中大部分方法。因此采用了一个公开分割比赛的数据集作为这个项目的数据。 在数据上用到的是一个公开的胶质瘤数据集–【BraTS2019】。是一个两分类的数据集,Hgg是高级别胶质瘤,Lgg是低级别的胶质瘤,数量上分别是240和76。每个病例有四个模态的磁共振图像。Hgg患者是带有age的年龄数据,但是lgg的患者是没有的。为了不影响临床+影像组学的建模。在(25到55)之间随机生成一个随机数赋予个Lgg患者作为他的年龄。所以Lgg患者的年龄是虚拟数据。提取影像组学的特征只用到T1增强的模态。 因为原始的数据集Hgg和Lgg的比例差不多达到3比1,所以为了搭到数据均衡一点,把Hgg删掉一部分,剩下101例。Lgg为76例。 因此在实际运用到自己的数据的时候,收集病例尽量做到数据均衡一点。不然有点自己坑自己的感觉。 论文地址 3.2论文大概内容这篇论文主要对腮腺肿瘤(Warthin和多形性腺瘤)的磁共振图像使用计算机提取大量高通量的特征,通过对特征进行筛选和建立模型来鉴别MR图像是Warthin还是多形性腺瘤。 两种腺瘤的治疗方法和预后不同,建立一个良好的分类模型对临床来说至关重要。论文通过建立三种不同特征的模型,分别是1.单纯临床特征模型,2.单纯影像组学特征模型,3.影像组学特征+年龄模型。经过实验得到模型3的auc最好。并根据模型3构建影像组学列线图(如下图),使预测模型的结果更具有可读性,让模型更加方便运用实际临床决策之中。
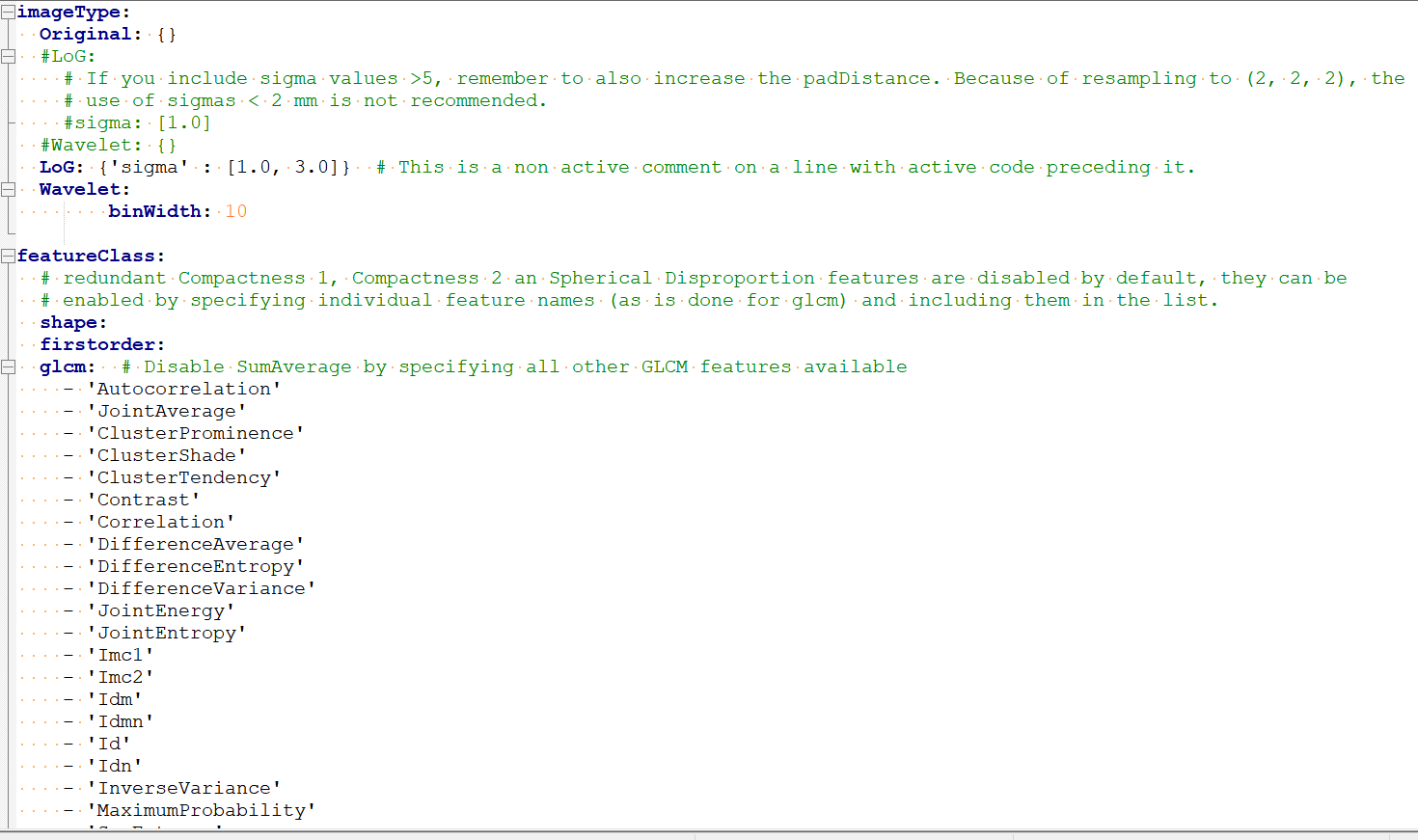
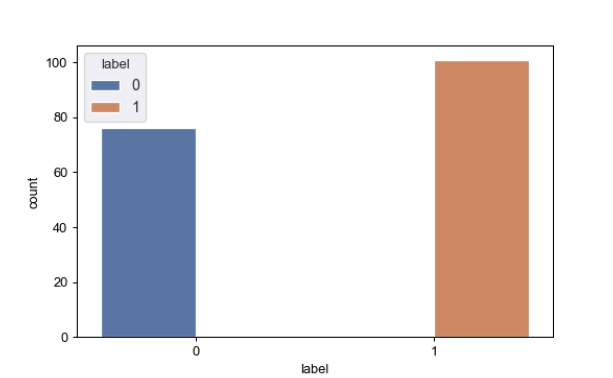
重要的一点就是临床特征的index列需要和每一例病例文件夹要一一对应 特征提取是采用pyradiomics库进行提取,可以通过配置文件来设置提取那些特征,例如设置提取特征前是否重采样,设置滤波核的大小等等。如下图 可以到pyradiomics官网查看地址 """ 经过一两个小时提取后会生成HGG.csv和LGG.csv文件, 生成的csv文件每一行都有接近一千个特征,数量会根据不同yaml文件设置不同而不同 """ #导入相关的库 import sys import pandas as pd import os import random import shutil import numpy as np import radiomics from radiomics import featureextractor import SimpleITK as sitk kinds = ['HGG','LGG'] #这个是特征处理配置文件,具体可以参考pyradiomics官网 para_path = 'yaml/MR_1mm.yaml' extractor = featureextractor.RadiomicsFeatureExtractor(para_path) dir = 'data/MyData/' for kind in kinds: print("{}:开始提取特征".format(kind)) features_dict = dict() df = pd.DataFrame() path = dir + kind # 使用配置文件初始化特征抽取器 for index, folder in enumerate( os.listdir(path)): for f in os.listdir(os.path.join(path, folder)): if 't1ce' in f: ori_path = os.path.join(path,folder, f) break lab_path = ori_path.replace('t1ce','seg') features = extractor.execute(ori_path,lab_path) #抽取特征 #新增一列用来保存病例文件夹名字 features_dict['index'] = folder for key, value in features.items(): #输出特征 features_dict[key] = value df = df.append(pd.DataFrame.from_dict(features_dict.values()).T,ignore_index=True) print(index) df.columns = features_dict.keys() df.to_csv('csv/' +'{}.csv'.format(kind),index=0) print('Done') print("完成") """ 再对HGG.csv和LGG.csv文件进行处理,去掉字符串特征,插入label标签。 HGG标签为1,LGG标签为0 """ import matplotlib.pyplot as plt import seaborn as sns hgg_data = pd.read_csv('csv/HGG.csv') lgg_data = pd.read_csv('csv/LGG.csv') hgg_data.insert(1,'label', 1) #插入标签 lgg_data.insert(1,'label', 0) #插入标签 #因为有些特征是字符串,直接删掉 cols=[x for i,x in enumerate(hgg_data.columns) if type(hgg_data.iat[1,i]) == str] cols.remove('index') hgg_data=hgg_data.drop(cols,axis=1) cols=[x for i,x in enumerate(lgg_data.columns) if type(lgg_data.iat[1,i]) == str] cols.remove('index') lgg_data=lgg_data.drop(cols,axis=1) #再合并成一个新的csv文件。 total_data = pd.concat([hgg_data, lgg_data]) total_data.to_csv('csv/TotalOMICS.csv',index=False) #简单查看数据的分布 fig, ax = plt.subplots() sns.set() ax = sns.countplot(x='label',hue='label',data=total_data) plt.show() print(total_data['label'].value_counts())
对影像组学数据集进行划分训练集合测试集。比例为8:2。同样把临床特征也进行同样的划分(顺序和影响组学特征是一致的)。 #导入常用R包 library(glmnet) library(rms) library(foreign) library(ggplot2) library(pROC) #设置种子为了保证每次结果都一样 set.seed(888) data if(tData_test2[tData_test2$index == i,]$index == i){ Age |
【本文地址】