| 转录因子详细介绍(motif) | 您所在的位置:网站首页 › 因子和质因子的关系 › 转录因子详细介绍(motif) |
转录因子详细介绍(motif)
|
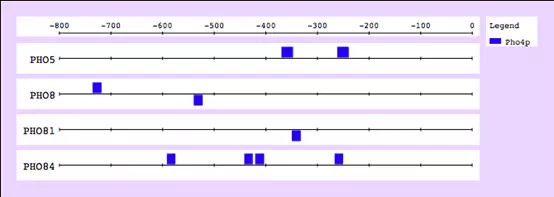
转录因子详细介绍(motif) TF: transcription factor转录因子TFBS: transcription factor binding site转录因子结合位点TFBS是序列内的location,TF特异结合在这里,这个site有这种特点 1 和一些参考相关的一个位置(开始,结束,strand),这些reference可以是染色体开始,geneTSS。也可以是一段sequence 2 A SITE可以是实验证实的(已知的),也可以是一些算法(预测的) 3 例子,下面这个图是酵母TF Pho4p的结合位点。坐标和起始密码相关。
从结合位点到结合motif binding motifs 定义:transcription factor binding site(TFBS)TFBS指的是TF特异结合的DNA分子的position(位置),延伸的话,就是DNA片段的序列边界。 注意,文献中经常把结合位点和结合域混淆(binding site ,binding motif) 我们推荐保留“site”这个词来定义某一特别sequence(基因的或artificial),这是一个factor结合的地方。而“motif”这个词进行结合特异性的genetic(遗传学的)描述,通过汇总一系列sites的信息进行汇总得到。 定义:TFBM (transcription factor binding motif) 转录因子结合域 “代表一个TF的结合特异性,通常通过汇总一系列结合位点的保守和可变位点而来,几个modes或representation可以用来描述TFBM:一致性,位点特异得分方阵,Hidden Markov Models(HMM)”。 1 我们使用术语”motif”或“pattern”在模型的意义上代表一个TF结合位点的特异性。 2 一个motif通常从一系列转录结合位点汇总产生 3 一个motif可以使用不同的形式描述3.1 一致性string(consensus string)A:核苷酸序列(nucleotide alphabet CACTTGGG)B:IUPAC alphabet CACGTGKKC:regular expression(规律表达) CACGTG[GT][GT]3.2 位置特异性得分矩阵(position-specific scoring matrix)(PSSM)3.3 Logo representation(Schneider,1986)3.4 Hidden Markov Models(HMM) Binding specificity结合特异性 1 Pho4p的结合特异性已经很好的被描述过了 2 High-affinity高亲和位点有核心CACGTG,后面跟着几个Gs或Cs 3 Medium-affinity中度亲和位点有核心的CACGTT,跟着几个Ts 4 一些单核苷酸突变足以阻止转录因子与其结合
Consensus reprentation 第一,酵母TF Pho4p在TRANSFAC数据库中包含8个结合位点 其中,5/8包含高亲和力结合位点(CACGTG) 3/8包含中度亲和力结合位点(CACGTT) 第二,IUPAC 模糊的核苷酸密码允许代表可变碱基 第三,15字母代表任何可能的结合在4个核苷酸之间(2-1=15) 第四,这种表示对残基的相对重要性提供了一个poor idea。
Building a position-specific scoring matrix from a collection of sites
TF Pho4p结合位点的排列(TRANSFAC注释)
Characteristics of yeast regulatory regions 在酵母中 第一,顺式作用元件(cis-regulatory elements)位于调控gene非编码区上游 第二,链敏感性strand-insensitive:活性不依赖于strand 第三,从起始密码子开始算,位于其~800bp以内:活性不依赖于精确位置 Cis-regulator modules(CRM)
1在后生动物中,一些非编码区域(典型的100-200bp)包含清晰的TF紧密压缩的结合位点. 2 这些区域称之为cis-regulatory regions(CRMs)顺式调控区域。 3 CRMs起着整合devices(设备)的作用 4 依赖于细胞中TF的结合,他们会激活或抑制靶基因的表达。(激活-增强子,抑制-沉默子) Cis-regulatory elements and their organization The localization of cis-regulatory regions varies depending on the type of organism
2.motif或pattern匹配 Pattern matching
Pattern matching in a small set of sequences 目标:知道motif,在感兴趣的序列中发现匹配的位置为每个位置指定一个得分 第一,显示匹配的质量 1 String-based 模式匹配替代 2 基于矩阵的模式匹配的权重得分 第二,为每个模式显示先验的重要性 例如从模式发现的重要性 Expected mathes for a consensus in whole genomes 从基因组范围模式匹配的期望值 Assuming a perfectly conserved hexanucleotide, with strand-insensitive activity Expected matching rate:1 occ/2kb
Genome-scale pattern matching 目标:给定一个模式,在整个基因组范围内发现匹配。也就是鉴定一个给定的TF调控的基因。 通常来说,基于单个信号的搜索会返回很多假阳性改进:第一,搜索重复信号(例如GATA框) 第二,搜索信号的结合 第三,限定位置 第四,结合编码区信息。 String-based pattern matching Word counting-Occurrences or matching sequences 如果一个序列包含一个给定motif的大量的出现,那么可以对他们所有进行得分或只计算每个序列的第一个出现,这种情况下,每一个序列都被记录为匹配motif或不匹配。
Treatment of self-overlap 对这样的words可以这样计数 每一个只计算更新发生(下面的例子是两次,意思不回头)或者所有的出现都算(2个renewing,2个重叠下面的例子) 计算模式的选择强烈影响后续的统计(依赖不依赖)
(renewing occurrences就是过去就过去了,重新开始计算Overlapping occurrences是不管怎么,只要首字母可以查到我想要的,就一直重复算) 3 Single or double starand count 1 DNA序列的特殊性是它的双链结构。 2 Words可以单链计算也可以双链,这依赖于期待的生物学信号的本质。 A:在RNA序列中,单链计算普遍合适 B:DNA序列中,对顺式作用元件来说,双链计数都可以,因为很多转录因子作用不依赖于方向定位。
Symmetrics in DNA sequences 回文序列:相对于中间的一个字母是对阵的,正读倒读都一样。 下面这个序列含有文字回文序列
但是,相应的DNA分子没有对称性
RSAT tool: dna-pattern 在匹配DNA序列上,尤其特定的模式匹配程序 1 支持部分特定核苷酸的IUPAC代码(例如TSWNATTK) 2 支持模式内固定或可变长度的空格例如GGGWn{0,30}WCCC 3 单链或双链 4 允许替代但不允许插入或删除 提取匹配的邻居(侧翼碱基) 返回(匹配位置,每个序列的匹配计数) 滑动窗口 检测包含多个模式组合的区域 具体的权重可以与每种模式相关联 Matching simple patterns 一个简单的字符串匹配模式通常信息量不足 第一,虚假匹配随处可见 第二,一致性的出现不是总依偎着TF结合 第三,一些motif比其他的有更高的重要性,比如一致性序列的核心区域
Matching a collection of overlapping patterns 模式匹配的结果可以通过匹配相互重叠的模式(单词或间隔二元组)的集合进行提高 可以使用多种模式来表示较大的结合位点的片段,或者可以使用多种模式 由共识退化引起的变种。 可以将特定的权重分配给集合的元素,以表示它们对绑定的相对重要性
Genome-scale pattern matching 知道给定的TF的一致性结合位点,一个人可以试图使用这个信息来预测整个基因组中的靶基因。 这个方法非常不准确,因为 第一,这个一致性很难反应结合的特异性 第二,结合和调控不是同义词 做一个实验,我们通过TRANSFAC和我们自己的注释来计数不同的酵母TF的一致性发生数目,针对下面中的每一个 第一,序列上游800bp,剪切来防止上游的ORFS 第二,对每一个gene,我们计算每个一致序列的出现数目。 Matrix-based pattern matching Regulatory motif:position-specific scoring matrix(PSSM) Binding motif of the yeast TF Pho4p(TRANSFAC matrix F$PHO4_01
Frequency matrix
Pseudo-count correction
Probability of a sequence segment under the matrix model
Probability of a sequence segment under the matrix model
Probability of the highest scoring sequence segment
Motif和domain的区别 Protein domain: 结构域的概念由Wetlaufer于1973年首次提出,他定义结构域为可以自动折叠的稳定的蛋白质结构单位。过去,结构域被描述为,折叠单位,致密结构单位,功能和进化单位。 每个定义都是有效的并且经常重叠。紧密结构单位结构域在很多不同的蛋白质中被发现, 它在结构环境内容易独立折叠。自然界经常把几个domains结合在一起形成多结构域和多功能蛋白质。在一个多结构域蛋白质中,每一个结构域可以独立行使它自己的功能,或者和它的临近蛋白协调一致的方式行驶。Domains既可以作为模块构建大的复合体像病毒颗粒或肌纤维,也可以提供特定的催化或结合位点,这些都在酶或调节蛋白中被发现。
Motif和domain的区别 完全不同的两个概念,但有时还有联系。 Motif:在生物学中是一个基于数据的数学统计模型,典型的是一段sequence也可以是一个结构,是特定的group的序列预测,例如一个DNA sequence可以定义为转录因子结合位点,也就是序列倾向于被这种factor结合。对蛋白质来说,sequence motifs可以被定义为蛋白质(蛋白质序列)属于一个给定的蛋白质家族。一个简单的motif可以是,例如,一个模式pattern,而这个模式被这个group中的所有成员共享。例如WTRXEKXXY(这里,X代表任何氨基酸)。当然也有更复杂的motif模型。Motif有时和特定的功能联系一起。
那,motif和domains之间有什么联系?当你考虑蛋白质家族的时候,不仅要看整个序列,还有关注单独结构域。因为,它们是一个基本的功能结构单位,因此找到单个结构域domain的序列motif是很有意义的。因此,你经常会发现一个蛋白质包含多个结构域,每个结构域都有一个与它所属的家族motif匹配的序列。
最主要的区别是,domain是独立的稳定的,motif不是。 |
【本文地址】




 (generated with Web Logo http://weblogo.berkeley.edu/logo.cgi)
(generated with Web Logo http://weblogo.berkeley.edu/logo.cgi)


 PAZAR http://www.pazar.info/ Unification of independent collection of transcription factor binding sites and motifs. YeasTract http://www.yeastract.com/ Yeast-specific database. Factors, binding sites and motifs + tools. FlyReg http://www.flyreg.org/ Drosophila DNase I Footprint Database PlantCARE http://bioinformatics.psb.ugent.be/webtools/plantcare/html/ Plant Cis-Acting Regulatory Elements
PAZAR http://www.pazar.info/ Unification of independent collection of transcription factor binding sites and motifs. YeasTract http://www.yeastract.com/ Yeast-specific database. Factors, binding sites and motifs + tools. FlyReg http://www.flyreg.org/ Drosophila DNase I Footprint Database PlantCARE http://bioinformatics.psb.ugent.be/webtools/plantcare/html/ Plant Cis-Acting Regulatory Elements






 下面这个序列不含有文字的回文序列
下面这个序列不含有文字的回文序列









 Protein domains:是一种结构实体,通常代表蛋白质结构中独立折叠和行驶功能的一部分。因此,蛋白质经常是这些结构域的不同的组合构建起来的。
Protein domains:是一种结构实体,通常代表蛋白质结构中独立折叠和行驶功能的一部分。因此,蛋白质经常是这些结构域的不同的组合构建起来的。
