| 单细胞转录组 | 您所在的位置:网站首页 › 删除harmony安装包 › 单细胞转录组 |
单细胞转录组
|
前言 上期推文单细胞转录组 | 多样本处理与锚定法整合介绍了使用锚定法进行多个样本整合,本期我们来介绍另一个多样本整合的主流方法:Harmony。 本文框架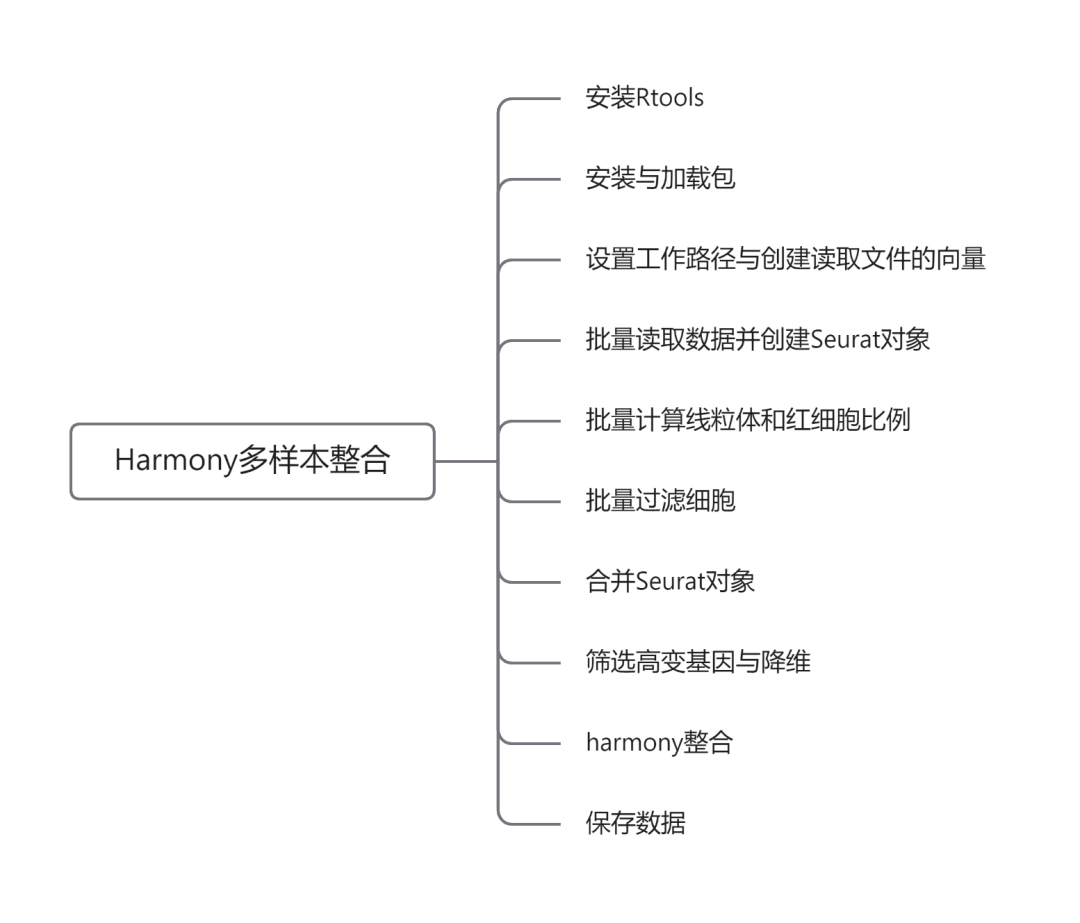 1. Rtools安装 1. Rtools安装使用harmony需要安装Rtools,如果不安装后续分析会报错。若已经安装此步请跳过。 ① 打开网站选择红色箭头指向的"RTools 4.0",进入详情页; https://cran.r-project.org/bin/windows/Rtools/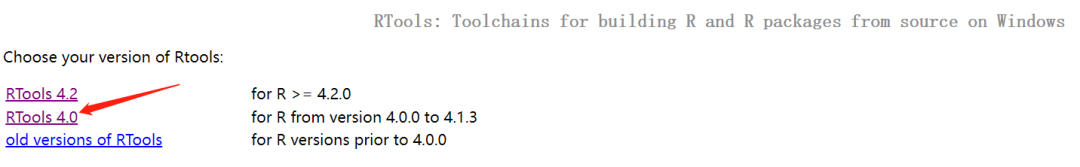 ② 选择红色箭头指向的"rtools40-x86_64.exe"下载; 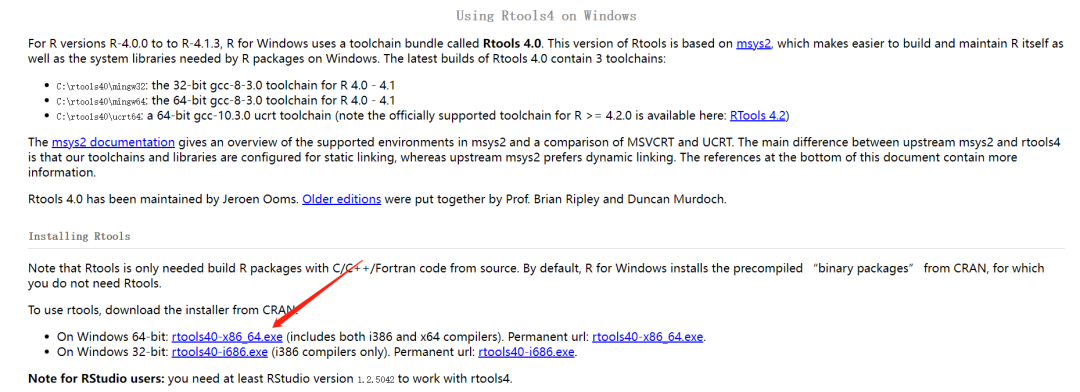 ③ 下载安装完成后(安装路径随意),进入Rstudio,在控制台输入; write('PATH="${RTOOLS40_HOME}\\usr\\bin;${PATH}"', file = "~/.Renviron", append = TRUE)④ 如果一切正常没有报错,重启Rstudio; ⑤ 测试路径配置是否成功。 Sys.which("make")只要输出不是空字符串就表明路径配置成功。 可能出现的报错:In file(file, ifelse(append, "a", "w")) : cannot open file 'D:/????/.Renviron': Invalid argument  解决办法: ① 重启Rstudio后运行getwd()命令,获取此时的工作目录。在工作目录中创建txt文件,将 PATH="  ② 再次重启Rstudio; ③ 输入"Sys.which("make")"测试路径配置是否成功。 Sys.which("make")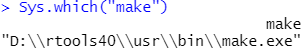 只要输出不是空字符串就表明路径配置成功。 2. 安装包如果已经安装,此步请跳过。 install.packages('Seurat') install.packages('dplyr') install.packages('tidyverse') install.packages('patchwork') install.packages("devtools") install_github("immunogenomics/harmony") 3. 加载包library(Seurat) library(dplyr) library(tidyverse) library(patchwork) library(devtools) library(harmony) 4. 设置工作路径setwd("D:/sc-seq/")请根据自己数据的存放位置自定义路径。 本次示例工作路径下存放了需要读取的10×数据文件夹:BC3和BC21。  5. 创建读取文件的向量## 将文件夹中的文件名存到"dir_name"中
dir_name 5. 创建读取文件的向量## 将文件夹中的文件名存到"dir_name"中
dir_name |
【本文地址】
公司简介
联系我们