| 高中生物:限制酶知识点及应用 | 您所在的位置:网站首页 › sds-page技术在下列方法中没有应用的是 › 高中生物:限制酶知识点及应用 |
高中生物:限制酶知识点及应用
|
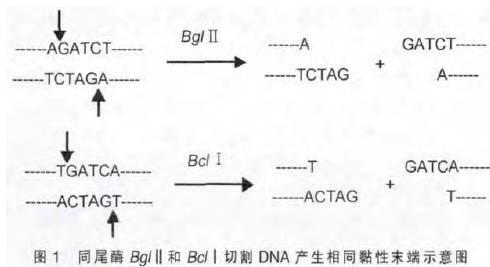
原标题:高中生物:限制酶知识点及应用 关于限制性核酸内切酶(下面简称限制酶)的定义是:能够识别和切割DNA 分子内一小段特殊核苷酸序列的酶。因为限制酶能切割DNA 分子,所以将其称为“基因的剪刀”、“分子手术刀”等。 1不同的限制酶产生的黏性末端一定不同吗? 不同限制酶识别的碱基序列一般不同,但切割后所产生的黏性末端可能是一样的,像这种能切割不同的DNA 片段但产生相同黏性末端的一类限制酶称为同尾酶。如Bgl Ⅱ和Bcl Ⅰ,它们的识别位点分别为AGATCT 和TGATCA, 但能切出相同的黏性末端,如图1 所示。
1不同的限制酶产生的黏性末端一定不同吗? 不同限制酶识别的碱基序列一般不同,但切割后所产生的黏性末端可能是一样的,像这种能切割不同的DNA 片段但产生相同黏性末端的一类限制酶称为同尾酶。如Bgl Ⅱ和Bcl Ⅰ,它们的识别位点分别为AGATCT 和TGATCA, 但能切出相同的黏性末端,如图1 所示。
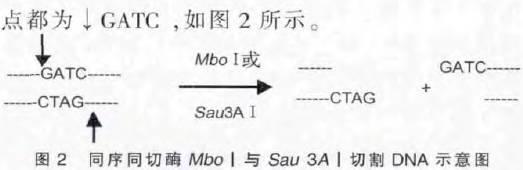
2不同的限制酶识别序列一定不同吗? 不一定。能识别相同序列的限制酶称为同裂酶,但其切割位点可能相同也可能不同。具体可分为以下几种情况: 1 )同序同切酶。这些酶的DNA 识别序列和切割位置都相同,如Mbo Ⅰ与Sau 3A Ⅰ识别切割位
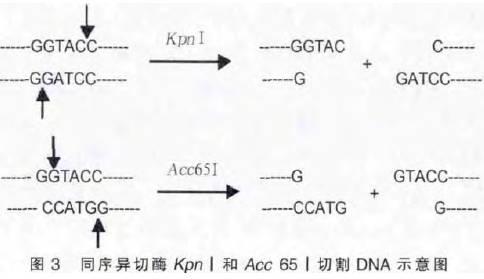
2 )同序异切酶。这些酶的识别序列相同但切割位点不同,如Kpn Ⅰ和Acc 65 Ⅰ识别的序列是都GGTACC ,但它们的切割位点不同,如图3 所示。
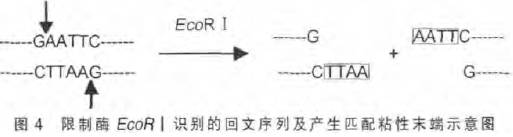
3 )同功多位酶。一些能识别简并序列的限制酶包含了另一种限制酶的功能。如EcoR Ⅰ识别和切割位点为G AATTC ,Apo Ⅰ识别和切割位点为NN AATTNNN ,后者可识别并切割前者的序列。 3限制酶的 DNA识别序列一定为回文序列吗? DNA 中的一段倒置重复序列,当该序列的双链被打开后,可形成发夹结构,这段序列被称为回文序列。其特点是在该段的碱基序列的互补链之间正读反读都相同(并非在同一条链上正读反读)。例如正读:5 ′GGTACC3 ′,反读:3 ′CCATGG5 ′。 1) 回文序列:限制酶识别的序列大多数为回文序列,切割位点在DNA2 条链相对称的位置。回文对称结构的序列经限制酶切割后,产生的末端为匹配黏性末端,这样形成的2 个末端是相同的,也是互补的,如图4 所示。
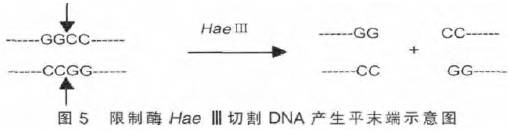
在回文对称轴上同时切割DNA 的2 条链,可能产生平末端,产生平末端的DNA 可任意连接,但连接效率较黏性末端低,如图5 所示。
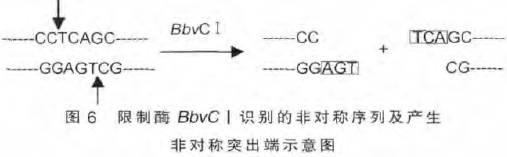
2) 非对称序列有一些限制酶的识别序列是不对称的。当识别序列为非对称序列时,切割DNA 产生的黏性末端是不同的,称为非对称突出端。如BbvC Ⅰ,它的识别切割位点见图6 。
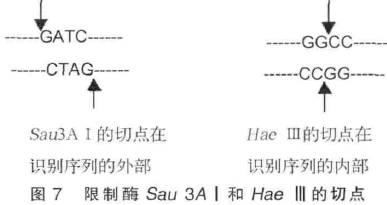
4限制酶的切点一定在识别序列内部吗? 限制酶对DNA 的切割位点大多数在识别序列内部,但也有在识别序列外部的,如图7 所示
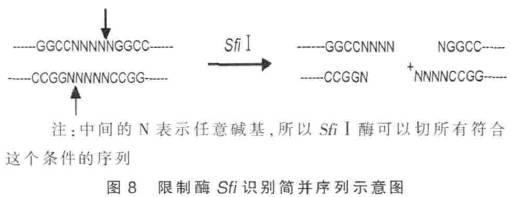
51种限制酶一定只能识别1种核苷酸序列吗? 多数的限制酶只能识别1 种特定的核苷酸序列,但也有例外, 如前面所讲的同功多位酶,能够识别多种核苷酸序列, 如图8 所示。
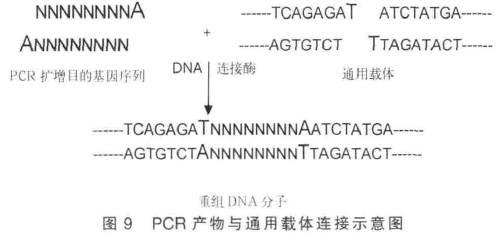
6 目的基因的获取一定用到限制酶吗? 在浙科版“现代生物科技专题”第5 页中提到获取目的基因通常有2 种方法,序列已知可以用化学方法合成或者用PCR 扩增目的基因,序列未知可以从基因文库中获取目的基因。但在实际题目中往往是让学生选择用哪种限制酶去获取目的基因,容易使学生形成“目的基因的获得一定用到限制酶”这样的错误认知。在构建某种生物的基因文库时常用到限制酶,通常是将某种生物材料的遗传物质DNA 用限制酶切成片段,插入到载体分子中,并将重组载体转入宿主细胞,然后再用某种探针选择、钓取目的基因。但是用化学合成法或PCR扩增目的基因过程中不一定用到限制酶,可见,目的基因的获得不一定需要限制酶。 7构建重组DNA时一定用到限制酶吗? 在浙科版“现代生物科技专题”第6 页中提到获得目的基因后,要将其与载体DNA 连接在一起,通常是用相同的限制酶分别切割目的基因和载体DNA,然后用DNA 连接酶将目的基因和载体DNA 连接在一起,形成重组DNA 分子。从“通常”2 个字可知构建重组DNA 时不一定用到限制酶,例如,利用PCR 技术获取目的基因时,通常是用耐热性DNA 聚合酶(如Taq DNA 聚合酶等)扩增目的基因序列。Taq DNA 聚合酶等扩增得到PCR 产物的3′端附有1 个“A”碱基,可以在DNA连接酶的作用下连接到PCR 产物克隆专用载体(也称为通用载体),如pMD18 -T 载体。在pMD18-T 载体等通用载体插入位点2侧的3′端添加了一个“T”碱基,这样恰好可以与PCR 产物末端的“A”碱基配对,得到含有目的基因序列的重组DNA 分子。这种重组DNA 分子可以很方便地转化到感受态细胞中,进行DNA 测序以及后续的相关实验,如图9 所示。
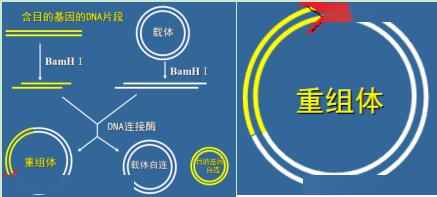
8构建重组DNA时一定用同种限制酶吗? 构建重组DNA 时,通常是用相同的限制酶分别切割目的基因和载体DNA ,这样会在目的基因和载体的两端形成相同的黏性末端,然后用DNA 连接酶将目的基因和载体连接在一起,形成重组DNA 分子。但是这种情况下的两两连接产物除了重组DNA 分子,还有目的基因- 目的基因连接产物、载体- 载体连接产物。为了防止目的基因和载体在酶切后产生的末端发生任意连接,酶切时常会选择不同的限制酶。 展开全文2不同的限制酶识别序列一定不同吗? 不一定。能识别相同序列的限制酶称为同裂酶,但其切割位点可能相同也可能不同。具体可分为以下几种情况: 1 )同序同切酶。这些酶的DNA 识别序列和切割位置都相同,如Mbo Ⅰ与Sau 3A Ⅰ识别切割位
2 )同序异切酶。这些酶的识别序列相同但切割位点不同,如Kpn Ⅰ和Acc 65 Ⅰ识别的序列是都GGTACC ,但它们的切割位点不同,如图3 所示。
3 )同功多位酶。一些能识别简并序列的限制酶包含了另一种限制酶的功能。如EcoR Ⅰ识别和切割位点为G AATTC ,Apo Ⅰ识别和切割位点为NN AATTNNN ,后者可识别并切割前者的序列。 3限制酶的 DNA识别序列一定为回文序列吗? DNA 中的一段倒置重复序列,当该序列的双链被打开后,可形成发夹结构,这段序列被称为回文序列。其特点是在该段的碱基序列的互补链之间正读反读都相同(并非在同一条链上正读反读)。例如正读:5 ′GGTACC3 ′,反读:3 ′CCATGG5 ′。 1) 回文序列:限制酶识别的序列大多数为回文序列,切割位点在DNA2 条链相对称的位置。回文对称结构的序列经限制酶切割后,产生的末端为匹配黏性末端,这样形成的2 个末端是相同的,也是互补的,如图4 所示。
在回文对称轴上同时切割DNA 的2 条链,可能产生平末端,产生平末端的DNA 可任意连接,但连接效率较黏性末端低,如图5 所示。
2) 非对称序列有一些限制酶的识别序列是不对称的。当识别序列为非对称序列时,切割DNA 产生的黏性末端是不同的,称为非对称突出端。如BbvC Ⅰ,它的识别切割位点见图6 。
4限制酶的切点一定在识别序列内部吗? 限制酶对DNA 的切割位点大多数在识别序列内部,但也有在识别序列外部的,如图7 所示
51种限制酶一定只能识别1种核苷酸序列吗? 多数的限制酶只能识别1 种特定的核苷酸序列,但也有例外, 如前面所讲的同功多位酶,能够识别多种核苷酸序列, 如图8 所示。
6 目的基因的获取一定用到限制酶吗? 在浙科版“现代生物科技专题”第5 页中提到获取目的基因通常有2 种方法,序列已知可以用化学方法合成或者用PCR 扩增目的基因,序列未知可以从基因文库中获取目的基因。但在实际题目中往往是让学生选择用哪种限制酶去获取目的基因,容易使学生形成“目的基因的获得一定用到限制酶”这样的错误认知。在构建某种生物的基因文库时常用到限制酶,通常是将某种生物材料的遗传物质DNA 用限制酶切成片段,插入到载体分子中,并将重组载体转入宿主细胞,然后再用某种探针选择、钓取目的基因。但是用化学合成法或PCR扩增目的基因过程中不一定用到限制酶,可见,目的基因的获得不一定需要限制酶。 7构建重组DNA时一定用到限制酶吗? 在浙科版“现代生物科技专题”第6 页中提到获得目的基因后,要将其与载体DNA 连接在一起,通常是用相同的限制酶分别切割目的基因和载体DNA,然后用DNA 连接酶将目的基因和载体DNA 连接在一起,形成重组DNA 分子。从“通常”2 个字可知构建重组DNA 时不一定用到限制酶,例如,利用PCR 技术获取目的基因时,通常是用耐热性DNA 聚合酶(如Taq DNA 聚合酶等)扩增目的基因序列。Taq DNA 聚合酶等扩增得到PCR 产物的3′端附有1 个“A”碱基,可以在DNA连接酶的作用下连接到PCR 产物克隆专用载体(也称为通用载体),如pMD18 -T 载体。在pMD18-T 载体等通用载体插入位点2侧的3′端添加了一个“T”碱基,这样恰好可以与PCR 产物末端的“A”碱基配对,得到含有目的基因序列的重组DNA 分子。这种重组DNA 分子可以很方便地转化到感受态细胞中,进行DNA 测序以及后续的相关实验,如图9 所示。
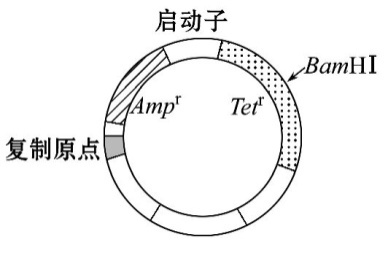
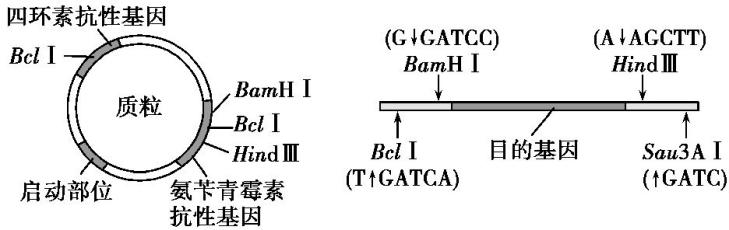
8构建重组DNA时一定用同种限制酶吗? 构建重组DNA 时,通常是用相同的限制酶分别切割目的基因和载体DNA ,这样会在目的基因和载体的两端形成相同的黏性末端,然后用DNA 连接酶将目的基因和载体连接在一起,形成重组DNA 分子。但是这种情况下的两两连接产物除了重组DNA 分子,还有目的基因- 目的基因连接产物、载体- 载体连接产物。为了防止目的基因和载体在酶切后产生的末端发生任意连接,酶切时常会选择不同的限制酶。 基因工程中常用两种限制酶酶切质粒和目的基因,可以为防止错误连接及自身环化、和反向链接。但实际操作中要考虑到目的基因两端可能没有两种限制酶的酶切位点;若是人工合成需要人为的加上粘性末端。 例1 2016·全国卷Ⅰ节选:某一质粒载体如图所示,外源DNA插入Ampr或Tetr中会导致相应的基因失活(Ampr表示氨苄青霉素抗性基因,Tetr表示四环素抗性基因)。有人将此质粒载体用BamHⅠ酶切后,与用BamHⅠ酶切获得的目的基因混合,加入DNA连接酶进行连接反应,用得到的混合物直接转化大肠杆菌,结果大肠杆菌有的未被转化,有的被转化。被转化的大肠杆菌有三种,分别是含有环状目的基因、含有质粒载体、含有插入了目的基因的重组质粒的大肠杆菌。回答下列问题:
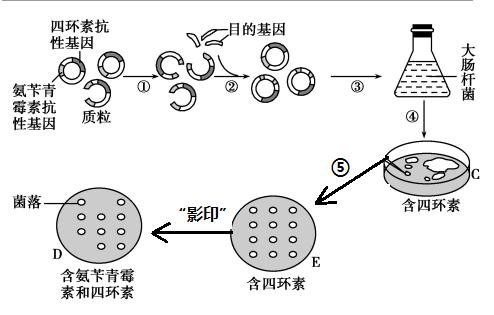
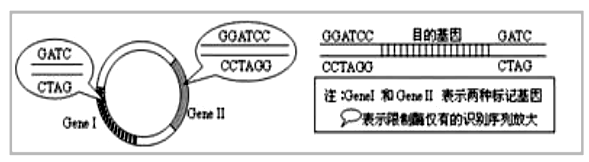
(2) 如果用含有氨苄青霉素的培养基进行筛选,在上述四种大肠杆菌细胞中,未被转化的和仅含环状目的基因的细胞是不能区分的,其原因是① ;并且② 和 ③ 的细胞也是不能区分的。其原因是 ④ 。在上述筛选的基础上,若要筛选含有插入了目的基因的重组质粒的大肠杆菌单菌落,还需使用含有⑤ 的固体培养基。 答案:① 二者均不含有氨苄青霉素抗性基因,在该培养基上均不生长; ②含有质粒载体(空载);③含有重组质粒;④二者均含有氨苄青霉素抗性基因,在该培养基上均能生长;⑤四环素 解析:目的基因和载体有同一种酶酶切后,会形成相同的粘性末端,再用DNA 链接酶催化时,会有下图的三种情况。且上述题目中,目的基因插入破坏了四环素的抗性基因,因此导入了重组质粒的受体菌,可以在含有青霉素的培养基生长,但不能在含有四环素的培养上生长。 例2 科学家将外源目的基因与大肠杆菌的质粒进行重组,并在大肠杆菌中成功表达。如图表示构建重组质粒和筛选含目的基因的大肠杆菌的过程。请据图回答问题。
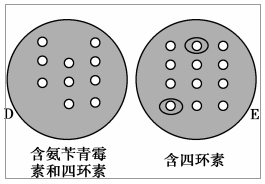
(1)图中质粒上有抗氨苄青霉素和抗四环素两个标记基因,经过①和②步骤后,有些质粒上的 基因内插入了外源目的基因,形成重组质粒,由于目的基因的分隔使得该抗性基因失活。 (2)步骤③是 的过程,为了促进该过程,应该用处理大肠杆菌。 (3)步骤④将三角瓶内的大肠杆菌接种到含四环素的培养基C上培养,目的是筛选 ,能在C中生长的大肠杆菌有种 (4)步骤⑤:用无取C上的单个菌落,分别接种到E(含四环素),然后用影印法接种到D(含氨苄青霉素和四环素)上,一段时间后,菌落的生长状况如图所示。含目的基因的菌落位于(填D或E)上,请在图中相应的位置上圈出来。 答案(1)氨苄青霉素抗性;(2)将重组质粒(目的基因)导入大肠杆菌( 受体细胞)CaCl 2溶液;(3) 含四环素抗性基因的大肠杆菌 2;(4)E
例3 2018浙江温州3月选考模拟 利用图示质粒和目的基因培育转基因醋化醋杆菌。图中标注了相关限制性核酸内切酶的识别序列和酶切位点,其中酶切位点相同的酶不重复标注。请回答下列有关转基因的问题:
(1) 利用图示质粒和目的基因构建重组质粒,应选用两种限制酶切割。 解析:为防止错误连接(包括 反向链接)和自身环化应 采用BamH Ⅰ和HindⅢ两种限制性核酸内切酶切割。
2020 年1月浙江高考29:为了提高目的基 因和质粒重组的成功率,选择使用____________,对含目的基因的DNA片段进行处理,使其两端形成不同的粘性末端,对质粒也进行相同的处理,然后用DNA连接酶连接形成重组质粒。通过在大肠杆菌中________,以获得大量的重组质粒,最终将其导入原生质体中。 答案:两种限制性核酸内切酶 扩增
第一空考查了限制酶切割的两种方式,即黏性末端和平末端是怎么产生的。
常规的考法就是这样: 裸奔式:目的基因的获取需要 酶切割。 情景式:从图中选择目的基因插入的位置,需要具体什么酶。 关于限制酶的考题方式: 一、酶切结果的推导 限制酶作为酶类,决定其催化作用具有高效性、专一性和作用条件温和等特点。其中专一性的具体表现为:某种限制酶只能催化DNA 上特定序列特定位点的磷酸二脂键水解,结果是使DNA 分子被分割成不同的DNA 片段。限制酶所识别的双链 DNA 上的序列具有“ 回文对称” 性质,即无论是奇数个碱基还是偶数个碱基,都可以找到一条中心轴线,中心轴线两侧的双链DNA 上的碱基是反向对称重复排列的。当限制酶在它识别序列的中心轴线两侧将DNA 的两条链分别切开时,即错位平切,产生的是黏性末端;当限制酶在它识别序列的中心轴线处切开时,即平切,产生平末端。错位切的限制酶在基因工程中最常使用,因为与平末端相比较,黏性末端更有利于DNA 片段的连接。 例1下面是5种限制性内切酶对DNA分子的识别序列和剪切位点图(↓表示剪切点、切出的断面为黏性末端): 限制酶1:——↓GATC——; 限制酶2:——CATG↓——; 限制酶3:——G↓GATCC——; 限制酶4:——CCGC↓GG——; 限制酶5:——↓CCAGG——。 请指出下列哪组表达正确() A.限制酶2和4识别的序列都包含4个碱基对 B.限制酶3和5识别的序列都包含5个碱基对 C.限制酶1和3剪出的黏性末端相同 D.限制酶1和2剪出的黏性末端相同 二、酶切方法的选择 构建重组质粒,需要用限制酶分别切割含目的基因的DNA 片段和质粒,切割方法主要有单酶切和双酶切。单酶切是指用同一种限制酶对含目的基因的DNA 和质粒进行切割,目的基因序列的两边必须各有一个能被该限制酶识别的序列及切点,而质粒至少有一个识别序列及切点,这样被切割的质粒与切下的目的基因两边形成相同的黏性末端,以实现连接而形成重组质粒,有时也可以用两种不同的限制酶(同尾酶)切割产生相同的黏性末端。双酶切是指用两种限制酶进行切割,含目的基因的DNA 和质粒上都必须有能被这两种限制酶识别的序列及切点,结果两者都会产生具有两种不同的黏性末端的DNA 片段,两个末端分别用A与B表示,然后混合起来,那么载体分子的A端与目的基因的A端连接,载体分子的B端与目的基因的B端连接,形成重组DNA分子,这样就可以避免DNA 连接酶催化时被切割后的质粒自连成环的现象,并能保证质粒与目的基因的定向连接。 例2基因工程中,需使用特定的限制酶切割目的基因和质粒,便于重组和筛选。已知限制酶I的识别序列和切点是—G↓GATCC-,限制酶Ⅱ的识别序列和切点是-↓GATC-。根据图示,判断下列操作正确的是( )
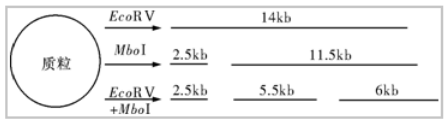
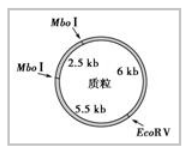
A.质粒用限制酶Ⅰ切割,目的基因用限制酶Ⅱ切割 B.质粒用限制酶Ⅱ切割,目的基因用限制酶Ⅰ切割 C.目的基因和质粒均用限制酶Ⅰ切割 D.目的基因和质粒均用限制酶Ⅱ切割 三、酶切图谱的构建 酶切图谱是指选取某一特定长度的DNA 分子,用多种限制酶单独或联合切割,再通过电泳技术将酶切片段进行分离,计算相对大小,以确定每一种限制酶在DNA分子中的切点位置和相对距离,并据此绘制而成的物理图谱(一般以直线或环状图式表示)。 例3.用限制酶EcoRⅤ、MboⅠ单独或联合切割同一种质粒,得到的DNA片段长度如下图(1kb即1000个碱基对),请在下图中画出质粒上EcoRⅤ、MboⅠ的切割位点。
参考答案:
▍ 来源:综合网络 ▍ 编辑: Wordwuli ▍ 声明:如有侵权,请联系删除;若需转载,请注明出处。返回搜狐,查看更多 责任编辑: |
【本文地址】