| R语言实践 | 您所在的位置:网站首页 › r语言聚类分析实验报告 › R语言实践 |
R语言实践
|
使用 rWCVP 生成自定义清单
介绍1. 特有物种清单2. 近特有物种清单2.1 在塞拉利昂和另一地区出现的物种2.2 在塞拉利昂和相邻地区出现的物种
3. 生成自定义报告
介绍
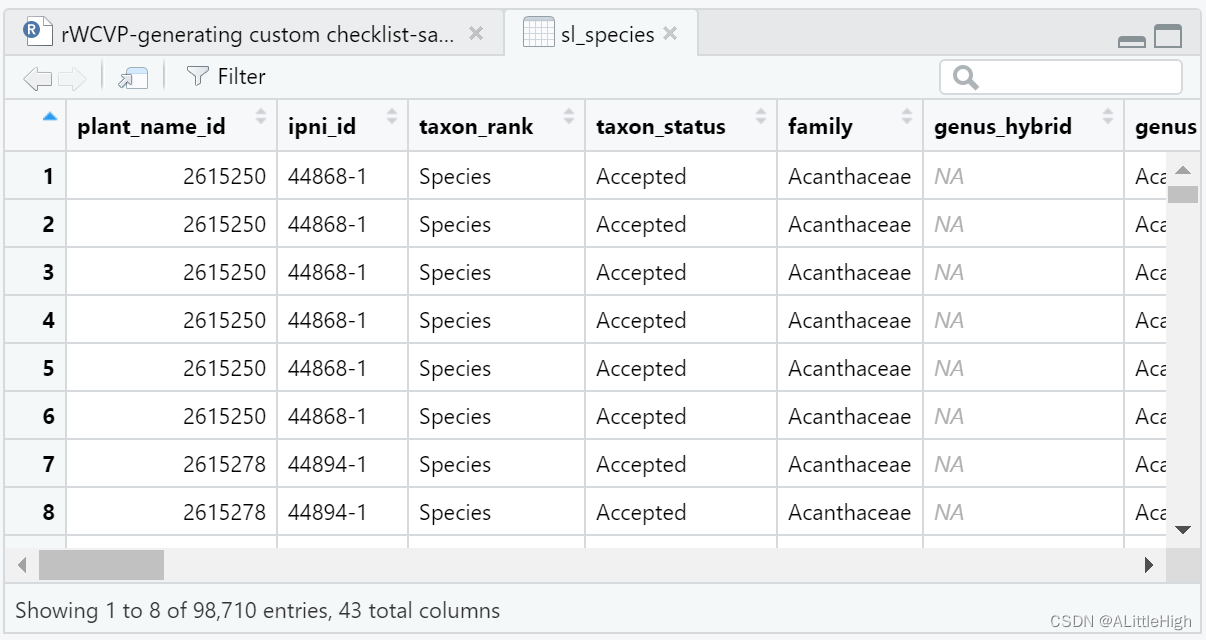
除了允许用户从世界维管植物名录(WCVP)创建清单外,rWCVP还提供了修改清单输出以生成自定义报告的功能。本文通过生成塞拉利昂特有(或近特有)物种列表来证明这一点。 除了 rWCVP 之外,我们还将使用 tidyverse 包进行数据操作和绘图,使用 gt 包来渲染漂亮的表格,并使用 sdpep 包来查找边界区域。 先做好准备工作 library(rWCVP) library(tidyverse) library(gt) library(spdep)在此示例中,使用==管道运算符 (%>%) 和 dplyr语法 ==- 如果不熟悉这些,我建议查看 https://dplyr.tidyverse.org/ 和其中的一些帮助页面。 1. 特有物种清单我们首先生成塞拉利昂物种清单。记住和/或找到合适的 WGSRPD 3 级代码是一种痛苦,因此我们可以在函数调用中使用 get_wgsrpd3_codes(“Sierra Leone”)为我们完成这项工作。 sl_code = get_wgsrpd3_codes("Sierra Leone") sl_species = wcvp_checklist(area=sl_code, synonyms = FALSE)synonyms = FALSE表示只保留接受名。结果有近10万条。 塞拉利昂有多少物种,有多少是特有的?我们可以在这里选取特有列,非常简单。 (endemic_summary = sl_species %>% distinct(taxon_name, endemic) %>% group_by(endemic) %>% summarise(number.of.sp=n())) # A tibble: 2 × 2 endemic number.of.sp 1 FALSE 3303 2 TRUE 45容易!对于特有物种列表,我们可以简单地使用特有列过滤我们的清单,但是近特有物种呢? 2. 近特有物种清单根据我们对近特有物种的定义,有两种办法进行筛选: 我们将近特有物种定义为出现在塞拉利昂和另一个WGSPRD3地区(L3)的物种。从数据的角度来看,这意味着过滤掉sl_species中有 >2 行的物种(因为每一行都是物种区域出现)。或者,我们可以考虑近特有物种,因为那些可能跨越边界的物种,因此在功能上是地方性的。为此,我们需要a)确定相邻的WGSPRD3区域,b)相应地过滤我们的物种列表。 2.1 在塞拉利昂和另一地区出现的物种我们只需直接从塞拉利昂物种列表中移除分布地超过2处的物种即可。 sl_near_endemics1 = sl_species %>% group_by(plant_name_id, taxon_name) %>% filter(n() % ungroup()
近特有物种有112种,还可以根据数字判断哪些是新增的,哪些是原有的特有物种。 首先,我们需要确定哪些WGSRPD地区与塞拉利昂接壤。 我们可以通过查看地图来做到这一点,但我们将使用编程方式操作图层文件。为此,我们采用 WGSRPD 级别 3 多边形并找到彼此接壤的所有区域。 sf_use_s2(FALSE) area_polygans = rWCVPdata::wgsrpd3 area_neighbours = poly2nb(area_polygans)请注意,我们必须为此使用 sf_use_s2(FALSE) 关闭 sf 中的球面坐标。 现在我们有一份邻近地区的清单,我们需要找到与塞拉利昂接壤的地区。 sl_index = which(area_polygans$LEVEL3_COD %in% sl_code) sl_neighbours_index = area_neighbours[[sl_index]] sl_plus_neighbours = area_polygans[c(sl_index, sl_neighbours_index),]
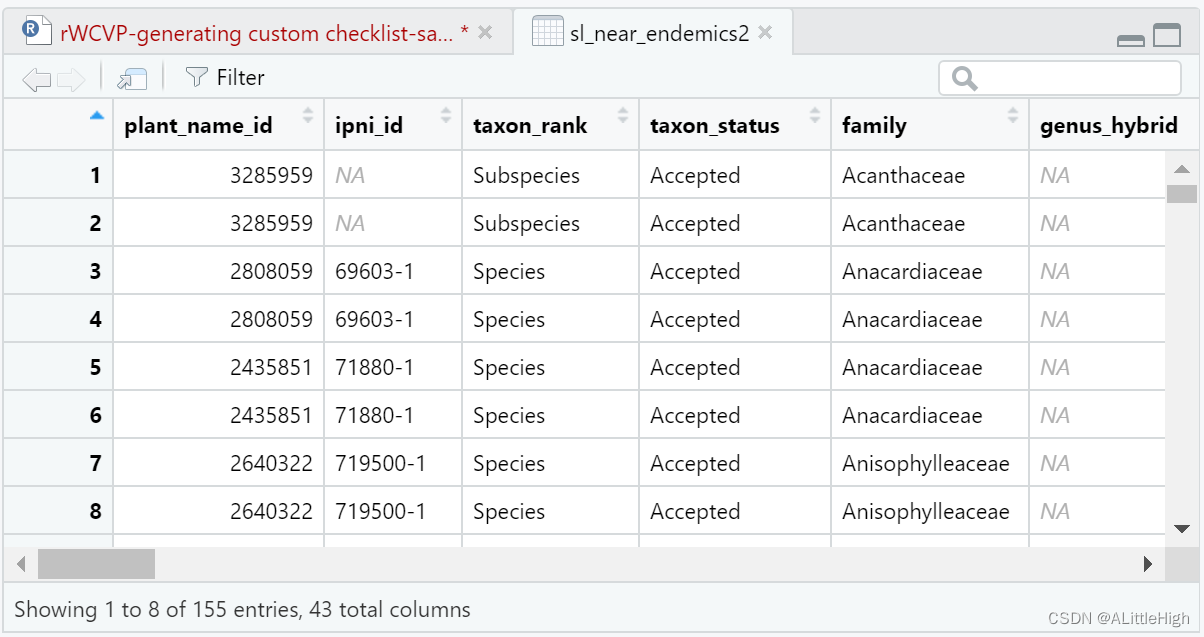
接下来,我们可以将近特有物种确定为仅在塞拉利昂,几内亚或利比里亚出现的物种。 sl_near_endemics2 = sl_species %>% group_by(plant_name_id) %>% filter(all(area_code_l3 %in% sl_plus_neighbours$LEVEL3_COD)) %>% ungroup()
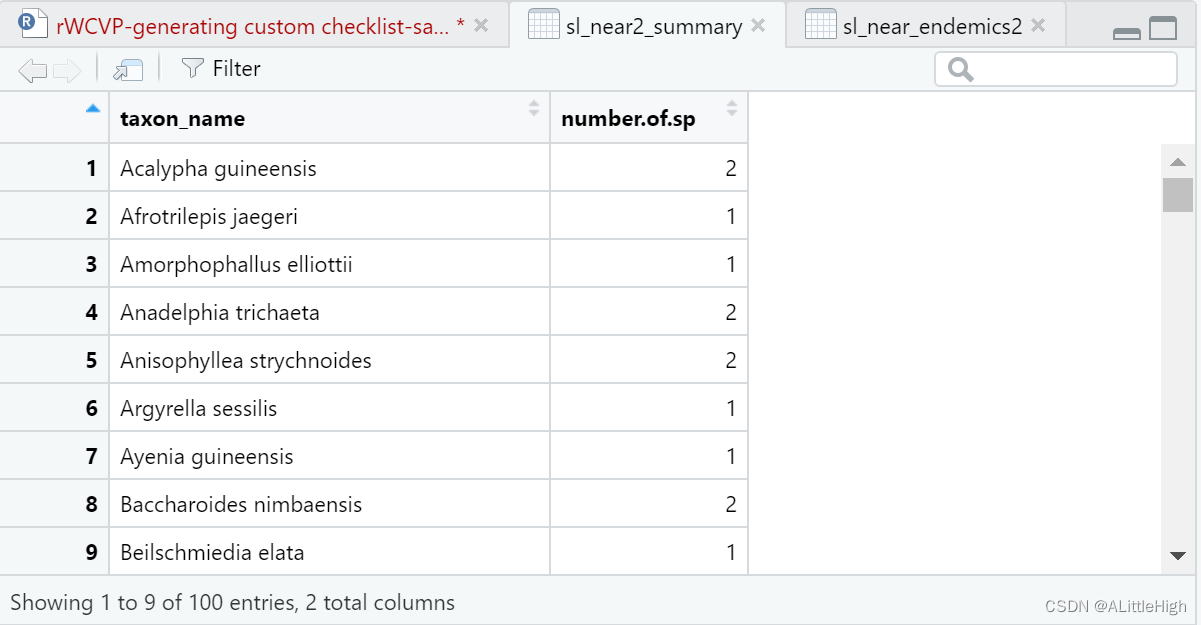
同样,除掉重复看实际上有多少个近特有物种: sl_near2_summary = sl_near_endemics2 %>% group_by(taxon_name) %>% summarise(number.of.sp=n())
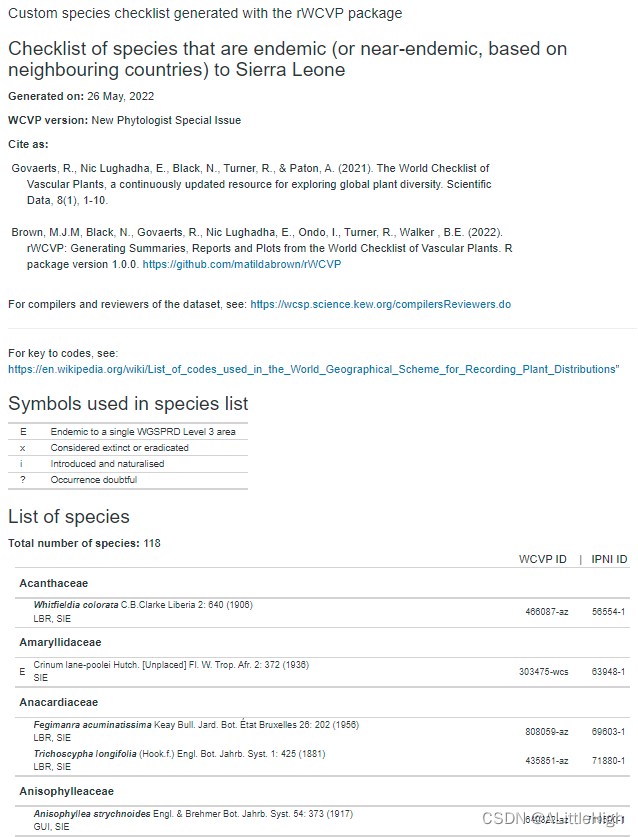
现在我们可以做一些花哨的事情 - 将我们的清单数据框转换为格式化报告。为此,我们将其插入名为“custom_checklist.Rmd”的模板文件中,该文件存储在 rWCVP 包文件夹(”rmd“子文件夹)中。我们使用参数传递数据(以及其他一些信息),并且需要使用output_file指定文件名。 library(rmarkdown) checklist_description = "Checklist of species that are endemic to Sierra Leone (or near-endemic, based on neighbouring countries)" wd = getwd() render(system.file("rmd", "custom_checklist.Rmd", package = "rWCVP"), quiet = TRUE, params=list( version = "New Phytologist Special Issue", mydata = sl_near_endemics2, description = checklist_description), output_file = paste0(wd,"/Sierra_Leone_endemics_and_near_endemics.html"))这个步骤报错,我没有能力解决 看看应该出现的结果吧: |
【本文地址】
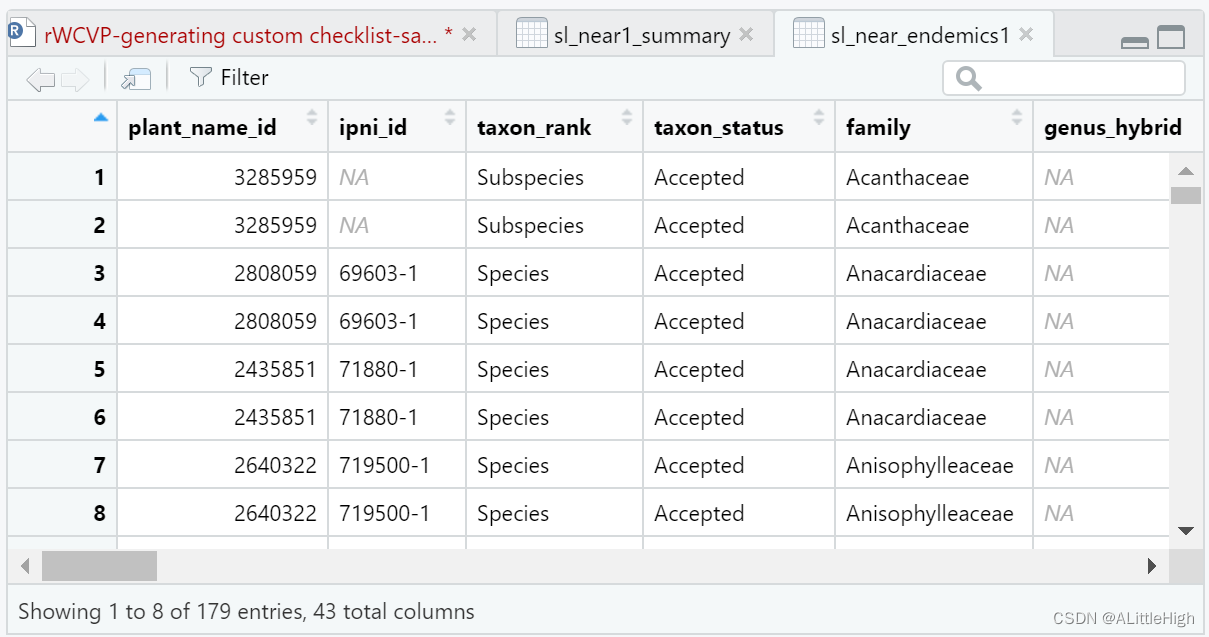 结果有179条,但里面有重复(即由于近特有物种定义引起的),总结后:
结果有179条,但里面有重复(即由于近特有物种定义引起的),总结后: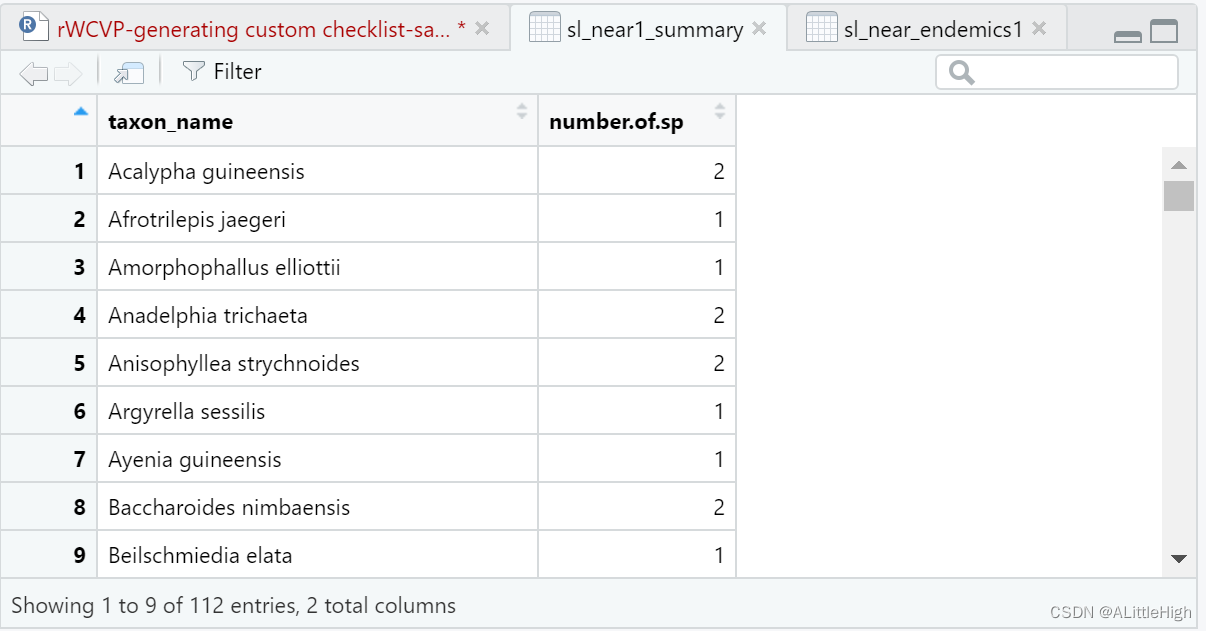
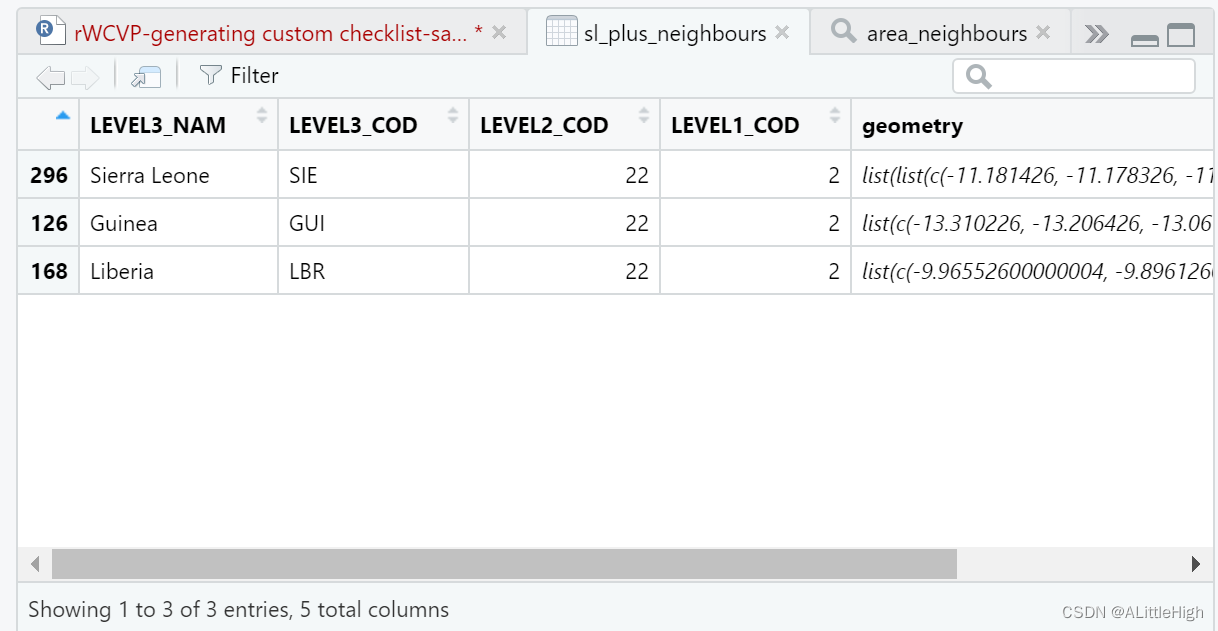 在生成最终清单之前,检查此自动邻居检测的健全性。然后可以将区域映射到地图中
在生成最终清单之前,检查此自动邻居检测的健全性。然后可以将区域映射到地图中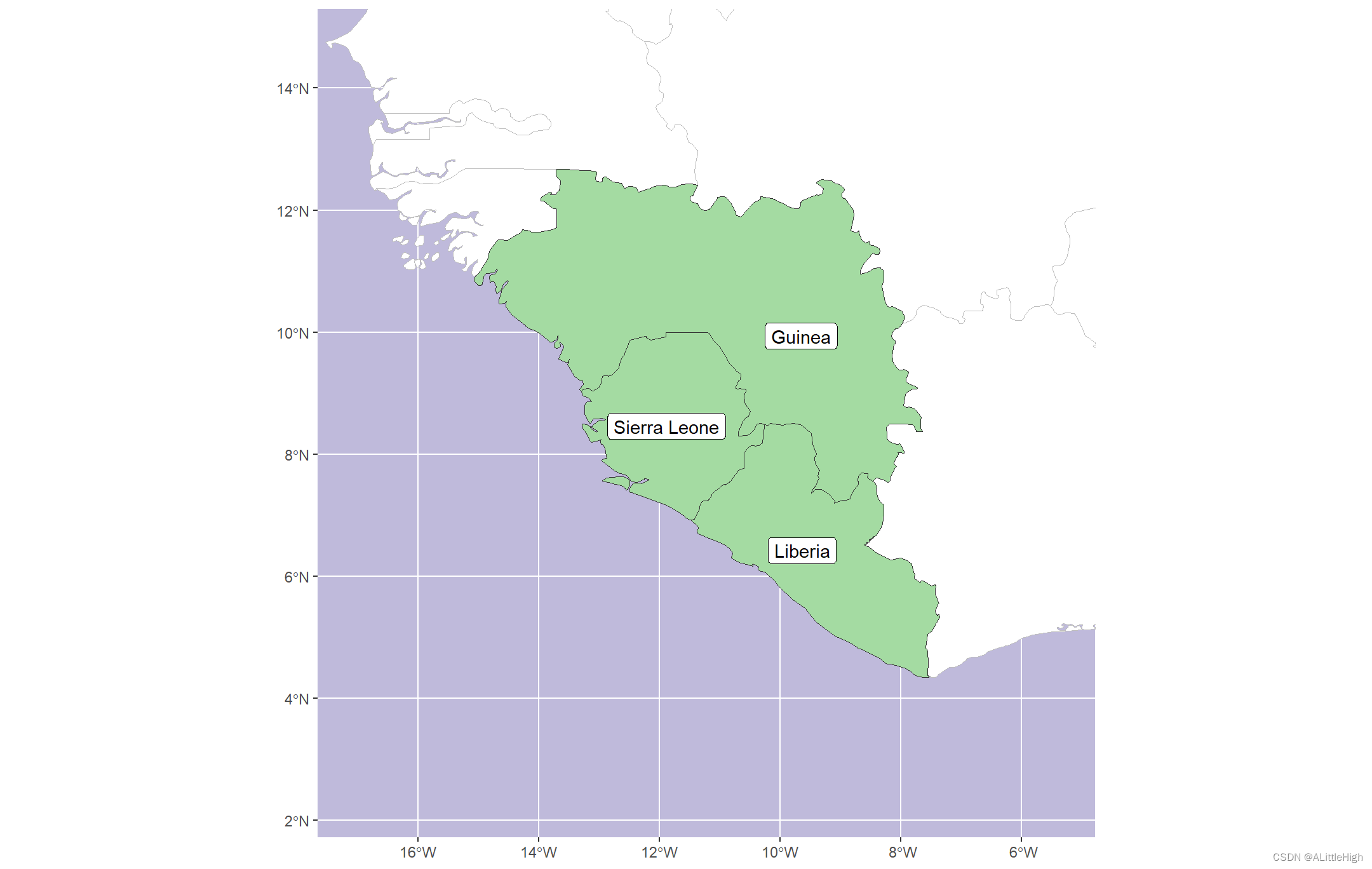 当然,我们可以从地图上将几内亚和利比里亚确定为邻国,然后使用get_wgsrpd3_codes(“利比里亚”)和get_wgsrpd3_codes(“几内亚”)找到代码,但这并不那么有趣!
当然,我们可以从地图上将几内亚和利比里亚确定为邻国,然后使用get_wgsrpd3_codes(“利比里亚”)和get_wgsrpd3_codes(“几内亚”)找到代码,但这并不那么有趣! 最后,我们将列表过滤到仅发生在塞拉利昂+一个邻国的物种,就像我们在选项1中所做的那样。从地图上看,一个物种似乎可能发生在三个国家之间的三重交界处,但对于这个例子,我们将排除这些物种。
最后,我们将列表过滤到仅发生在塞拉利昂+一个邻国的物种,就像我们在选项1中所做的那样。从地图上看,一个物种似乎可能发生在三个国家之间的三重交界处,但对于这个例子,我们将排除这些物种。