| 一种增强放线菌基因编辑效率的方法及其应用与流程 | 您所在的位置:网站首页 › pkc1139质粒构建丢失原件 › 一种增强放线菌基因编辑效率的方法及其应用与流程 |
一种增强放线菌基因编辑效率的方法及其应用与流程
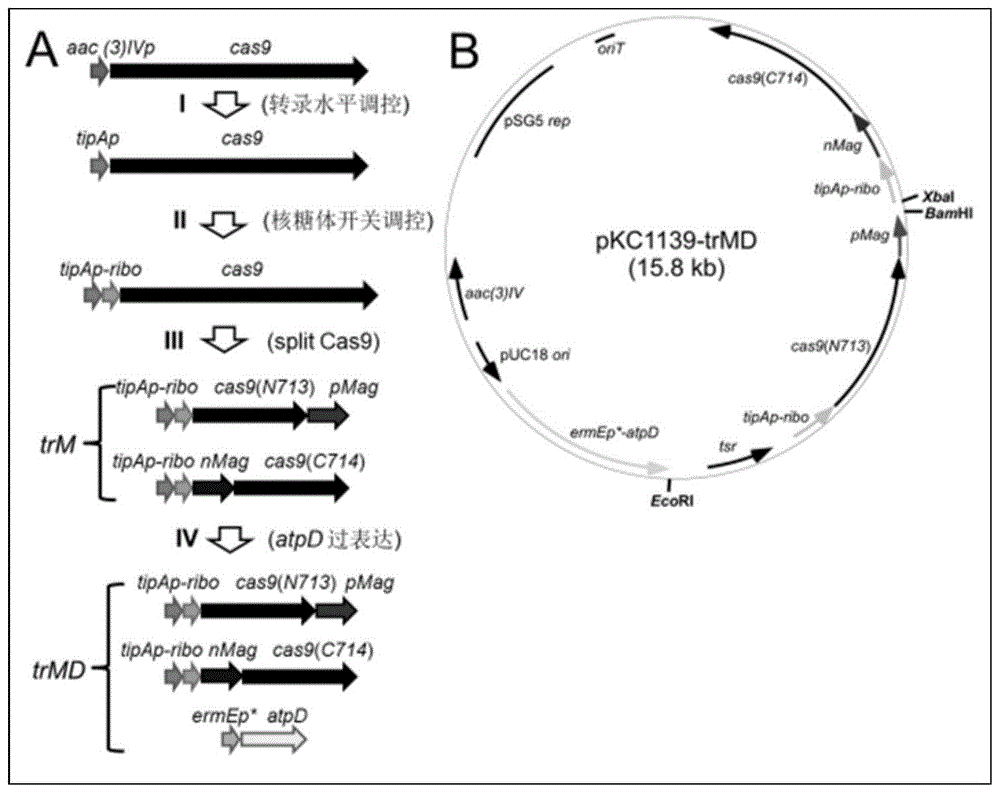 本发明属于基因工程和基因遗传修饰领域,明确地说,涉及在放线菌中通过控制cas9活性及atp浓度增强基因编辑效率的方法及应用。 背景技术: crispr(clusteredregularlyinterspacedshortpalindromicrepeats)/cas(crispr-associated)系统是原核生物中特有的免疫系统,伴随有特异性序列的rna介导,对外源dna识别并降解,引起外源dna功能缺使其失活。近几年,crispr/cas9系统作为一种针对特异性位点的基因定向编辑技术,具有操作简易便捷、投入少、效率高、具有良好特异性和普适性等优势,近两年被认为是分子机制解剖和基因表达控制最强大的基因组编辑工具包,在哺乳动物蛋白质工程,治疗干预和药物发现的合成生物学的细胞重编程方面表现出巨大的潜力,并且还用于微生物代谢工程,巨型染色体构建甚至操纵遗传上棘手的微生物。 crispr/cas9系统可对目的基因精确编辑。将外源的dna指引到宿主细胞染色体的特异性位点上,实现定位定向地基因编辑。该系统首先利用设计的引导rna(single-guiderna,sgrna)介导cas9蛋白与目的位点特异性结合,完成dna的特异性切割,并通过同源重组或非同源末端连接的方式自我修复,从而实现基因的敲除、敲入等。 然而,crispr/cas9系统的脱靶效应和毒性活性会极大程度地妨碍其应用。cas9毒性在微生物操作中的可见后果是转化体的急剧减少,例如在优秀的基因工程宿主大肠杆菌、酵母和链霉菌中,最常导致遗传修饰失败。其中链霉菌在临床使用药物生产中具有特殊工业价值。因此,如何在提高crispr/cas技术基因编辑效率的同时提高其靶向性,是该技术目前面临的一大难题。 目前研究表明,链霉菌中发现一种硫链丝菌素诱导型启动子tipap,可用于crispr/cas9编辑系统中抑制cas过表达的调控元件。并可以通过人为添加硫链丝菌素,诱导tipap启动子开放转录,在编辑过程中实现cas9活性重建,达到在转录水平控制cas9活性的目的。 另一方面相关研究报导,长度达85bp的茶碱依赖性核糖体开关,在非诱导条件下转录出的非编码rna存在高级结构,可以阻止下游编码基因表达的mrna与核糖体结合,抑制多肽链合成使蛋白表达处于极低水平,可用于转化效率的控制。但是在茶碱诱导下可以解开高级结构,因此加入茶碱后可产生数百倍的诱导表达,这种遗传工具可实现在翻译水平上控制cas9活性。 此外,目前已有研究开发了一种在人类细胞中实现cas9活性控制的蓝光诱导型cas9蛋白重建技术。真菌光受体vivid被设计分成两种蛋白质,即正极蛋白(pmag)和负极蛋白high1(nmaghigh1),这两种蛋白在蓝光诱导条件下异质二聚化,重建cas9结构与活性。这种光可切换的优势已被用于开发可光活化的裂解cas9(n713-pmag和nmaghigh1-c714),实现在蛋白质水平精确控制cas9活性和基因编辑。 此外,由cas9引起的双链断裂需通过同源定向重组(hdr)进行精确修复,该同源定向重组(hdr)由atp依赖性dna修复系统施加,包括真核细胞中的rad51/rad52和大肠杆菌中的reca触发的sos途径。其中reca是一种atp依赖性重组酶,在dsb后在单链dna上形成细丝,随后促进同源搜索和交换以进行同源重组,从而保证基因组的完整性和稳定性。而atp在hdr期间起着重要作用,因为atp与dna上的rad51/reca细丝相关,并且atp的水解至少在reca-ssdna,reca沿dna滑动的之间动态相互作用、防止长重复序列之间的配对以及rad51细丝拆解过程中的构象变化等方面起重要作用。因此在编辑过程中提升atp浓度利于同源定向重组(hdr),进一步提升基因编辑效率。 出于上述考虑和背景研究,本发明公开了一种在放线菌中通过控制cas9活性及atp浓度增强基因编辑效率的方法及其应用。该方法明确,高效,易操作,提供了一种有效提高微生物宿主中目的基因编辑效率的新方法。 技术实现要素: 本发明的目的是提供一种增强放线菌基因编辑效率的方法及其应用,是一种在放线菌中通过控制cas9活性及atp浓度增强基因编辑效率的方法,针对的是为目的基因编辑效率的提高,提供一种新型的crispr/cas9系统。这是一种基于添加控制cas9活性及atp浓度的元件,高效调控转化效率和编辑效率的新技术。本发明方法具体通过如下步骤实现: (1)根据目的宿主生理特性挑选合适的诱导型启动子、强启动子和骨架质粒; (2)将步骤(1)筛选的诱导型启动子、核糖体开关、splitcas9以及强启动子-atpd高供能基因盒插入到步骤(1)所选骨架质粒中; (3)设计目的编辑位点的间隔序列(左臂加右臂),分别插入步骤(2)所构质粒,用于引导cas9蛋白向靶序列移动; (4)根据宿主生理性质和质粒元件,选取合适的筛选转化子的抗生素或营养缺陷型等筛选方式,也在步骤(3)构建的质粒中另外添加表达抗生素和营养缺陷型等的相关基因; (5)在非诱导条件下,将步骤(4)中的重组质粒转化(或接合转导等适合于目的宿主的遗传操作)进入目的宿主中,通过添加筛选条件的培养基筛选,筛选得到携带重组质粒的转化子; (6)将步骤(5)中的转化子接种带有合适筛选条件的富集培养基,恒温发酵; (7)取步骤(6)中发酵24h–48h的发酵液,离心收取菌体,用无菌水清洗两次,离心弃上清,更换添加合适浓度启动子诱导条件和茶碱的培养基,随后在蓝光诱导条件下摇床恒温培养; (8)取(7)中发酵液离心,弃上清,收集的沉淀就是完成目的基因编辑的菌株。 本发明的目的是提供所述方法在有效提高目的宿主基因编辑效率中的应用。 1.本发明方法是通过筛选合适的诱导型启动子(如硫链丝菌素诱导型启动子tipap)实现在转化过程中抑制csa9蛋白过表达,后续添加启动子诱导条件(如硫链丝菌素)诱导重建cas9活性,实现在转录水平精准控制cas9活性。 2.本发明方法是通过茶碱依赖性核糖体开关在转化过程中抑制cas9蛋白多肽链的合成,后续添加茶碱诱导核糖体开关开启多肽链的合成,实现在翻译水平精准控制cas9活性。 3.本发明方法是通过蓝光诱导型分裂cas9蛋白在转化过程中分裂成两个蛋白而导致功能缺失,后续添加蓝光诱导两种蛋白异质二聚化,重建cas9结构与活性,实现在蛋白质水平精准控制cas9活性。 4.本发明方法是通过编码atp合酶的β-亚基的atpd基因在强组成型启动子(如ermep*)下表达,提供atp利于同源定向重组(hdr),提高编辑效率。 5.本发明提出一种在放线菌中通过控制cas9活性及atp浓度增强基因编辑效率的新型crispr/cas9系统,该系统添加合适的诱导型启动子(如硫链丝菌素诱导型启动子tipap)、核糖体开关、以及splitcas9实现对cas9活性的三重控制,最大限度抑制未诱导时crispr/cas9系统的细胞毒性与脱靶概率;构建的atp合酶的β-亚基的atpd基因保证atp的有效供给,进一步提高目的宿主中的基因编辑效率,本发明可适用于包含链霉菌在内的其他模式或工业放线菌的基因工程和高效遗传改造。 与现有技术相比,本发明的增益效果和明显优点是:1)本发明为放线菌等模式微生物的基因工程以及遗传修饰改造提供了人工诱导调控cas9活性及atp浓度,并有效提高编辑效率的方法工具,本发明的步骤快速通用,且操作简单,效果显著。2)本发明构建一种添加有合适的诱导型启动子(如硫链丝菌素诱导型启动子tipap)、核糖体开关、以及splitcas9三重控制的crispr/cas9系统,非诱导条件下明显增加转化效率。3)本发明通过添加诱导型启动子诱导条件(如硫链丝菌素)、茶碱以及蓝光诱导cas9恢复表达,并重组cas9结构及其活性。4)本发明编码atp合酶的β-亚基的atpd基因在强组成型启动子(如ermep*)下表达,提升放线菌体内atp浓度,进一步显著提升基因编辑效率。5)本发明在放线菌等微生物的基因工程中应用广泛,所有可以进行遗传操作的模式或者工业放线菌都可以使用本发明进行高效遗传改造,提高微生物生产工业的经济效益。 附图说明 图1为天蓝色链霉菌通用编辑质粒pkc1139-trmd构建过程示意图。 图2为天蓝色链霉菌a3(2)中敲除actii-orf4基因原理图。 图3为pkc1139-trmd-cas9-actii-orf4以及各对照组质粒对于天蓝色链霉菌a3(2)接合转导效率验证。接合转导后通过抗生素筛选,得到肉眼可见,实现计数的转化子。平板计数可知:逐步添加各个调控原件直到终质粒pkc1139-trmd-cas9-actii-orf4,转化子数目逐步增多,且最终转化效率相比于传统的组成型cas9表达,提高约260倍。 图4为非诱导条件下,成功导入pkc1139-trmd-cas9-actii-orf4质粒的菌株pcr敲除验证。将成功的转化子进行菌落pcr验证。结果所示,在非诱导条件下,仅19#用两对引物都扩增出1.35kb条带,即cas9体系的敲除效率仅为5% 图5为三重诱导条件下,成功导入pkc1139-trmd-cas9-actii-orf4质粒的菌株pcr敲除验证。将成功的转化子进行菌落pcr验证。结果所示,在三个诱导条件都具备且自主复制的情况下,优化后的cas9体系的敲除效率可高达80% 具体实施方式 下面结合附图和具体实施例作进一步详细描述本发明的实施方法。 实施例1 实施例1中选取的菌种为天蓝色链霉菌streptomycescoelicolora3(2)(bentleysdetal.nature,2002)(保藏号:cgmcc4.7168),其全基因组序列(序列编号:genbank:nc_003888.3)。以使用本方法敲除天蓝色链霉菌a3(2)中actii-orf4基因为例,详细描述本发明,其中天蓝色链霉菌streptomycescoelicolora3(2)是作为模式菌。具体步骤如下: (1)根据相关链霉菌启动子调控的文献报导(hehuangetal.actabiochimbiophyssin,2015),筛选出受硫链丝菌素诱导的tipap启动子和ermep*启动子,得到可用作目的菌株天蓝色链霉菌a3(2)的组成型启动子和atpd基因表达的强启动子。(详见表1与附图1) (2)根据天蓝色链霉菌a3(2)质粒库和确定元件的大小、位置构建过程合适的酶切位点和适合于转化子筛选的抗生素抗性基因,筛选出pkc1139质粒作为编辑质粒的骨架质粒。 (3)根据步骤(1)和步骤(2)中确定的启动子和骨架质粒,构建编辑质粒 为了保证csa9在tipap(序列详情见seqno.1)和riboswitch(序列详情见seqno.2)的调控下表达,将质粒pwhu2653和pet28a(novagene)采用ecori/xbai酶切,并将长4.7kb且包含有cas9-grna的酶切片段连接到pet28a载体酶切后得到的长5.2kb的载体骨架上,便可得到质粒pet28a-cas9-grna。然后用引物对1+2从pij8600(2)质粒上pcr扩增出tsr-to-tipap片段,通过无缝克隆(vazyme,china)将tsr-to-tipap片段插入到saci/noti酶切后的pet28a-cas9-grna中得到pet28a-tipap-cas9-grna。随后,pet28a-tipap-cas9-grna再通过ecori/bglii酶切,得到长6.3kb的片段,并将该片段与pwhu2653质粒通过ecori/bamhi酶切得到的长9.4kb质粒骨架连接,实现pwhu2653-tipap质粒的构建。同时,通过引物对3+4+5在体外扩增出一段长114bp且包含核糖体开关tiboswitch的片段,并插入到pet28a-tipap-cas9-grna的ndei酶切位点,完成pet28a-tipap-ribo的构建。进一步,将pet28a-tipap-ribo质粒通过ecori/bglii酶切,并将酶切片段连接到pwhu2653经酶切得到的长9.4kb的质粒骨架上,最终得到了pwhu2653-tipap-ribo质粒。 为了实现cas9的分裂表达,参照文献指导采用化学合成的方法,构建了pmag(序列详情见seqno.3)和nmaghigh1(nmag)(序列详情见seqno.4),具体方法是通过将在pmag羧基末端的gsggssgsgg连接体和在nmag氨基末端的ggsgssggsg连接体,分别克隆到pta2(toyobo,japan)中,得到pta2-nmag-cas9c与pta2-cas9n-pmag。之后,选取引物对6+7,以质粒pij8600为模板pcr扩增出包含xbai/ndei酶切位点的to-tipap片段,并将其插入到pta2的xbai位点以得到pta2-tipap。然后再通过引物对8+9,和10+11分别以pta2-nmag和pwhu2653为模板,扩增出nmag和ccas9(cas9羧基末端的1965bp)片段,并将两个片段通过gibson组装一起插入到pta2-tipap质粒的ndei酶切位点,组装成质粒pta2-tipap-nmag-ccas9。相似地,通过引物对12+13和14+15分别以pwhu2653和pta2-pmag为模板,扩增出ncas9(cas9氨基末端的2136bp)和pmag片段,也通过gibson组装一起插入到pet28a-tipap-cas9-grna质粒经ndei/nhei酶切得到的载体,组装成质粒pet28a-tipap-ncas9-pmag。而pet28a-tipap-ncas9-pmag进一步被xbai/bglii酶切,并连接到pta2-tipap-nmag-ccas9经xbai/bglii酶切得到的tipap-nmag-ccas9质粒骨架上,以此得到pet28a-tipap-mag-cas9质粒。最终,pet28a-tipap-mag-cas9质粒选用ecori/bglii酶切,插入到pwhu2653经ecori/bamhi酶切得到的长9.4kb的质粒骨架,得到质粒pwhu2653-tipap-mag-cas9。 为了保证分裂csa9能够进一步受核糖体开关的调控,上述的114bp片段分别用gibson组装插入到由ndei酶切的pet28a-tipap-ncas9-pmag和pta2-tipap-nmag-ccas9载体,分别得到pet28a-tipap-ribo-ncas9-pmag和pta2-tipap-ribo-nmag-ccas9质粒。随后ta2-tipap-ribo-nmag-ccas9由xbai/bglii酶切得到tipap-ribo-nmag-ccas9片段,并将其连接至pet28a-tipap-ribo-ncas9-pmag质粒通过xbai/bglii酶切后的载体骨架,得到pet28a-tipap-ribo-mag-cas9质粒。最后,pet28a-tipap-ribo-mag-cas9再通过ecori/bglii酶切,得到一段8.6kb片段,再将该片段与pwhu2653经ecori/bamhi酶切后得到的9.4kb载体骨架连接,最终得到pwhu2653-trm-cas9质粒。 为了实现reca和aptd的异位结合表达,分别选用引物对16+17,和18+19,并以plm1(4)和s.coelicolora3(2)的基因组为模板扩增出组成型启动子ermep*和reca,并将两者插入pwhu2653-trm-cas9的ndei位点,得到pwhu2653-trm-cas9-reca质粒。除此之外,分别通过引物对20+21,和22+23,以plm1(4)和s.coelicolora3(2)的基因组为模板扩增出组成型启动子ermep*和atpd,并分别插入pwhu2653-trm-cas9的ecori位点,得到pwhu2653-trmd-cas9质粒,以及插入pwhu2653-trm-cas9-reca的ecori位点,得到pwhu2653-trmd-cas9-reca质粒。 为了实现pkc1139上的psg5ori替换掉pwhu2653上的pij101ori,pwhu2653-trm-cas9经ecori/bglii酶切得到的8.0kb片段,插入到pkc1139经ecori/bglii酶切得到的6.0kb质粒骨架上,得到pkc1139-trm-cas9质粒。随后用引物对24+21和22+25分别以plm1(4)和s.coelicolora3(2)的基因组为模板扩增出ermep*和atpd,最后将两者gibson组装插入到pkc1139-trm-cas9的ecori位点,得到pkc1139-trmd-cas9质粒。 为了得到编辑psg5位点的质粒,actii-orf4的间隔序列分别插入到pkc1139-trm-cas9和pkc1139-trmd-cas9质粒的baei位点。actii-orf4的左臂用引物对26+28扩增的同时,两actii-orf4的右臂分别通过引物对27+29和引物对27+30扩增。结合有第一右臂的左臂插入pkc1139-trm-cas9-spacer得到pkc1139-trm-cas9-actii-orf4质粒,而结合有第二右臂的左臂插入kc1139-trmd-cas9-spacer得到pkc1139-trmd-cas9-actii-orf4质粒。(详见表1与附图1) (4)根据天蓝色链霉菌a3(2)生理性质和骨架质粒pkc1139元件,本例选取氨苄青霉素作为合适的筛选转化子的抗生素,用于后续步骤筛选成功导入质粒的转化子筛选条件; (5)在非诱导条件下,将步骤(4)中的重组质粒接合转导进入天蓝色链霉菌a3(2)中,具体为:先将目的质粒导入e.coliet12567/puz8002(kiesert,2000)中,挑取单克隆至含有氨苄青霉素的lb试管中过夜培养,接着将过夜培养的含目的质粒的e.coliet12567/puz8002按照1%接种量转接到25ml含有相应抗生素的lb培养基中,培养至a260约为0.4时,4000rpm离心5分钟收集菌体;加入1mllb液体培养基重悬菌体,4000rpm离心5分钟,弃上清,最后用0.5mllb液体培养基重悬菌体;取适量事先收集的链霉菌孢子,分装到各个离心管中,类似上述步骤洗涤孢子2次,然后用2×yt培养基重悬菌体,接着将装有孢子的离心管置于水浴锅中萌发10分钟,具体温度根据链霉菌的不同种类进行调整;将0.5mlcoli菌液和0.5ml链霉菌孢子悬液混合;将混合液用5000rpm离心5分钟,吸除部分上清,余下200微升上清重悬菌体和孢子,再将混合液均匀涂布于ms平板上;将ms平板倒置于30度培养箱中培养,16小时后涂布相应的抗生素和萘啶酮酸,30℃培养5天左右长出转化子。此时得到的便是成功编辑的转化子; (6)将步骤(5)中的单克隆转化子接种到添加氨苄青霉素的富集培养基(装液量为35ml/250ml),摇床恒温发酵24h–48h(30℃,220rpm); (7)取步骤(6)中的发酵液,离心收取菌体,用无菌水清洗两次,离心弃上清,更换添加有终浓度为5μg/ml的硫链丝菌素和终浓度为4mm的茶碱的培养基,随后在蓝光诱导条件下摇床恒温培养24h–48h; (8)取(7)中发酵液离心(4000rpm,5min),弃上清,得到完成目的编辑的菌株沉淀。 表1构建编辑质粒所用(相关)引物 序列表 浙江大学 一种增强放线菌基因编辑效率的方法及其应用 34 siposequencelisting1.0 1 183 dna streptomycescoelicolor 1 tccgctcccttcttctctgacgccgtccacgctgcctcctcacgtgacgtgaggtgcaag60 cccggacgttccgcgtgccacgccgtgagccgccgcgtgccgtcggctccctcagcccag120 atccccgatccggtccagtaatgacctcagaactccatctggatttgttcagaacgctcg180 gtt183 2 60 dna 人工合成(unknow) 2 catcttgttgcctccttagcagggtgctgccaagggcatcaagacgatgctggtatcacc60 3 2136 dna 人工合成(unknow) 3 gacctgggccttctggatgtcctccttgaaggtcaggctgtcgtcgtggatgagctgcat60 gaagttgcggttggcgaagccgtcggacttgaggaagtccaggatggtcttgccgctctg120 cttgtcccggatgccgttgatcagcttccgggagaggcggccccagccggtgtaccggcg180 ccgcttcagctgcttcatcaccttgtcgtcgaacaggtgggcgtaggtcttgagccgctc240 ctcgatcatctcgcggtcctcgaagagggtcagggtgaggacgatgtcctccaggatgtc300 ctcgttctcctcgttgtcgaggaagtccttgtccttgatgatcttgagcaggtcgtggta360 ggtgcccagggaggcgttgaagcggtcctccacgccgctgatctcgacggagtcgaagca420 ctcgatcttcttgaagtagtcctccttcagctgcttcacggtgaccttgcggttggtctt480 gaagagcaggtcgacgatcgccttcttctgctcgcccgacaggaaggccggcttccgcat540 gccctcggtcacgtacttgaccttggtcagctcgttgtacacggtgaagtactcgtagag600 cagggagtgcttgggcaggaccttctcgttcgggaggttcttgtcgaagttggtcatgcg660 ctcgatgaacgactgggcggaggcgcccttgtccacgacctcctcgaagttccacggggt720 gatggtctcctccgacttccgggtcatccacgcgaaccgggagttgccgcgggccagggg780 gccgacgtagtacgggatgcggaaggtcaggatcttctcgatcttctcgcggttgtcctt840 caggaaggggtagaagtcctcctggcgccggaggatggcgtgcagctcgcccaggtggat900 ctggtgcgggatggagccgttgtcgaaggtccgctgcttgcggagcaggtcctcgcggtt960 cagcttgacgagcagctcctcggtgccgtccatcttctccaggatcggcttgatgaactt1020 gtagaactcctcctgcgacgcgccgccgtcgatgtagccggcgtagccgttcttggactg1080 gtcgaagaagatttccttgtacttctcgggcagctgctggcgcacgagggccttgagcag1140 ggtcaggtcctggtggtgctcgtcgtaccgcttgatcatgctcgccgacagcggggcctt1200 ggtgatctcggtgttgacccgcaggatgtcgctgagcaggatggcgtccgagaggttctt1260 cgcggccaggaagaggtccgcgtactggtcgccgatctgggcgagcaggttgtccaggtc1320 gtcgtcgtaggtgtccttggacagctggagcttcgcgtcctcggccaggtcgaagttgct1380 cttgaagttgggggtcaggccgagcgacagcgcgatcaggttgccgaagaggccgttctt1440 cttctcgcccggcagctgggcgatgaggttctccaggcgccgggacttgctcaggcgcgc1500 ggagaggatcgccttggcgtcgacgccgctggcgttgatggggttctcctcgaacagctg1560 gttgtaggtctgcaccagctggatgaagagcttgtcgacgtcggagttgtccgggttcag1620 gtcgccctcgatgaggaagtggccgcggaacttgatcatgtgcgcgagggccaggtagat1680 gagccgcaggtccgccttgtcggtcgagtcgaccagcttcttgcggaggtggtagatggt1740 ggggtacttctcgtggtaggccacctcgtcgacgatgttgccgaagatcgggtggcgctc1800 gtgcttcttgtcctcctccaccaggaagctctcctcgagccggtggaagaacgagtcgtc1860 gaccttggccatctcgttggagaagatttcctggaggtagcagatgcggttcttgcgccg1920 ggtgtagcgccggcgcgcggtccgcttcaggcgggtcgcctcggcggtctcgccgctgtc1980 gaagagcagggcgccgatcaggttcttcttgatcgagtgccggtcggtgttgcccaggac2040 cttgaacttcttggaggggaccttgtactcgtcggtgatgaccgcccagcccacgctgtt2100 ggtgccgatgtccaggccgatgctgtacttcttgtc2136 4 1965 dna 人工合成(unknow) 4 gtcgccgcccagctggctcaggtcgatgcgggtctcgtacaggccggtgatgctctggtg60 gatcagggtcgcgtcgaggacctccttggtggaggtgtaccgcttgcggtcgatggtggt120 gtcgaagtacttgaaggcggccggggcacccaggttggtgagggtgaacaggtggatgat180 gttctccgcctgctcccggatcggcttgtcgcggtgcttgttgtaggcggacagcacctt240 gtcgaggttcgcgtcggccaggatgacgcgcttgctgaactcgctgatctgctcgatgat300 ctcgtccaggtagtgcttgtgctgctccacgaagagctgcttctgctcgttgtcctcggg360 cgagcccttcagcttctcgtagtgggacgcgaggtacaggaagttgacgtacttcgacgg420 gagggccagctcgttgcccttttgcagctcgcccgcggaggcgagcatccgcttgcggcc480 gttctccagctcgaacaggctgtacttgggcagcttgatgatgaggtccttcttgacctc540 cttgtagcccttggcctccaggaagtcgatcgggttcttctcgaacgaggagcgctccat600 gatggtgatgccgagcagctccttcacggacttcagcttcttgctcttgcccttctcgac660 cttcgccacgaccagcacggagtaggcgacggtggggctgtcgaagccgccgtacttctt720 cgggtcccagtccttcttccgggcgatcagcttgtcgctgttgcgcttggggaggatgga780 ctccttgctgaagccgccggtctgcacctcggtcttcttcacgatgttgacctgcggcat840 cgacagcaccttccggacggtggcgaagtcgcggcccttgtcccagacgatctcgccggt900 ctcgccgttggtctcgatcaggggccgcttgcggatctcgccgttggccagggtgatctc960 ggtcttgaagaagttcatgatgttggagtagaagaagtacttcgcggtggccttgccgat1020 ctcctgctcgctcttggcgatcatcttgcgcacgtcgtagaccttgtagtcgccgtagac1080 gaactcggactccagcttcgggtacttcttgatcagcgcggtgcccacgacggcgttcag1140 gtacgcgtcgtgggcgtggtggtagttgttgatctcccggaccttgtagaactggaagtc1200 cttgcggaagtcggagaccagcttgctcttgagggtgatcaccttgacctcgcggatcag1260 cttgtcgttctcgtcgtacttggtgttcatccgggagtccaggatctgggccacgtgctt1320 ggtgatctgccgggtctcgaccagctggcgcttgatgaagcccgccttgtccagctcgct1380 caggccgccccgctcggccttggtcaggttgtcgaacttgcgctgggtgatgagcttggc1440 gttgagcagctggcgccagtagttcttcatcttcttcacgacctcctccgagggcacgtt1500 gtcggacttgccccggttcttgtccgagcgggtcaggaccttgttgtcgatcgagtcgtc1560 cttcaggaaggactgcggcacgatgtggtcgacgtcgtagtcggacagccggttgatgtc1620 cagctcctggtccacgtacatgtcgcggccgttctggaggtagtagaggtacagcttctc1680 gttctggagctgggtgttctcgaccgggtgctccttcaggatctggctgcccagctcctt1740 gatgccctcctcgatccgcttcatccgctcgcgcgagttcttctggcccttctgggtggt1800 ctggttctcccgggccatctcgatcacgatgttctcgggcttgtggcggcccatcacctt1860 gaccagctcgtccacgaccttgacggtctggaggatgcccttcttgatcgccggggagcc1920 cgccaggttggcgatgtgctcgtggaggctgtcgccctggcccga1965 5 46 dna 人工合成(unknow) 5 gtggtggtggtggtgctcgagtgaattcgatggggatcaaggcgaa46 6 45 dna 人工合成(unknow) 6 ccgatgctgtacttcttgtccatatgtccgctcccttcttctctg45 7 55 dna 人工合成(unknow) 7 gaagaagggagcggacataatacgactcactataggttccggtgataccagcatc55 8 58 dna 人工合成(unknow) 8 cttgttgcctccttagcagggtgctgccaagggcatcaagacgatgctggtatcaccg58 9 34 dna 人工合成(unknow) 9 ctgtacttcttgtccatcttgttgcctccttagc34 10 42 dna 人工合成(unknow) 10 tgtactgagagtgcaccatctagagattactgtcgtttaatg42 11 41 dna 人工合成(unknow) 11 caccgcggtggcggccgccatatgtccgctcccttcttctc41 12 40 dna 人工合成(unknow) 12 gagaagaagggagcggacatatgcacaccctgtacgcccc40 13 18 dna 人工合成(unknow) 13 gccgcccgagccggagcc18 14 40 dna 人工合成(unknow) 14 ggctccggctcgggcggcgattcgggccagggcgacagcc40 15 43 dna 人工合成(unknow) 15 accgcggtggcggccgcagatctttagtcgccgcccagctggc43 16 41 dna 人工合成(unknow) 16 gagaagaagggagcggacatatggacaagaagtacagcatc41 17 36 dna 人工合成(unknow) 17 gagccggagccgccgatgacctgggccttctggatg36 18 20 dna 人工合成(unknow) 18 atcggcggctccggctcgtc20 19 39 dna 人工合成(unknow) 19 tagacacgtctgaagctagcctcactcggtctcgcactg39 20 39 dna 人工合成(unknow) 20 ttcagcgtgacatcattcataggcggcttgcgcccgatg39 21 19 dna 人工合成(unknow) 21 atgtccgcctcctttggtc19 22 39 dna 人工合成(unknow) 22 accaaaggaggcggacatatggcaggaaccgaccgcgag39 23 40 dna 人工合成(unknow) 23 gatcaaggcgaatacttcatatgatccgtctcgtacgggg40 24 37 dna 人工合成(unknow) 24 gaggccctttcgtcttcaaggcggcttgcgcccgatg37 25 40 dna 人工合成(unknow) 25 ggtctcaacagtggtggtcatatgtccgcctcctttggtc40 26 18 dna 人工合成(unknow) 26 atgaccaccactgttgag18 27 36 dna 人工合成(unknow) 27 ttcgccttgatccccatcgcgaccagcgcgacgtgc36 28 37 dna 人工合成(unknow) 28 acagctatgacatgattacggcggcttgcgcccgatg37 29 41 dna 人工合成(unknow) 29 ttcgccttgatccccatcgaattcgcgaccagcgcgacgtg41 30 18 dna 人工合成(unknow) 30 ctgcgcccccgtcgagat18 31 36 dna 人工合成(unknow) 31 atctcgacgggggcgcaggagaaggtgctcgtgtag36 32 40 dna 人工合成(unknow) 32 cgccttgatccccatcgaattcaccaagccggagtcggtg40 33 40 dna 人工合成(unknow) 33 cgacctgcaggcatgcaagcttgtggtcgtcgtcatcgtc40 34 41 dna 人工合成(unknow) 34 gtgcttgcggcagcgtgaagcttagcagcgagacgacgagg41 |
【本文地址】