| 【实用讲解】NCBI使用教程3:基因序列的比对 | 您所在的位置:网站首页 › ncbi怎么看基因序列 › 【实用讲解】NCBI使用教程3:基因序列的比对 |
【实用讲解】NCBI使用教程3:基因序列的比对
|
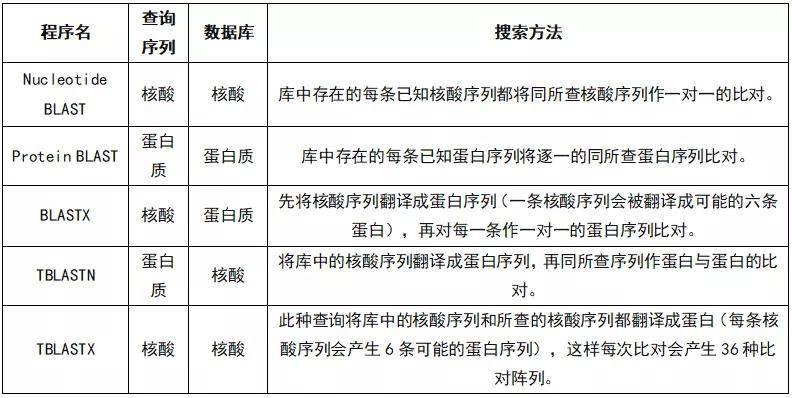
原标题:【实用讲解】NCBI使用教程3:基因序列的比对 BLAST是“局部相似性基本查询工具”(Basic Local Alignment Search Tool)的缩写。是由美国国立生物技术信息中心(NCBI)开发的一个基于序列相似性的数据库搜索程序。该程序将DNA/蛋白质序列与公开数据库所有序列进行匹配比对,从而找到相似序列。 生物序列的相似性 相似性(similarity):是指一种很直接的数量关系,比如部分相同或相似的百分比或其它一些合适的度量。比如说,A序列和B序列的相似性是80%,或者4/5。这是个量化的关系。当然可进行自身局部比较。 序列相似性比较:就是将待研究序列与DNA或蛋白质序列库进行比较,用于确定该序列的生物属性,也就是我出与此序列相似的已知序列是什么。完成这一工作只需要使用两两序列比较算法。常用的程序包有BLAST、FASTA等。 Blast主要程序
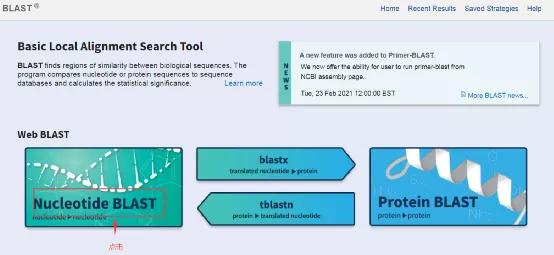
1.打开blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi)
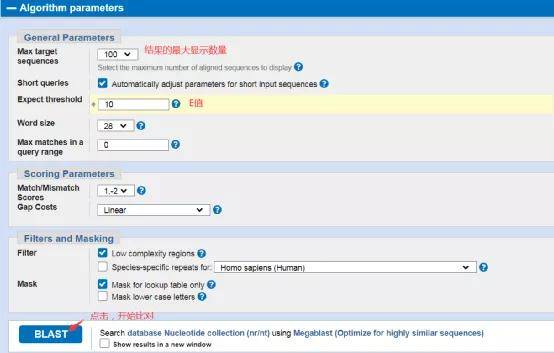
2.输入比对序列及参数
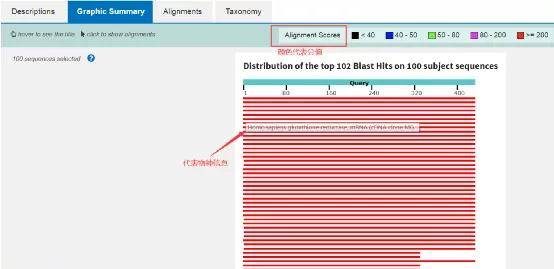
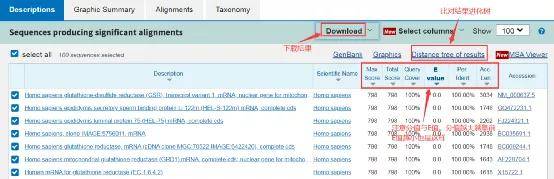
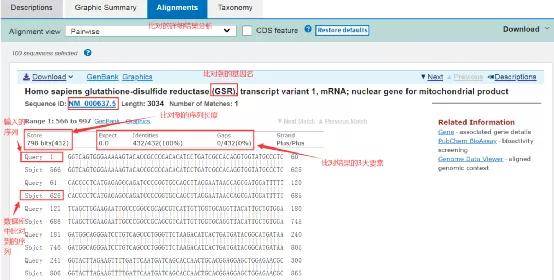
3. BLAST结果分析
一般评价一个blast结果的标准主要有三项,E值(Expect),一致性(Identities),缺失或插入(Gaps)。通用还会额外加上序列长度(length)。 Score:序列比对过程中计算的得分值,得分越高,序列匹配结果越好。 Expect:表示随机匹配的可能性。E值越小,序列越相似,E值越大,随机匹配的可能性也越大。E值接近零或为零时,具本上就是完全匹配了。 Identities:序列相似性,匹配上的碱基数占总序列长的百分数。 Gaps:插入或缺失。用"—"来表示。
好,推送的关于NCBI的三篇文章就都讲解完毕啦 分别是: 目的基因查找 QPCR引物设计 基因序列比对 你学会了么?返回搜狐,查看更多 责任编辑: |
【本文地址】