| 下拉菜单实现树状结构 | 您所在的位置:网站首页 › motif分析图怎么看 › 下拉菜单实现树状结构 |
下拉菜单实现树状结构
|
我们在日常分析中,有时候会比较不同物种间motif序列的保守性。今天小编教大家使用R包“motifStack ”绘制美观的motif序列结构图!  ## 安装R包
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("motifStack")
## 安装R包
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("motifStack")
安装好R包后,我们就可以准备输入文件啦~ 比如我想绘制拟南芥中HSF结合的motif结构。 在JASPAR(JASPAR 2020: An open-access database of transcription factor binding profiles)中搜索“HSF”。 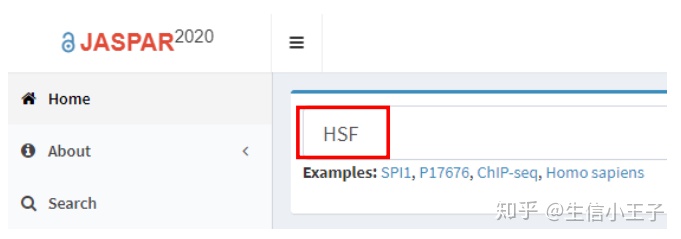
找到自己感兴趣的motif,点击ID号。 
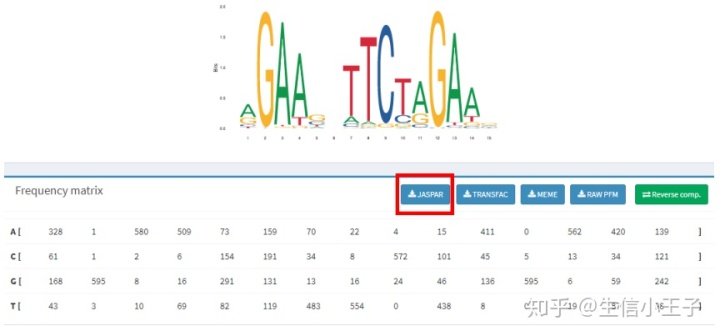
下载JASPAR 格式的矩阵。 
我们需要将它处理成以下格式(空格分隔),并命名为"motif_ID.pcm"。 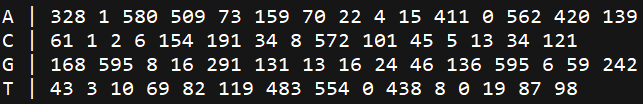
如果你想展示你自己鉴定的motif结构的话,可以使用小编写的脚本(https://github.com/biozhp/motifStack_input)。 使用脚本前需要准备两个输入文件: 输入文件一:mofit序列(第一列为ID,第二列为序列,Tab分隔)。 
输入文件二:mofit ID。 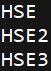 ## 输入mofit序列, motif ID及输出文件位置
sh ./run.sh motif.seq motif.id out_path
## 输入mofit序列, motif ID及输出文件位置
sh ./run.sh motif.seq motif.id out_path
执行脚本完毕后即可获得矩阵文件,开始绘制motif序列结构图。 ## 加载R包 library("motifStack") ## 导入motif文件 pcm |
【本文地址】
公司简介
联系我们