| 【精选】R机器学习:分类算法之K最邻进算法(KNN)的原理与实现 | 您所在的位置:网站首页 › k邻近算法原理 › 【精选】R机器学习:分类算法之K最邻进算法(KNN)的原理与实现 |
【精选】R机器学习:分类算法之K最邻进算法(KNN)的原理与实现
|
从今天开始给大家写机器学习算法,这个东西并不是大多数人想象的那么高深,也不是说编程的人,搞计算机的人才能学习使用,医学领域、社会科学领域的研究越来越多运用机器学习的,在我的理解中每个人都应该掌握基本的机器学习思想和基本的编程能力。 这个系列的第一篇文章从简单的分类算法KNN开始:这个算法真的非常的简单,简单到初中生都可以掌握,所以大家一定要有信心: kNN is arguably the simplest machine learning algorithm. In spite of its simplicity, kNN can provide surprisingly good classification performance, and its simplicity makes it easy to interpret. KNN算法思想解释K最近邻算法是一种分类算法,算法思想是在数据集中找到与样本最相似的K个样本,如果这K个样本中的大多数属于某一类别,则该样本也属于某一类别。 用例子说明:现在有3个爬行动物,其中两个是毒蛇分别叫做:grass snake and the adder,一个是虫子叫做slow worm。这个虫子和蛇长得挺像,很多人都会把它们搞混。 现在你参与了一个项目:去调查一片草地上到底这三种动物到底有多少个,在实地调查的时候你只能通过动物的身长Body length和攻击性aggression这两个指标来对其的类型做出判断,你和一个动物专家已经收集了一些动物数据(已经有带标签的数据集了),现在你自己想训练一个KNN模型对动物进行自动分类。 你收集的数据可视化如下图: 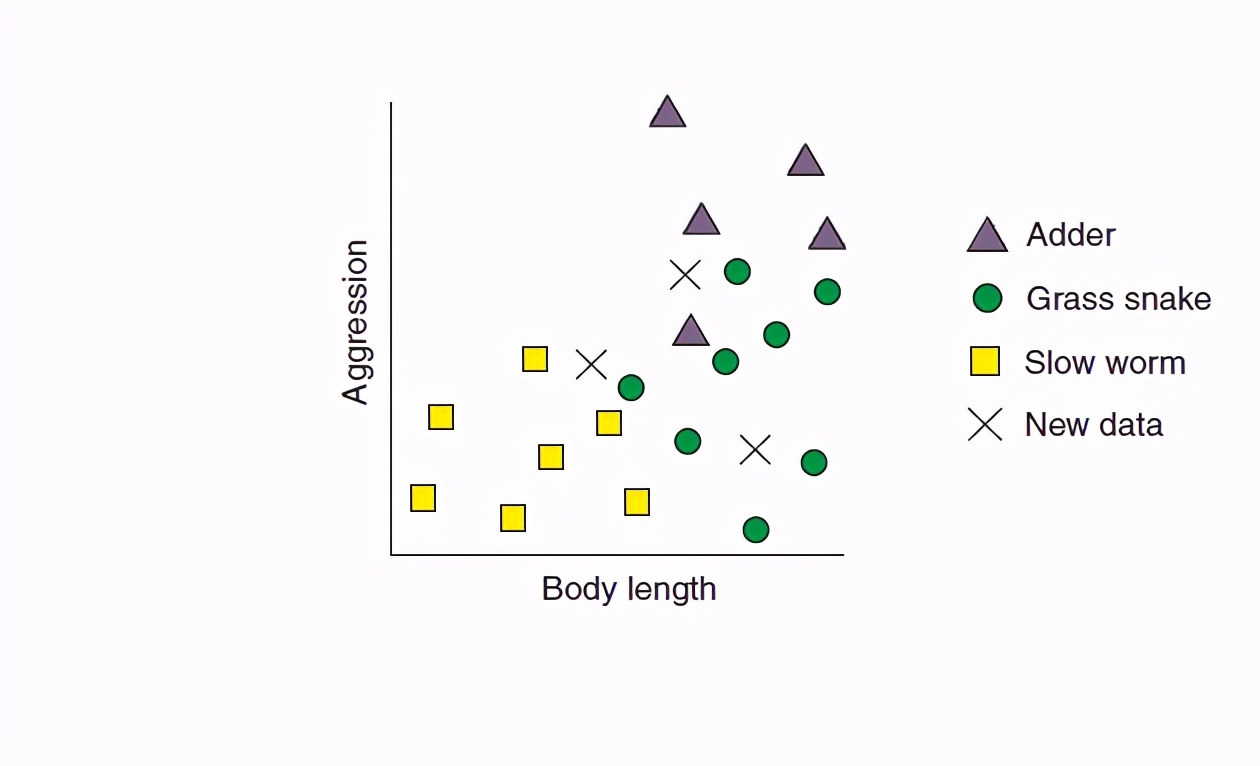
图中有每个爬行动物在身长和攻击性两个维度的值,XX就是待分类的动物的身长和攻击性,我们现在需要用KNN模型去判断这三个X分别是哪一类。 KNN算法是如何做的呢? 对于每一个X,算法会去计算这个X到周围已知点的距离,比如对最上面的X我们计算距离可以如下(所有的线就代表欧几里得距离):
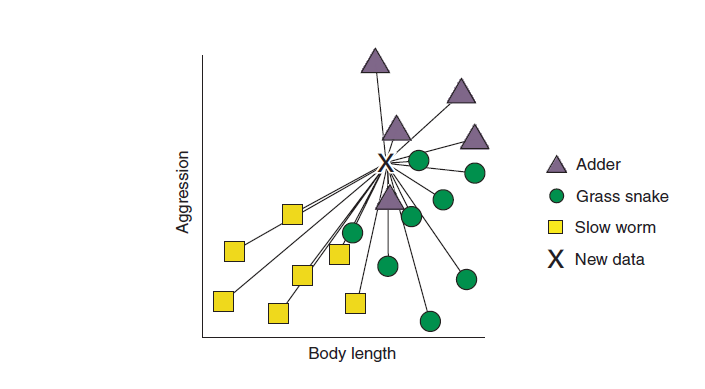
距离有了之后我们对距离进行排序:
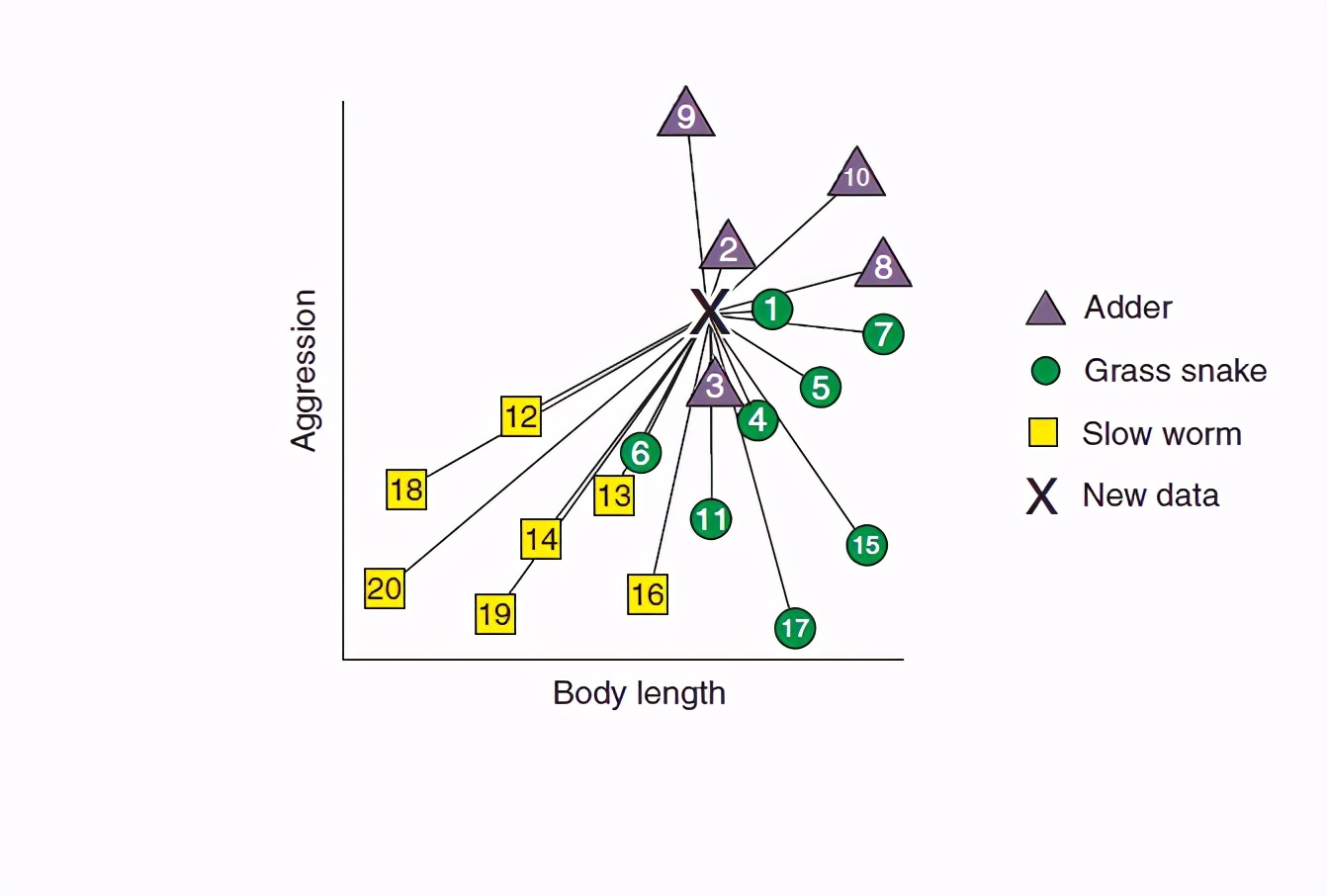
然后我们取前K个最近距离的点(这个K是自己设定的,叫做超参数),然后这个K个点中类别占比最多的那一类就是我们的X的所属类别。 我们还是把上面的步骤在例子中给大家走一遍: 我把K分别设定为1,3,5,就是说我希望我新收集的X的类别和它最邻近的1,3,5个爬行动物的类别一样。 那么整个分类过程就如下图:
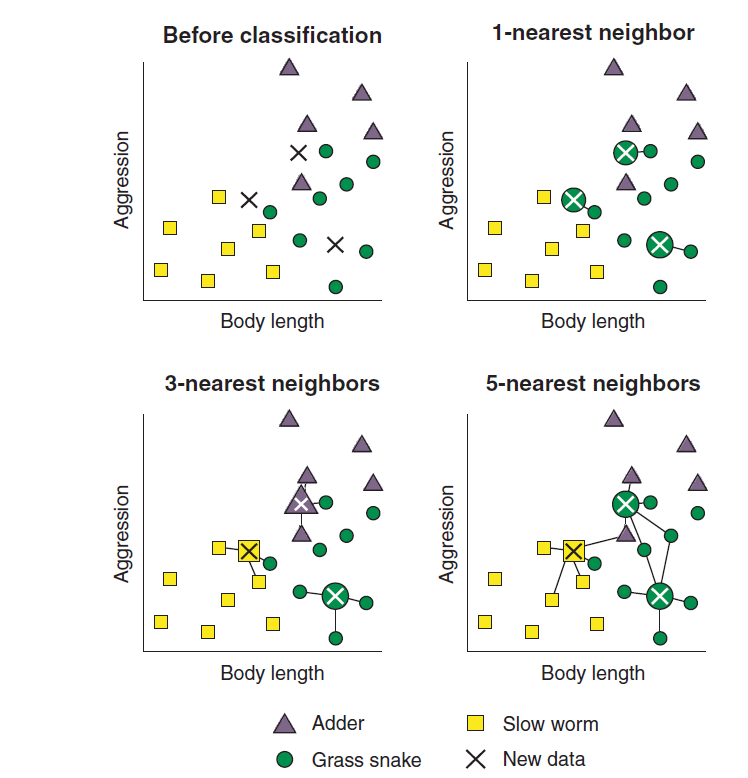
从图中可以看出:当K为1时3个X都被KNN认为是grass snake,当K为3时3个X被KNN模型分为了3个不同的类别,当K为5时我们的3个X被分为了2个不同的类别。所以,K会影响KNN算法的表现,K不同算法结果不同。 有没有人好奇如果我把K设置为4然后就找了前4个最近的已知样本,2个是A类,两个是B类,A和B打了个平局,这个时候X到底是哪一类呢? 这个时候,如果我们的分类问题本身是一个两分类问题我们一定记住把K设置为奇数,就可以避免平局的出现,如果为多分类问题,算法会直接进行随机分,因为实际操作中K个邻居中有类别数量等同的情形还是比较少的,对整体结果不会产生实际影响。 最后给大家回顾一下KNN的实现过程: 构建一个已经分类好的数据集。计算一个新样本与数据集中所有数据的距离。按照距离大小进行递增排序。选取距离最小的K个样本。确定前K个样本所在类别出现的频率,并输出出现频率最高的类别。 KNN建模实操写完原理,我们继续用一个例子给大家写实际操作,假如你是一个医生,手上有很多糖尿病病人的诊断数据,你现在想建立一个KNN模型实现对新病人的诊断,诊断结果有3类:healthy, chemically diabetic, overtly diabetic。 一个很简单的3分类问题哈。 我们的数据长这样: 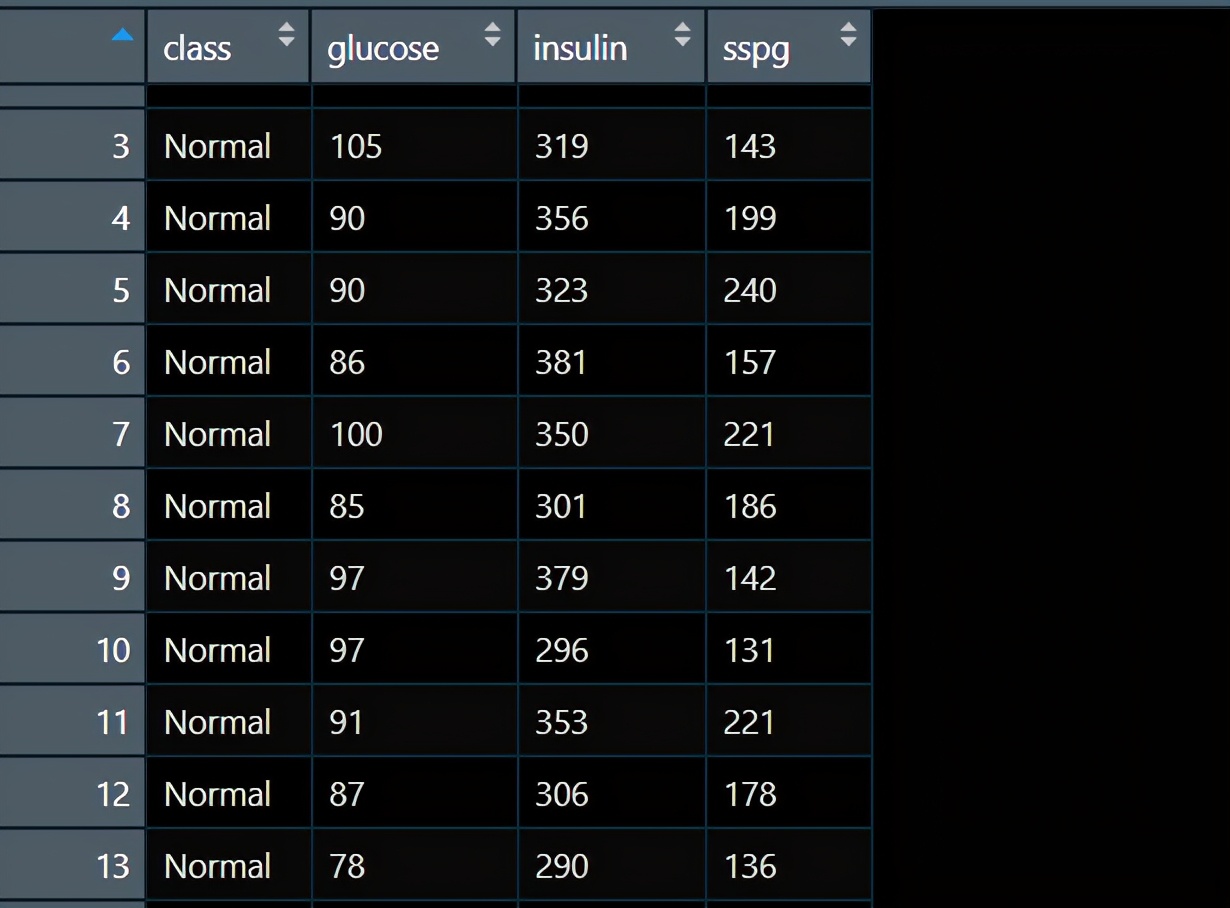
4个变量,第一个变量就是分类结局,取值可以为non-diabetic (Normal),chemically diabetic (Chemical), overtly diabetic (Overt) 训练模型之前我们可以先画图看看数据分布: ggplot(diabetesTib, aes(glucose, insulin, col = class)) + geom_point() + theme_bw() ggplot(diabetesTib, aes(sspg, insulin, col = class)) + geom_point() + theme_bw() ggplot(diabetesTib, aes(sspg, glucose, col = class)) + geom_point()+ theme_bw()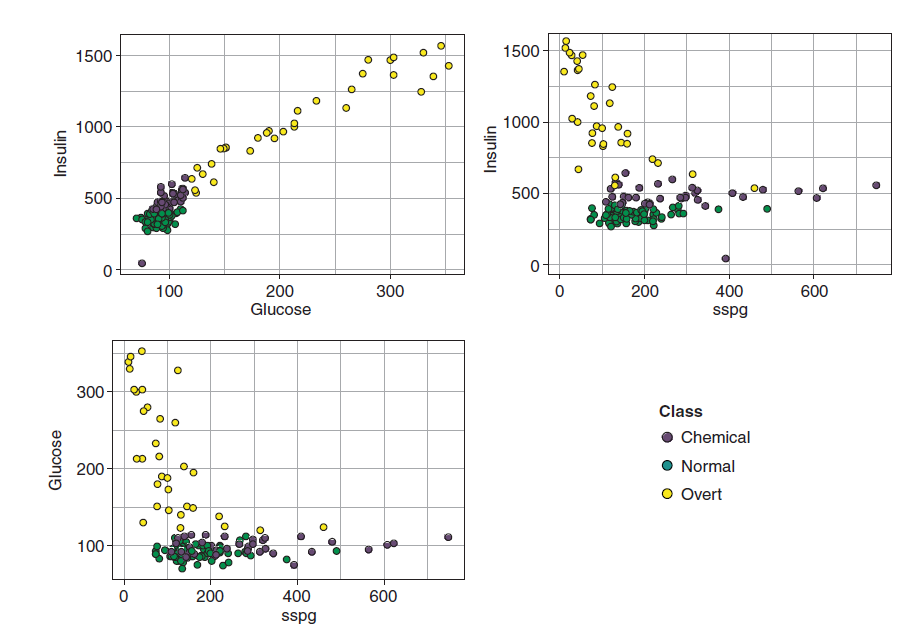
上面的图是不同的自变量组合下的病人类别分布,其实可以看出来仅仅使用glucose和insulin两个变量就可较好地区分三种类别了,不过在本例中我们依然使用所有的预测变量进行模型的训练,我们用到的包是非常经典的mlr包,第一步是定义学习任务makeClassifTask,第二部是定义学习器makeLearner,第三步就是训练模型了,具体代码如下: library(mlr) diabetesTask |
【本文地址】